 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 13 篇 doi: 10.5376/gab.cn.2011.30.0013
收稿日期: 2011年03月09日 接受日期: 2011年03月24日 发表日期: 2011年04月02日
引用格式(中文):
刘超等, 2011,不依赖于连接反应克隆(LIC)的技术进展,基因组学与应用生物学(online), Vol.30 No.13 pp.1081-1085 (doi: 10.5376/gab.cn.2011.30.0013)
引用格式(英文):
Liu et al., 2011, Research advance on ligation-independent cloning (LIC) method, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.13 pp.1081-1085 (doi: 10.5376/gab.cn.2011.30.0013)
不依赖于连接反应的高效克隆方法(ligation–independent cloning, LIC)是以同源重组的基本原理,利用T4 DNA聚合酶的3′-5′外切酶活性在待插入片段以及线性载体的末端产生单链突出,通过同源序列在体外完成交换重组,达到基因克隆目的。本文在总结国内外不依赖于连接反应克隆(LIC)研究的基础上,概述了LIC技术的实现方法特点及改进,并阐述了LIC技术在目的基因克隆、某些蛋白的融合表达方面的应用及其前景。希望读者对此领域有一个比较深入和系统的认识,同时也能挖掘LIC技术在基因重组领域的应用潜力
DNA的限制酶酶切和酶促连接作为基因工程的基础技术,是20世纪分子生物学方法的重要成就之一。在此基础上,研究者利用PCR (Rumsby, 2006)和定点突变(Baldrich and Goebel, 1990)等技术极大地拓展了重组DNA技术的应用。目前克隆方法多采用依赖连接酶的克隆方法,此法是一种经典的克隆方法,主要分为以下两种:1)粘性末端连接(Stols et al., 2002),即载体质粒和待插入的外源片段都通过合适的限制性内切酶切割,产生相互匹配的粘性末端,然后在连接酶作用下重新形成磷酸二酯键而得到环化的重组质粒;2)平末端连接(Dieckman et al., 2002),平末端连接中,载体和片段均为平端,都没有粘性末端突出。其优点是步骤简单,不需要多次酶切,但是克隆效率较低,而且不能实现定向克隆。总的来说,依赖连接酶的克隆方法简单高效,是克隆单个基因的常用的方法。但该技术也存在明显的不足:一方面,插入片段要经过限制性内切酶处理,这样在实验设计时就要考虑目的基因的DNA序列,不能实现多基因的平行操作,不适合高通量;另一方面,该过程使用连接酶,一方面延长了实验周期,另一方面也会带来载体自连背景,增加了筛选工作量。
然而在很多情况下,重组DNA技术的应用都会受到基因序列中原有的酶切位点的限制,尤其是多基因片段的重组往往更容易受到限制酶酶切位点序列的限制(Cheo et al., 2004),研究者不得不使用一些非常稀有的酶切位点或者采用其它的DNA重组方法,大大增加了DNA重组的工作量和难度。因此,开发一种不受序列限制而且需要准确重组的新方法就显得十分重要。
Aslanidis和de Jong (1990)在1990年所提出的不依赖连接反应克隆(ligation-independent cloning, LIC)充分满足了上述要求。这种方式不仅克服了常规依赖连接酶克隆方法的缺点,而且操作简便、方法快捷及连接效率高,应用日益广泛。本文综述了LIC的实现方法特点,发展历程及其在目的基因克隆和蛋白融合表达中的应用。
1不依赖连接反应的克隆
建立高通量表达和纯化蛋白质的方法在蛋白组时代是必需的。受到基因序列中原有的酶切位点的限制传统的依赖连接酶的克隆方法在应用中受到一定的局限,目前不依赖连接反应的克隆(LIC)方法利用独特新颖的思路解决了这一问题。
1.1不依赖连接反应的克隆实现方法
在T4 DNA聚合酶的3′-5′外切酶活性下和一种特定的dNTP存在的缓冲液条件下反应,插入片段能产生10~15个碱基的粘性末端(Aslanidis and de Jong, 1990)。载体和PCR片段依靠长的粘性末端重组,此过程不需要连接酶。这种克隆方法不需要考虑插入片段的序列,任何基因都能被克隆到该载体中。而且,整个流程简单到不需要任何限制性内切酶和连接酶,两者在体外条件下孵化,互补的粘性末端退火形成环状分子,经转化进大肠杆菌细胞后,细菌的修复系统修复这些突出和缺口而得到正确重组子(图1)。图1中显示的是插入片段和载体均用PCR方式获得,然后用T4 DNA聚合酶处理,体外退火后直接转化大肠杆菌得重组质粒。
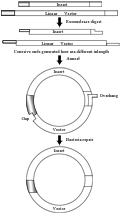 图1 不依赖于连接的克隆方法 |
1.2不依赖连接反应的克隆特点
LIC方法依赖于T4 DNA聚合酶的核酸外切酶活性,这样就要求被处理的线性载体和片段的末端仅含有3种碱基,这样才可以有效控制反应以产生特定大小的粘性末端。要求载体有较固定的粘性末端,且对于如何使载体线性化有特殊的要求。LIC方法的优势(Doyle, 2005)在于:1)待克隆的PCR片段不需要限制性内切酶处理。这样就省去了在设计引物时考虑各种酶切位点的麻烦,而且实验过程中可以实行并行操作,对所有的样品进行同样的处理。2)克隆效率高且操作简便。如前所述,该方法不依赖连接酶,完全是靠粘性末端配对退火,所以不匹配的末端不可能形成双链,载体自连背景低。另外,也省去了载体去磷酸化等消化步骤。3)费用低。该方法中所需要的引物虽然有额外10~15 bp附加碱基,但是相对而言位点特异性重组中所要求的引物是较短的,而且克隆过程中省去了连接酶,也没有使用价格不菲的特殊重组酶。综合以上几个方面考虑,LIC是一种适合于高通量基因克隆和蛋白质表达的优秀方法。
2不依赖连接反应的克隆方法的改进
Haun和Moss (1992)在此后的研究中,利用限制性内切酶消化使载体线性化,然后经T4 DNA聚合酶处理以产生长的粘性末端(图2),这样的方式可以避免PCR过程中载体上产生突变(Kaluz and Flint, 1994)。这个过程中,载体的两粘性末端彼此无同源性,也没有使用连接酶,因而载体自连背景低,重组效率高。
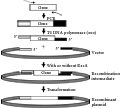 图2 改进的不依赖于连接的克隆方法 |
在LIC理论基础上,2007年,Li和Elledge (2007)报道了一种不依赖于基因序列和连接反应的高效基因克隆方法(sequence and ligation-independent cloning, SLIC)。SLIC用途广泛,因为它不仅可用于任何2个基因片段的重组,更重要的是可实现多个DNA片段的一次性连接重组(图3)。由于不同基因片段之间长达30 bp的同源片段的存在,可以严格保证连接顺序与预期一致。SLIC只需要在PCR引物上加入与载体同源的20~30 bp DNA片度,不需要酶切和连接反应,极大地简化了DNA重组过程,由于在这种情况下不存在DNA分子自连接的可能性,更加简化了重组子的筛选过程。在不超过5个片段的情况下,可以保证转化子100%为重组子;在10个基因片段连接的情况下,16%的转化子为正确的重组子。
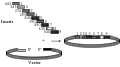 图3 SLIC在多基因片段重组中的作用 |
在Li和Elledge (2007)的研究中没有使用基因功能或片段两侧的酶切位点验证插入片段是否完整,而是通过最后的测序确定其重组片段的可靠性,白帆等(2008)人用Ned I和Hind Ⅲ双酶切pUC18载体,并以pUC18作为模板克隆Ned I和Hind Ⅲ位点之间的LacZ基因片段,引物5′端加上与载体同源的30 bp同源DNA。如果正确发生重组,则可恢复pUC18上LacZ基因活性,在X-gal平板上显示蓝色菌落,同时恢复Ned I和Hind Ⅲ酶切位点。该方法使用大肠杆菌蓝白斑筛选来验证在使用SLIC方法时的基因功能是否能够完整保留,大大提高了可靠性。
在实际操作中王广珺等(2010)发现需要重组的DNA片段接头处结构并不能达到理想的状态,即经过T4 DNA聚合酶处理后形成的ssDNA非常容易形成二级结构,这些结构在37℃下不能完全打开,从而影响DNA分子的互补复性并降低重组效率。为解决这个问题他们将退火时使用的连接缓冲液改为退火缓冲液,并使用退火程序使DNA片段的单链区序列配对复性,通过此方法提高SLIC的重组效率达10倍,从而使得SLIC重组效率进一步提高。
3不依赖连接的克隆技术的应用
由于LIC技术在基因重组方面的简便,快捷和连接效率高等优点,一经报道就引起人们极大地兴趣和关注,国外公司已经开始尝试将其用于商业(Nick et al., 2007),比如Novagen公司的RadianceTM 系统及InVitrogen公司二步Gateway TM系统都是基于LIC技术的原理开发的。
2002年,Stols等(2002)报道了第一例将His标签和LIC克隆方式组合在一起的表达载体pMCSG7,该载体被用于大规模的靶基因克隆和表达。2006年,Donnelly等(2006)将此载体稍加改进,在TEV蛋白酶识别位点上游加上一个MBP标签,使得该载体表达的蛋白能够在体内被酶切,除去标签而得到目标蛋白。
谈蓉和马立新(2009)构建了依次包含6His-tag,部分TEV位点,N.BbvCIA识别位点,Swa I位点的pET30a-LIC载体。LIC载体通过N.BbvCIA和Swa I双酶切产生5′粘性末端。使用LIC相配的引物扩增得到的PCR片段在dGTP缓冲液环境中被T4 DNA聚合酶处理后产生了互补的粘性末端。线性化的载体和PCR片段简单地混合,接着被转化进大肠杆菌体内。在重组质粒的亲和标签和靶蛋白之间会重构出完整的TEV蛋白酶切割位点,提高了LIC载体的使用性。此载体整合了LIC和TEV蛋白酶优势,提供了一个快速克隆靶向基因的平台和一个有效筛选可溶性蛋白和表达天然蛋白质的平台。与此方法类似,带有MBP,GST等其他几种不同的亲和融合标签的LIC表达载体已经被用于基因组表达计划(Cabrita et al., 2006; Dan et al., 2009)。Eschenfeldt等(2009)构建了15个LIC载体用于高通量克隆和表达蛋白,在一系列的LIC载体中采用MBP,GST亲和融合标签,改善表达蛋白的可溶性,这一系列的LIC载体在实际应用过程中经验证都具有很好的高通量可溶表达蛋白的能力。
在实际的运用过程中,有研究者(Akira et al., 2009)将目前已得到有效应用的商业化载体进行改造,设计缺失四种碱基之一的一段特殊序列代替繁杂的限制性内切酶位点,此载体通过T4 DNA聚合酶处理,产生长的粘性末端,可利用LIC原来插入各类目的基因进行有效的表达。
与此同时,LIC改进技术在目的基因的克隆中也得到了应用。王中山(2010)通过PCR方法扩增得到大肠杆菌谷胱甘肽转运系统中底物结合蛋白gsiB基因的编码区序列,利用SLIC方法直接将其插入pWaldo-GFPe中成功构建了重组表达质粒pWaldo-GFP-GsiB。将重组质粒转化不同的大肠杆菌表达菌株进行诱导表达,得到了能够大量表达结合蛋白的重组子。
4展望
LIC技术目前已经在目的基因克隆、某些蛋白的融合表达方面得到了广泛应用(Weeks et al., 2007; Chanda et al, 2006)。大规模的基因表达是研究蛋白质功能的基础。随着人类基因组计划草图的完成,生命科学研究逐渐要求对基因进行大规模、并行性分析。由于蛋白质的特性比DNA更加复杂多变,无法用均一的模式来处理不同的样品,也不可能用同样的条件来表达所有的蛋白质,这就要求要发展高效快速的克隆方法,快速鉴定目的基因是否表达,表达量的高低等。在抗体库构建过程中,高通量的筛选能力是抗体库技术的重要特点。而库容量是长期以来一直存在的技术瓶颈,受限于目前的连接和转化技术。LIC技术提高了载体构建过程中的连接效率,插入片段和LIC载体处理后直接转化宿主细胞,免去了工程菌构建的过程,达到提高抗体库库容的目的。基于LIC原理发展的不依赖于基因序列和连接反应的高效基因克隆方法(SLIC)能够做到多片段基因的克隆,这一技术改进使得抗体基因库构建过程更加简化,高效。借助于易错PCR技术,不依赖于连接反应的克隆重组技术为构建酶和抗体基因突变库提供了技术支持,将来可用于酶的分子定向进化和抗体分子的体外成熟。LIC技术的进步对于疾病机理研究,开发基因工程产品,促进生物医药研究等各种领域具有重要意义。
作者贡献
刘超完成文献的收集、选阅和文章初稿的写作;林陈水负责文章的整体构思、修改和审阅。
致谢
感谢浙江省自然科学基金资助项目(Y206760)资助,感谢两位匿名的同行评审人的评审意见和修改建议。
参考文献
Akira T., Kazuo T., Takayuki U., Yuichi S., Ayako H., and Toshizumi T., 2009, Preparation of long sticky ends for universal ligation-independent cloning: Sequential T4 DNA polymerase treatments, Journal of Bioscience and Bioengineering, 107(6): 668-669
Aslanidis C. and de Jong P.J., 1990, Ligation-independent cloning of PCR products (LIC-PCR), Nucleic Acids Res., 18(20): 6069-6074 doi:10.1093/nar/18.20.6069 PMid:2235490 PMCid:332407
Bai F., Liu X., and Zhang Y.Z., 2008, Liability of a novel gene cloning method: Sequence and ligation independent cloning, Sichuan Daxue Xuebao (Ziran Kexueban) (Journal of Sichuan University (Natural Science Edition)), 45(Suppl.): 33-36 (白帆, 刘殉, 张义正, 2008, 一种新的基因克隆方法SLIC的可靠性分析, 四川大学学报(自然科学版), 45(Suppl.): 33-36)
Baldrich M. and Goebel W., 1990, Rapid and efficient site-specific mutagenesis, Protein Eng Des Sel, 3(6): 563 doi:10.1093/protein/3.6.563
Cabrita D.L., Dai W., and Bottomley P.S., 2006, A family of E. coli expression vectors for laboratory scale and high throughput soluble protein production, BMC Biotechnol., 6: 12-20 doi:10.1186/1472-6750-6-12 PMid:16509985 PMCid:1420288
Chanda P.K., Edris W.A., and Kennedy J.D., 2006, A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli, Protein Expression and Purification, 47(1): 217-224 doi:10.1016/j.pep.2005.10.019 PMid:16325426
Cheo D.L., Titus S.A. and Byrd D.R.N., 2004, Concerted assembly and cloning of multiple DNA fragments using in vitro site-specific recombination: Functional analysis of multi-site expression clones, Genome Research, 14: 2111-2120 doi:10.1101/gr.2512204 PMid:15489333 PMCid:528927
Dan H., Balachandran A., and Lin M., 2009, A pair of ligation-independent Escherichia coli expression vectors for rapid addition of a polyhistidine affinity tag to the N- or C-termini of recombinant proteins, J. Biomol. Tech., 20(5): 241-248 PMid:19949695 PMCid:2777343
Dieckman L., Gu M., Stols L., Donnelly M.I., and Collart F.R., 2002, High throughput methods for gene cloning and expression, Protein Expression and Purification, 25(1): 1-7 doi:10.1006/prep.2001.1602 PMid:12071692
Donnelly M.I., Zhou M., Millard C.S., Clancy S., Stols L., Eschenfeldt W.H., Collart F.R. and Joachimiak A., 2006, An expression vector tailored for large-scale, high-throughput purification of recombinant proteins, Protein Expression and Purification, 47(2): 446-454doi:10.1016/j.pep.2005.12.011 PMid:16497515 PMCid:2792030
Doyle S.A., 2005, High-throughput cloning for proteomics research, Methods in Molecular biology, 310(11): 107-113 doi:10.1007/978-1-59259-948-6_7
Eschenfeldt W.H., Lucy S., Millard C.S., Joachimiak A., and Mark I.D., 2009, A family of LIC vectors for high-throughput cloning and purification of proteins, Methods Mol. Biol., 498: 105-115 doi:10.1007/978-1-59745-196-3_7 PMid:18988021 PMCid:2771622
Haun R.S. and Moss J., 1992, Ligation-independent cloning of glutathione S-transferase fusion genes for expression in Escherichia coli, Gene, 112(1): 37-43 doi:10.1016/0378-1119(92)90300-E
Kaluz S., and Flint A.P., 1994, Ligation-independent cloning of PCR products with primers containing nonbase resdues, Nucleic Acids Research, 22(22): 4845-4849 doi:10.1093/nar/22.22.4845 PMid:7984441 PMCid:308542
Li M.Z., and Elledge S.J., 2007, Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC, Nature methods, 4(3): 251-256 doi:10.1038/nmeth1010 PMid:17293868
Nick S.B., David A., Sarah S., Joanne N., Rene A., Nahid R., David I.S. and Raymond J.O., 2007, A versatile ligation-independent cloning method suitable for high-throughput expression screening applications, Nucleic Acids Research, 35(6): e45
Rumsby G., 2006, An introduction to PCR techniques, Methods in Molecular Biology, 324: 75-89 PMid:16761372
Stols L., Gu M., Dieckman L., Raffen R., Collart F.R. and Donnelly M.I., 2002, A now vector for high-throughput, ligation-independent cloning encoding a tobacco etch virus protease cleavage site, Protein Expression and Purification, 25(1): 8-15 doi:10.1006/prep.2001.1603 PMid:12071693
Tan R., and Ma L.X., 2009, Novel ligation-independent cloning vectors for production of native proteins, Hubei Daxue Xuebao (Journal of Hubei University(Natural Science)), 31(4): 415-418 (谈蓉, 马立新, 2009, 一种新的产生天然蛋白的不依赖于连接的克隆载体, 湖北大学学报, 31(4): 415-418)
Wang G.J., He C., and Zhang Y.Z., 2010, Improvement of the sequence and ligation-independent cloning (SLIC) method, Sichuan Daxue Xuebao (Ziran Kexueban) (Journal of Sichuan University (Natural Science Edition)), 47(4): 893-898 (王广珺, 何川, 张义正, 2010, 不依赖基因序列和连接反应克隆(SLIC)方法改进, 四川大学学报(自然科学版), 47(4): 893-898)
Wang Z.S., Xiang Q.J., Wang H.Y. and Zhang Y.Z., 2010, Cloning and optimizing expression of a periplasmic solute-binding gene gsiB from Escherichia coli, Yichuan (Hereditas (Beijing)), 32(5): 505-511 (王中山, 向泉桔, 王海燕, 张义正, 2010, 大肠杆菌细胞周质底物结合蛋白gsiB基因的克隆及其表达条件的优化, 遗传, 32(5): 505-511)
Weeks S.D., Drinker M., and Loll P.J., 2007, Ligation independent cloning vectors for expression of SUMO fusions, Protein Expression and Purification, 53(1): 40-50 doi:10.1016/j.pep.2006.12.006 PMid:17251035 PMCid:1892228