2. 北京林业大学微生物研究所, 北京, 100083
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 26 篇 doi: 10.5376/gab.cn.2011.30.0026
收稿日期: 2011年04月28日 接受日期: 2011年05月20日 发表日期: 2011年06月20日
引用格式(中文):
杜萍等, 2011,野生毛木耳SRAP-PCR反应体系的建立与优化,基因组学与应用生物学(online), Vol.30 No.26 pp.1166-1173 (doi: 10.5376/gab.cn.2011.30.0026)
引用格式(英文):
Du et al., 2011, Establishment and optimization of SRAP-PCR reaction system for wild Auricularia polytricha, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.26 pp.1166-1173 (doi: 10.5376/gab.cn.2011.30.0026)
为获得理想的野生毛木耳(Auricularia polytricha) SRAP扩增效果,本文采用单因子试验对影响SRAP-PCR反应的各个反应参数(模板DNA、dNTPs、Taq DNA聚合酶、Mg2+浓度和引物)进行了优化。建立了野生毛木耳SRAP-PCR最佳的反应体系:即25 μL体系中包含Taq DNA聚合酶1.5 U、Mg2+ 1.5 mmol/L、dNTPs 0.48 mmol/L、模板DNA 25 ng、正反向引物浓度均为0.4 µmol/L,10×PCR buffer 2.5 μL,ddH2O 14.7 μL。然后采用最适反应体系筛选出10对扩增稳定、多态性丰富的SRAP引物组合,为野生毛木耳的遗传多样性分析及育种工作提供了理论基础。
毛木耳(Auricularia polytricha (Mont.)Sacc.属广布种,在世界的温带到热带地区均有分布,生长在多种阔叶树倒木、腐朽木、腐朽树桩以及活树的枯死部分或落枝上(Musngi et al., 2005; Yu et al., 2008),在森林生态系统中起着重要的分解作用。毛木耳与其近缘种木耳(Auricularia auricula-judae (Bull.) Quél.)营养丰富,是两种天然的食药用菌,因子实体的高蛋白低脂肪及丰富的粗纤维含量而深受广大消费者的青睐。现代药理学研究表明,毛木耳还具有抑制肿瘤、保护肝脏和提高免疫力等多方面的药用功能(戴玉成和杨祝良, 2008; 戴玉成等, 2010)。
SRAP (sequence-related amplified polymorphism, 相关序列扩增多态性)是由Li和Quiros (2001)研发的一种新型分子标记技术,通过独特的引物设计对ORFs (open reading frames)进行扩增,SRAP标记将RAPD和AFLP两者的优点有机地结合在一起,克服了RAPD、AFLP和SSR等分子标记中的许多不足(Budak et al., 2004),以其简便、稳定、中等产率、易于测序和在基因组中分布均匀等特点现已应用于金针菇(朱坚等, 2007)、香菇(应正河等, 2006)、灵芝(Sun et al., 2006)、毛木耳(Yu et al., 2008)及木耳(Tang et al., 2010)的菌株鉴定和遗传多样性分析中。
SRAP分子标记技术在栽培毛木耳的遗传多样性分析中虽有应用,但未见对其反应体系进行优化,而同一反应体系在不同的实验条件下,针对不同的DNA模板对扩增效果的影响较大。因此,实验前对反应体系的建立和优化是十分必要的。本文通过单因子试验对SRAP-PCR反应体系中的Taq DNA聚合酶、模板DNA、引物、Mg2+、dNTPs用量进行了优化,经多次重复比较试验,建立了野生毛木耳SRAP-PCR最佳反应体系。并从10条正向引物和13条反向引物共130对引物组合中筛选出适合野生毛木耳SRAP-PCR扩增的引物组合,旨在为野生毛木耳遗传多样性分析、遗传育种和品种改良等工作的开展奠定基础。
1结果与分析
1.1 Taq DNA聚合酶用量对SRAP扩增的影响
在SRAP-PCR反应中,Taq酶直接影响扩增反应的成功与否。当Taq酶用量过高时容易使非特异性产物增加,并加大实验成本;用量过低则反应不完全。本试验不同的Taq酶用量对扩增条带的数量影响不大(图1),但当Taq酶用量为0.5~1.0 U时,在250 bp、300 bp和700 bp处的条带较模糊,达到1.5 U时扩增条带最清晰,因此,Taq酶用量在1.5 U时为最佳条件。
.png) 图1 不同Taq DNA聚合酶用量对野生毛木耳(Cui 6654) SRAP-PCR扩增的影响 |
1.2模板DNA含量对SRAP扩增的影响
从图2可以看出,模板DNA量在12.5 ng时有些扩增条带较模糊,在25~75 ng时均能扩增出相同的带型。因此,25 ng的模板用量可以满足野生毛木耳SRAP–PCR反应的要求。
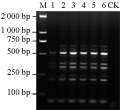 图2 不同模板DNA用量对野生毛木耳(Cui 6654) SRAP-PCR扩增的影响 |
1.3引物浓度对SRAP扩增的影响
引物的浓度影响扩增产物的质量,浓度过低,与模板的结合几率降低;浓度过高易形成引物二聚体。由图3可见,引物浓度在0.1 μmol/L时有些扩增条带较弱;0.2~0.5 μmol/L时均能扩增出清晰的条带,经多次重复比较试验,以0.4 μmol/L时,反应最稳定,条带清晰,故将引物浓度确定为0.4 μmol/L。
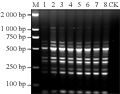 图3 不同引物浓度对野生毛木耳(Cui 6654) SRAP-PCR扩增的影响 |
1.4 Mg2+浓度对SRAP扩增的影响
Mg2+是Taq酶的激活剂,浓度过低,Taq酶的活性低;浓度过高,则使引物的错配频率增加。由图4可见,Mg2+浓度为0.5 mmol/L时仅扩增出3条弱带,1.0 mmol/L时扩增条带有所增加,但仍有缺带现象,且有些条带模糊不清,而当Mg2+浓度为1.5 mmol/L时扩增产物最为丰富,且条带最清晰,随着浓度增加,条带逐渐模糊,当浓度为3.0 mmol/L时缺失300 bp处的一条带。因此,最终确定为1.5 mmol/L的Mg2+浓度为最适浓度。
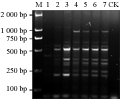 图4 不同Mg2+浓度对野生毛木耳(Cui 6654) SRAP-PCR扩增的影响 |
1.5 dNTPs浓度对SRAP扩增的影响
dNTPs浓度是SRAP-PCR扩增的一个主要变化因素,浓度过低,扩增产物减少,浓度过高会与Taq酶竞争Mg2+,其特异性和精确性降低。由图5可见,当dNTPs浓度为0.08 mmol/L时扩增产物最少;0.16~0.24 mmol/L时有严重的条带缺失现象,且多数条带模糊不清;当dNTPs浓度为0.32~0.56 mmol/L时扩增带型基本相同,但0.48 mmol/L时扩增条带最亮,最清晰,大于0.56 mmol/L时扩增条带较少,因此dNTPs浓度的较小变动就能引起PCR产物的较大改变。最终以0.48 mmol/L的dNTPs浓度为最佳。
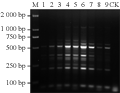 图5 不同dNTPs浓度对野生毛木耳(Cui 6654) SRAP-PCR扩增的影响 |
1.6引物筛选
引物的筛选工作量较大,操作繁琐,却是SRAP分析的重要环节。应用最佳反应体系,即25 μL反应体系中:模板DNA 25 ng、dNTPs 0.48 mmol/L、Taq DNA聚合酶1.5 U、Mg2+ 1.5 mmol/L,正反向引物浓度均为0.4 µmol/L,10×PCR buffer 2.5 μL,ddH2O 14.7 μL。采用130对SRAP引物组合(由10条正向引物和13条反向引物两两自由组合)对两号(22, 23)野生毛木耳菌株的基因组DNA分别进行扩增,结果有22对引物在两号模板DNA上均未扩增出条带,分别为m1-em12、m10-em3、m10-em4、m10-em12、m3-em1、m4-em1、m4-em4、m4-em5、m4-em8、m4-em9、m4-em10、m4-em11、m5-em3、m6-em5、m6-em7、m7-em10、m8-em6、m8-em7、m9-em2、m9-em3、m9-em5和m9-em12;有29对引物只在一号模板DNA上有扩增条带,分别为m1-em4、m2-em2、m2-em9、m3-em2、m3-em4、m3-em5、m3-em7、m4-em2、m4-em7、m5-em4、m5-em9、m5-em11、m6-em8、m6-em11、m7-em5、m7-em7、m7-em8、m7-em9、m7-em13、m8-em3、m8-em4、m8-em9、m8-em12、m9-em7、m9-em10、m9-em11、m9-em13、m10-em2和m10-em8。另有69对引物虽在两号模板DNA上都有扩增条带,但扩增条带要么数量较少、要么多态性较低、要么条带弥散、模糊不清或扩增不稳定,本试验均不采用。最终从130对引物组合中筛选出10对扩增条带数量较多、多态性较高、条带清晰稳定的引物组合有10对,分别为m1-em11、m2-em3、m2-em4、m2-em11、m3-em9、m7-em2、m8-em13、m10-em5、m10-em11和m10-em13。图6中编号为7,8的泳道为引物m3-em9组合对第22,23号模板DNA扩增的效果,泳道13~20分别为m8-em13、m10-em5、m10-em11和m10-em13对两号模板DNA的扩增效果。
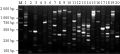 图6 10对引物组合对野生毛木耳SRAP-PCR扩增结果 |
1.7 SRAP-PCR反应体系的确立及稳定性鉴定
按照优化后的反应体系,从筛选出的较优引物组合中随机选取m2-em4,m2-em11两对组合对21号野生毛木耳菌株进行扩增验证。由图7可见扩增谱带清晰,菌株间有不同程度的多态性,重复试验也能得到一致结果,表明该体系稳定可靠,可满足对野生毛木耳基因组DNA进行SRAP分析的要求。
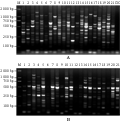 图7 m2-em4(A), m2-em11(B)引物对部分野生毛木耳菌株的扩增结果 |
2讨论
SRAP分子标记技术在食药用真菌的遗传多样性分析中虽有应用,但仅检索到关于香菇(应正河等, 2006; 付立忠等, 2006)和松口蘑(吴松权等, 2009)反应体系优化的报道,而在植物中关于SRAP-PCR反应体系的建立与优化的报道较多(黄春琼等, 2009; 邹枚伶等, 2009; 郑婷婷等, 2010)。PCR反应各组分(Taq DNA聚合酶、模板DNA、引物、Mg2+和dNTPs)的浓度都会影响到PCR结果的灵敏度和稳定性,本试验所优化的反应体系与已经报道的体系有一定的差异,所用Taq酶用量和引物浓度与香菇的大致相同,却高于松口蘑SRAP-PCR反应的用量(1.5 U, 0.3 µmol/L);Mg2+浓度低于香菇和松口蘑(2.5 mmol/L),dNTPs浓度却高于两者(0.2 mmol/L, 0.25 mmol/L)。Mg2+浓度影响Taq酶活性的发挥,浓度过低时无扩增产物出现或得到的产物偏少,过高会增加引物错配几率,导致非特异性的扩增,本试验Mg2+的最适用量为1.5 mmol/L,与油茶(郑婷婷等, 2010) SRAP-PCR反应体系中的用量相同,也与在野生毛木耳ISSR-PCR反应体系的建立与优化实验中得出的野生毛木耳的PCR扩增需要较低的Mg2+浓度的结论是一致的。dNTPs直接影响PCR扩增产物的生成,浓度过低使扩增反应不完全,本试验浓度在0.16~0.24 mmol/L时有严重的条带缺失现象,且多数条带模糊不清,以0.48 mmol/L为最佳。在遗传多样性研究中反应体系的稳定性和重复性很重要,因此,采用m2-em4和m2-em11两对引物组合按照优化后的反应体系,对21号野生毛木耳菌株进行了扩增验证(图7),重复试验也能得到一致结果,表明建立的体系稳定可靠,可满足对后续野生毛木耳遗传多样性SRAP分析的要求。
通常,扩增程序对PCR扩增效果影响较大,但由于SRAP引物具有通用性,所以无论在植物、动物还是微生物的应用上所用的反应程序大致相同,只是预变性的时间和扩增结束后保存的温度有些差异,对SRAP-PCR扩增的结果影响不大。因此,本实验没有对反应程序进行优化。SRAP引物虽具有通用性,但引物组合并不适用于所有物种,针对不同的实验材料所筛选出的最佳引物组合是不同的。本试验从130对引物组合中仅筛选出10对较优组合,其他组合要么无扩增条带,要么扩增条带较少或多态性较差,都没有采用;而王守现等(2006)人从153对引物中选取14对扩增效果较好、稳定性较强的引物对六个鸡腿菇菌株的多态性进行SRAP分析;朱坚等(2007)从153对引物组合中最终选出7对扩增条带数目较多、多态性好的引物组合对金针菇的种质资源进行SRAP分析,可以看出大多数引物组合不可用,尽管引物筛选工作繁琐,实验前对引物组合进行筛选还是十分必要的。
3材料与方法
3.1供试菌株
本研究所用野生毛木耳标本采自于广东、台湾、海南和辽宁4个省份,各标本的采集地及寄主情况如表1所示。将标本带回室内自然阴干或于28℃的烘箱烘干,干燥后即可用于干耳组织分离菌种,经1~2次的纯化培养,置于4℃冰箱保存。
 表1 供试野生毛木耳菌株 |
3.2主要试剂及仪器
PCR扩增所用试剂(dNTPs和Taq DNA聚合酶)及DL2000 Marker均购自Promega生物技术有限公司。所用SRAP引物序列参考朱坚等(2007)和Yu等(2008),由英骏生物技术有限公司(invitrogen)合成(表2)。
 表2 SRAP引物序列 |
MagNA Lyser全自动组织匀浆机、eppendorf centrifuge 5415R低温离心机和Smart SpecTm Plus紫外可见分光光度计(Bio-Rad)用于野生毛木耳基因组DNA提取及浓度检测;Mastercycler PCR仪(Eppendorf)、电泳仪(Bio-Rad)、DYY-6C电泳槽和凝胶成像仪(Bio-Rad)用于基因组SRAP-PCR扩增、电泳及检测。
3.3基因组DNA的提取
基因组DNA的提取与检测按照杜萍等(2010)的方法进行。
3.4 SRAP扩增反应条件优化
3.4.1 SRAP-PCR基础扩增反应体系
基础扩增反应体系如表3所示,稳压80 V,电泳80~ 90 min。其它电泳方法及条件同前文(杜萍等, 2010)。
 表3 SRAP-PCR基础扩增反应体系 |
3.4.2 PCR扩增反应程序
反应程序为94℃预变性3 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,50℃退火l min,72℃延伸1 min,35个循环;72℃延伸8 min;4℃保存。
3.4.3 SRAP-PCR反应体系的优化试验设计
从供试菌株中随机选取来自海南乐东的第22号(Cui 6654)野生毛木耳菌株,以其基因组DNA为模板从引物初筛中选择能扩增出条带的引物组合m2-em4,按照基础扩增反应体系,对模板DNA含量、Taq DNA聚合酶用量、正反向引物浓度、Mg2+和dNTPs浓度5个影响SRAP-PCR扩增的主要因素进行单因子试验,各反应参数作6~9个水平的梯度优化(表4),每个优化好了的参数直接用于下一个优化参数的反应体系中,且每次PCR反应均设有阴性对照。
 表4 SRAP-PCR反应体系优化设计 |
3.5引物筛选
利用优化后得出的最佳反应体系对引物进行筛选。将10条正向引物和13条反向引物的130对引物组合分别与地理位置较远的第22号(Cui 6654)和第23号(Dai 11007)DNA模板进行扩增,以筛选出扩增条带丰富、多态性高、重复性好的引物组合。
3.6 SRAP-PCR反应体系的稳定性鉴定
从筛选出的较优组合中随机选取2个不同的引物组合对21号野生毛木耳菌株的基因组DNA进行扩增,以鉴定所优化体系的稳定性和可靠性。
作者贡献
作者杜萍是本研究的实验设计和实验研究的执行人;杜萍完成数据分析,论文初稿的写作;作者崔宝凯参与实验设计,试验结果分析;崔宝凯是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
作者十分感谢北京林业大学的戴玉成、李海蛟、王伟、司静等人在标本采集、菌株分离、纯化及DNA提取等方面所提供的帮助。本研究由国家自然科学基金(30910103907)、中央高校基本科研业务费专项资金(BLYJ200901)共同资助。感谢两位匿名的同行评审人的评审意见和修改建议。
参考文献
Budak H., Shearman R.C., Parmaksiz I., and Dweikat I., 2004, Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs, SSRs, RAPDs, and SRAPs, Theor. Appl. Genet., 109(2): 280-288 doi:10.1007/s00122-004-1630-z
Dai Y.C., and Yang Z.L., 2008, A revised checklist of medicinal fungi in China, Junwu Xuebao (Mycosystema), 27(6): 801-824 (戴玉成, 杨祝良, 2008, 中国药用真菌名录及部分名称的修订, 菌物学报, 27(6): 801-824)
Dai Y.C., Zhou L.W., Yang Z.L., Wen H.A., Bau T., and Li T.H., 2010, A revised checklist of edible fungi in China, Junwu Xuebao (Mycosystema), 29(1): 1-21 (戴玉成, 周丽伟, 杨祝良, 文华安, 图力古尔, 李泰辉, 2010, 中国食用菌名录, 菌物学报, 29(1): 1-21)
Du P., Cui B.K., and Dai Y.C., 2010, Establishment and optimization of ISSR-PCR reaction system for Auricularia polytricha, Shengwu Jishu Tongbao (Biotechnology Bulletin), 215(6): 130-137 (杜萍, 崔宝凯, 戴玉成, 2010, 野生毛木耳ISSR-PCR反应体系的建立与优化, 生物技术通报, 215(6): 130-137)
Fu L.Z., Wei H.L., Li H.B., Wu Q.Q., Wu D.F., and Wu X.Q., 2006, Optimization of a sequence-related amplified polymorphism (SRAP) amplification system for Lentinula edodes, Shiyongjun Xuebao (Acta Edulis Fungi), 13(4): 10-15 (付立忠, 魏海龙, 李海波, 吴庆其, 吴大丰, 吴学谦, 2006, 香菇SRAP扩增体系的建立与优化, 食用菌学报, 13(4): 10-15)
Huang C.Q., Zhou S.Y., Liu G.D., and Bai C.J., 2009, The optimization of SRAP-PCR system of Bermuda grass, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 28(5): 981-989 (黄春琼, 周少云, 刘国道, 白昌军, 2009, 狗牙根SRAP-PCR反应体系的优化, 基因组学与应用生物学, 28(5): 981-989)
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103(2): 455-461doi:10.1007/s001220100570
Musngi R.B., Abella E.A., Lalap A.L., and Reyes R.G., 2005, Four species of wild Auricularia in Central Luzon, Philippines as sources of cell lines for researchers and mushroom growers, J. Agricultural Tech., 1(2): 279-299
Sun S.J., Gao W., Lin S.Q., Zhu J., Xie B.G., and Lin Z.B., 2006, Analysis of genetic diversity in Ganoderma population with a novel molecular marker SRAP, Appl. Microbiol. Biotechnol., 72(3): 537-543 doi:10.1007/s00253-005-0299-9 PMid:16411085
Tang L.H., Xiao Y., Li L., Guo Q., and Bian Y.B., 2010, Analysis of genetic diversity among Chinese Auricularia auricula cultivars using combined ISSR and SRAP markers, Curr. Microbiol., 61(2): 132-140 doi:10.1007/s00284-010-9587-4 PMid:20127246
Wang S.X., Liu Y., Geng X.L., Meng L.L., and Xie B.G., 2006, Polymorphism analysis of six Coprinus strains using a novel molecular marker SRAP, Jiangxi Nongye Daxue Xuebao (Acta Agiculturae Universitatis Jiangxiensis), 28(5): 753-757 (王守现, 刘宇, 耿小丽, 孟莉莉, 谢宝贵, 2006, 应用SRAP标记对六个鸡腿菇菌株的多态性分析, 江西农业大学学报, 28(5): 753-757)
Wu S.Q., Piao X.C., Quan X.L., Jin M.Y., 2009, Optimization of SRAP reaction systems for Tricholoma matsutake, Liaoning Linye Keji (Journal of Liaoning Forestry Science & Technology), (5): 13-14, 34 (吴松权, 朴炫春, 全雪丽, 金美玉, 2009, 松口蘑SRAP反应体系的优化, 辽宁林业科技, (5): 13-14, 34)
Ying Z.H., Wu X.P., Xie B.G., Chen L.N., Lu Q.Q., Gao W., and Sun S.J., 2006, Optimization of a sequence-related amplified polymorphism (SRAP) reaction system for Lentinula edodes, Shiyongjun Xuebao (Acta Edulis Fungi), 13(4): 1-5 (应正河, 吴小平, 谢宝贵, 陈丽娜, 卢启泉, 高巍, 孙淑静, 2006, 香菇SRAP反应体系的优化, 食用菌学报, 13(4): 1-5)
Yu M.Y., Ma B., Luo X., Zheng L.Y., Xu X.Y., and Yang Z.R., 2008, Molecular diversity of Auricularia polytricha revealed by inter-simple sequence repeat and sequence-related amplified polymorphism markers, Curr. Microbiol., 56(3): 240-245 doi:10.1007/s00284-007-9067-7 PMid:18180993
Zheng T.T., Lin P., Wang K.L., Yao X.H., and Yang S.P., 2010, Optimization of SRAP-PCR system for Camellia oleifera, Linye Kexue Yanjiu (Forest Research), 23(2): 302-307 (郑婷婷, 林萍, 王开良, 姚小华, 杨水平, 2010, 油茶SRAP-PCR反应体系的优化, 林业科学研究, 23(2): 302-307)
Zhu J., Gao W., Lin B.D., Sun S.J., Zheng Z., Yang J., and Xie B.G., 2007, SRAP analysis of Flammulina velutipes germplasm resources, Fujian Nonglin Daxue Xuebao (Ziran Kexueban) (Journal of Fujian Agriculture and Forestry University(Natural Science Edition)), 36(2): 154-158 (朱坚, 高巍, 林伯德, 孙淑静, 郑昭, 杨洁, 谢宝贵, 2007, 金针菇种质资源的SRAP分析, 福建农林大学学报 (自然科学版), 36(2): 154-158)
Zou M.L., Xia Z.Q., Ji J.M., Chen X., Wang H.Y., Zeng C.Y., and Wang W.Q., 2009, Establishment of SRAP-PCR reaction conditions in Aquilaria sinensis (Lour.) Spreng, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 28(1): 137-140 (邹枚伶, 夏志强, 吉家敏, 陈新, 王海燕, 曾长英, 王文泉, 2009, 白木香SRAP-PCR反应体系的建立, 基因组学与应用生物学, 28(1): 137-140)