 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 22 篇 doi: 10.5376/gab.cn.2011.30.0022
收稿日期: 2011年04月02日 接受日期: 2011年05月04日 发表日期: 2011年06月09日
引用格式(中文):
李煌等, 2011,植物响应低温胁迫的信号转导和转录调控机制,基因组学与应用生物学(online), Vol.30 No.22 pp.1139-1145 (doi: 10.5376/gab.cn.2011.30.0022)
引用格式(英文):
Li et al., 2011, The signal transduction and transcriptional regulation mechanism of plant to low-temperature stress, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.22 pp.1139-1145 (doi: 10.5376/gab.cn.2011.30.0022)
低温是典型的植物非生物胁迫,直接影响到许多植物的生长和地域分布。植物对低温胁迫的响应是一种积极主动的应激过程。利用现代生物学技术阐述其分子机制已成为研究热点。本文对响应过程中的信号转导进行归纳总结,并重点介绍了近年来发现的在转录控制中起关键作用的调控因子及相关的调控体系,最后说明该方向研究存在的问题和展望,从而为植物低温胁迫分子机制的深入研究提供参考和借鉴。
低温是最为常见的环境胁迫因素之一,低温胁迫一方面能直接降低生物反应速率,阻碍代谢反应,另一方面间接地通过基因表达调控影响细胞的代谢机制。在非致死低温下处理一段时间, 植物可以获得抵抗或降低低温伤害的能力,这个过程叫做冷驯化。在此过程中,低温信号被膜系统识别后,经一系列信号分子和多种传递途径,引发大量低温诱导基因和相关蛋白质的表达。其中转录调控承接着上游低温信号的感知和下游特异蛋白质的表达,是整个复杂信号网络的重要组成部分。本文对信号转导和转录调控机制的研究成果进行综述, 有助于启迪研究思路,为植物冷驯化分子机制研究提供理论参考。
1低温胁迫响应中的信号转导
植物细胞膜蛋白可以通过感知由低温引起的在膜的流动性,蛋白质和核酸构象及代谢产物聚集状态等方面的变化去感知低温胁迫(Orvar et al., 2000; Los and Murata, 2004; Vaultier et al., 2006)。Sangwan等(2001)研究发现,在芸薹(Brassica napus L.)的冷驯化过程中,质膜出现固化(rigidation)现象后,才检测到低温诱导的COR (cold-regulated gene)基因的表达。研究还发现,在紫花苜蓿的细胞悬浮培养中,当加入可以显著增加细胞膜流动性的苯甲醇时,冷诱导基因的表达和Ca2+流动在4℃时受到抑制,相反地,当加入膜固化剂二甲基亚砜后,冷诱导基因的表达和Ca2+流动能在25℃即被诱导(Orvar et al., 2000; Sangwan et al., 2001)。以上结果证明植物细胞能够通过细胞膜固化去感知低温胁迫。
钙离子信号途径是目前研究最为透彻的信号转导途径。植物响应低温过程中有暂时的胞质钙离子增加,钙调蛋白可以通过感受到这种增加,进而改变不同蛋白质的磷酸化状态,行使信号传递功能。例如,磷脂酶C (phospholipase C, PLC)通过水解磷脂酰肌醇二磷酸(phosphatidylinositol-4-monophos-phate, PIP2),产生信号分子1,4,5-三磷酸肌醇(inositol 1,4,5-trisphosphate, IP3)来传递信息(Vergnolle et al., 2005)。同时,胞质钙离子的增加或者与钙调蛋白结合可以激活钙依赖蛋白激酶,引起基因表达的变化,进而导致相应的生理变化(Komatsu et al., 2007)。
除了钙离子,其它信号分子也参与了低温信号转导。例如,低温条件下,脱落酸(abscisic acid, ABA)合成缺失的拟南芥突变体los5-1中一些低温诱导基因的表达水平显著降低,从而不能抵抗低温,初步推测ABA可能通过协同Ca2+信号体去影响低温信号的传递(Xiong et al., 2001)。活性氧(reactive oxygen species, ROS)在受到低温及其它非生物胁迫的细胞内显著增加,可能作为一种信号传递体,直接通过激活相关转录因子和蛋白激酶发挥作用(Lee et al., 2002a)。另外,低温诱导产生的其它代谢物质也能作为引发基因表达的信号体。例如,脯氨酸能诱导许多启动子含有脯氨酸响应元件的基因的表达(Satoh et al., 2002)。
总之,植物低温胁迫信号转导是一个涉及到众多信号分子和调控途径的网络体系。不同的信号途径可以通过某个特定结点进行交叉对话,实现综合调控的目标。目前,依托模式植物拟南芥为平台,对低温胁迫下的野生型植株和相应突变体或过量表达植株进行比对分析,获取特定的转导信息是十分重要的研究途径。
2低温胁迫响应中的转录调控
通过转录因子调控功能基因的表达是植物应答低温胁迫的关键环节。根据调控体系是否涉及CBF因子(C-repeat binding factors)的参与,通常可将其分为CBF途径和非依赖CBF途径(图1)。
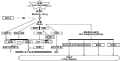 图1 植物中的低温信号传导调控体系
|
2.1 CBF途径
2.1.1 CBF简介
CBF转录因子是一类受低温诱导的,最初在拟南芥中发现的反式作用因子,它可以与CRT/DRE顺式作用元件特异性结合,激活启动子中具有这一调控元件的冷诱导和脱水诱导基因的表达,诱导抗寒及抗旱的发生(Stockinger et al., 1997)。目前发现的与低温响应密切相关的CBF因子有4个,即CBF1、CBF2、CBF3和CBF4。其中CBF1、CBF2和CBF3受低温诱导,启动子中都含有核心序列CANNTG,编码蛋白质都含AP2-DNA结合域(Liu et al., 1998)。CBF4基因编码的蛋白质与其它3种CBF蛋白有63%的同源性,在AP2/ERF-DNA结合区域中则有91%~94%的同源性,CBF4不受低温诱导,但其过量表达也可增强转基因拟南芥植株的抗寒性(Haarke et al., 2002)。在以CBF为“总开关”的复杂调控机制中,存在其他许多调节因子调控CBF或受到CBF调控(图1)。
2.1.2 CBF上游调控因子
当拟南芥处于低温环境中时,CBF基因的转录水平能在15 min内迅速提高,2 h后含有CRT/DRE元件的一组COR基因被诱导表达(Stockinger et al., 1997)。CBF对低温的快速响应,说明CBF基因的诱导表达无需新蛋白质的合成。于是Gilmour等(1998)认为存在一种能作用于CBF基因的启动子上的转录因子,命名为ICE (inducer of CBF expression)。Chinnusamy等(2003)对拟南芥突变体ice1进行图文克隆首次分离到ICE1基因。ICE1基因编码1个MYB类型的螺旋-环-螺旋(HLH)转录因子,它通过结合CBF3启动子中的MYC顺式作用元件,激活冷驯化过程中CBF3和COR基因的表达,从而提高转基因植株的抗寒性和冷驯化能力。另外,对CBF2启动子进行突变分析鉴定出两个新的片段,分别命名为ICEr1和ICEr2。该发现预示植物的冷驯化过程中可能存在其它ICE-类似蛋白协同调控CBF基因的表达(Fursova et al., 2009; Zarka et al., 2003)。
利用拟南芥突变体鉴定分析调控因子及其功能极大促进了冷驯化机制的研究。Agarwal等(2006)发现经过MYB15 (编码一种R2R3-MYB家族蛋白)敲除的突变植株在冷驯化中表现出CBFs的高水平表达,而在过量表达MYB15的转基因拟南芥中表现出CBFs表达水平的下降及抗寒能力的降低。因此,MYB15能对CBFs进行负调控。有趣的是,在低温胁迫下,相对于野生型植株,突变体ice1表现出较高的MYB15转录水平,可见ICE1能对MYB15进行负调控(Vogel et al., 2005)。利用酵母双杂交技术分析发现MYB15也能作用于ICE1,但ICE1和MYB15两者的相互作用在冷驯化机制中具体发挥什么功能尚不清楚(Agarwal et al., 2006)。
Lee等(2001)发现拟南芥突变体hos1 (high expression of osmotically responsive gene 1)暴露于低温环境中一段时间后表现出CBF2、CBF3及下游低温响应基因的诱导量增加,表明HOS1可能参与到对CBF2、CBF3的负调控。研究证实,HOS1是一种E3泛素连接酶,当植株受到低温胁迫时,HOS1积聚到细胞核中,并通过与磷酸化的ICE1相互作用介导ICE1的泛素化降解,从而抑制CBFs及其下游调控基因的表达(Dong et al., 2006)。与HOS1相对的是拟南芥细胞中还存在另一种E3连接酶SIZ1,它可能通过ICE1的蛋白SUMO化减缓HOS1参与的降解过程,增强胞内ICE1的稳定性,间接地参与到CBFs的上游调控(Miura et al., 2007)。同时,HOS2/FIERY1 (FIERY编码一种1-肌醇多磷酸磷酸酶,此酶调节IP3的降解)也具有类似HOS1的调控功能,研究发现低温胁迫下突变体hos2中CBF2和CBF3的转录水平远高于野生型植株(Xiong et al., 2004)。
Xiong等(2002)利用生物荧光标记遗传筛选,鉴定了两种新的突变体fry2 (FIERY2)和los2 (low expression of osmotically responsive genes)。FIERY2能编码一种新的转录抑制因子,该因子含有2个dsRNA结合区域(可能作用于mRNA的形成)和一个类似RNA多聚酶II-CTD (羧基末端结构域)磷酸化酶的区域。该区域的突变能导致含有CRT/DRE序列的基因的显著增加。LOS2通过作用与c-myc基因有相似启动子序列的STZ/ZAT10 (一种能特异结合EP2元件序列的锌指蛋白基因),来抑制STZ/ZAT10的活性,从而负调控低温响应基因的表达(Davletova et al., 2005)。
Doherty等(2009)研究表明钙调控蛋白转录激活因子CAMTA可能是低温响应过程中钙信号转导和冷调控基因表达的结合位点,在CBF2的启动子中存在ICE1和CAMTA的结合位点,进一步推断两种转录因子可能相互调控CBF,诱导COR基因的表达。
基于以上研究成果,可以看出ICE1与其它大部分CBF上游调控因子存在直接或间接的调控关系。另外,盐胁迫和ABA胁迫也能增强ICE1的表达,ICE1可能参与到ABA-介导的CBF基因的表达调控(Chinnusamy et al., 2003)。因此,深入研究分析ICE1调控机理,一方面有助于冷驯化CBF调控网络的完善,另一方面能为植物抗逆机制的进一步研究提供突破点。
2.1.3 CBF下游调控因子
除了受到上游众多基因的调控,CBF还可通过调节其它因子间接地调控COR基因表达(图1)。许多启动子中含有CRT/DRE元件的低温诱导基因,如LTI78、COR47、COR15a和RD29A等可被转录因子CBF或DREB家族成员激活,引发低温诱导基因的表达(Yamaguchi-Shinozaki and Shinozaki, 2006)。
Knight等(2008)利用基因敲除及分子标记技术,筛选到冷冻敏感型拟南芥突变体sfr6 (sensitive to freezing 6),研究发现,低温胁迫下sfr6中含有CRT/DRE元件的COR基因的诱导量显著降低,而CBF的表达量未发生变化,表明SFR6可能通过影响CBF下游信号通路的一个组分,或通过影响与COR基因表达密切相关的独立信号通路中的某一组分来提高植物抗寒能力(Boyce et al., 2003)。同样,Maruyama等(2004)发现过量表达CBF3的转基因植株中STZ/ZAT10的表达量相应得到增强。相反地,在拟南芥突变体ice1中,CBF3表达缺失能降低STZ/ZAT10的冷诱导水平(Chinnusamy et al., 2003)。因此推断ZAT10的表达可能是受CBF调控,进而调控下游与冷驯化相关基因的表达。瞬时表达分析表明,STZ/ZAT10通过抑制CBF目的基因RD29A的表达来参与到调控体系(Lee et al., 2002b)。
另外,研究发现冷驯化过程中,AP2蛋白RAP2.1和RAP2.6的诱导表达晚于CBF1-3的表达,其中RAP2.1的启动子中含有CRT/DRE元件的核心序列CCGAC,且在组成型表达CBF1-3的转基因拟南芥中具有较高的表达量,预示RAP2.1可能是CBF转录激活因子的目标蛋白(Okamuro et al., 1997)。
2.1.4 CBF之间的交叉调控
CBF因子之间的交叉调控对调控CBF自身的表达水平也起着重要作用。Novillo等(2004)对CBF2缺失的拟南芥突变体进行分子生物学分析表明,在冷驯化过程中CBF2是CBF1和CBF3的负调控因子。另外研究发现,突变体ice1中,CBF3表达受到抑制的同时,CBF2的表达有一定量的增加。因此,CBF也可能负调控CBF2的表达(Chinnusamy et al., 2003)。在非胁迫条件下,CBF2是CBF1和CBF3转录水平的5~8倍,3种CBF因子的表达方式相同,只是非胁迫条件下恒量表达的CBF2抑制了CBF1和CBF3的表达,从而保证了CBF表达的瞬时性和严谨性(Novillo et al., 2004)。这些研究表明CBF因子之间的相互调控也参与到了整个调控体系。对cbf1和cbf3缺失突变体进行分析将有助于冷驯化过程中CBF交叉调控的研究。
2.2非依赖CBF途径
Fowler和Thomashow (2002)通过对野生型和过量表达CBF的拟南芥植株转录检测比对,发现至少有60种低温响应基因不受已发现的CBF家族转录因子所调控,预示植物冷驯化过程中存在非依赖CBF途径。
近年来,一系列参与非依赖CBF途径的低温胁迫调控因子被逐渐发现。例如,被鉴定为低温响应基因的ZAT12和RAV1,两者都具有与CBF相似的转录形式,分别编码一种功能未知的锌指蛋白和AP2-DNA结合因子(Davletova et al., 2005; Kagaya et al., 1999)。但两者的转录水平在CBF1-3组成型表达的植株中都不受影响,所以它们极可能通过类似CBF的途径发挥作用。
运用遗传筛选技术,另外两种转录因子HOS9和HOS10也被鉴定(Zhu et al., 2004; 2005)。HOS9基因编码一种位于核内的同源结构域转录因子。相对于野生型植株,低温处理提高了突变体hos9-1中内源RD29A基因和另外一些低温响应基因的转录水平;HOS10基因编码一种位于核中的R2R3型MYB转录因子,突变体hos10-1即使在低温胁迫中出现一些COR基因表达量的增加,但其抗寒能力比野生型更弱。根据hos9-1和hos10-1突变体中多个COR基因的表达量增加的同时,CBF的积累却没有明显变化进行推断,HOS9和HOS10可能是作为COR基因的负调控因子,通过非依赖途径CBF起作用。
通过遗传突变发现另一个对低温胁迫有负调控作用的基因是ESK1 (Eskimo1)。拟南芥突变体esk1中亮氨酸迅速积累,耐寒能力也相应增强(Xin and Browse, 1998)。对esk1中表达发生改变的312种基因进行转录分析鉴定,只有12种基因在esk1突变体中和过量表达CBF2的植株中同时存在(Xin et al., 2007)。因此,ESK1参与的冷驯化过程与CBF调控的冷驯化可能具有完全不同的分子机制。另外,低温,干旱和盐胁迫的上游调控因子MYB4和OsMYB3R-2也能够通过非依赖CBF途径正调控水稻的低温胁迫和其它非生物胁迫(Vannini et al., 2004; Dai et al., 2007)。
非依赖CBF途径的发现和研究进一步预示了植物冷驯化调控机制的复杂性。同时也表明植物体内存在多种不同的调控途径来响应各种逆境胁迫。而且,不同逆境胁迫下,植物可能通过某些特定的调控因子或途径来建立联系(Kreps et al., 2002)。
3展望
近年来,随着基因组测序、基因芯片、基因的差异表达分析及基因敲除和突变体筛选分析等技术日趋完善,植物冷驯化过程中大量基因被鉴定以及未知功能的新的基因及途经被发现,同时这些进展也反映出目前我们对植物低温响应机制的了解仅仅只是冰山一角。
正是由于植物的冷驯化是一个涉及多基因、多信号途径、多基因产物的复杂过程,所以单个基因功能的阐明不足以弄清整个低温响应调控机制,单个基因的导入也不足以提高整个植株抗寒能力。由于低温胁迫的众多方面都受到转录调控,因此许多转录因子被选作为作物抗寒工程中的目标工具。比如CBF因子已经成功地被用于许多物种的抗逆基因工程。同时,我们还应更多关注低温响应中不同转录因子之间,顺式作用元件与反式作用因子之间及冷胁迫和其它非生物胁迫之间的相互关系。
另外,转录控制只是复杂的植物冷驯化信号网络的一个方面,还应对转录控制上游的胁迫信号的跨膜感知和识别,转录后翻译以及下游的胁迫相关蛋白质的功能进行研究,才能绘制出精细的低温应答调控体系。最后,为了能有效地应用低温诱导因子去提高作物的抗寒能力,新的策略及技术手段亟待发明。
作者贡献
李煌完成文献资料的收集,查阅,文章的撰写;杨华提供整体写作思路和参考资料;张金梅参与文章的修改;杜一凡参与文章中图表的绘制;饶力群本文的通讯作者,指导文章的写作,审阅和修改。
致谢
感谢湖南省自然科学基金(08JJ3066)和湖南省研究生科研创新项目的资助;感谢两位匿名的评审专家的评审和修改建议。
参考文献
Agarwal M., Hao Y.J., Kapoor A., Dong C.H., Fujii H., Zheng X.W., and Zhu J.K., 2006,A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance, J. Biol.Chem., 281(49): 37636-37645 doi:10.1074/jbc.M605895200 PMid:17015446
Boyce J.M., Knight H., Deyholos M., Openshaw M.R., Galbraity D.W., Warren G., and Knight M.R., 2003, The sfr6 mutant of Arabidopsis is defective in transcriptional activation via CBF/DREB1 and DREB2 and shows sensitivity to osmotic stress, Plant J., 34(4): 395-406 doi:10.1046/j.1365-313X.2003.01734.x PMid:12753580
Chinnusamy V., Ohta M., Kanrar S., Lee B.H., Hong X.H., Agarwal M., and Zhu J.K., 2003, ICE1: A regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis, Gene Dev., 17(8): 1043-1054 doi:10.1101/gad.1077503
Dai X.Y., Xu Y.Y., Ma Q.B., Xu W.Y., Wang T., Xue Y.B., and Chong K, 2007, Over expression of an R1R2R3 MYB gene, OsMYB3R-2, increases tolerance to freezing, drought, and salt stress in transgenic Arabidopsis, Plant Physiol., 143(4): 1739-1751 doi:10.1104/pp.106.094532 PMid:17293435 PMCid:1851822
Davletova S., Schlauch K., Coutu J., and Mittler R., 2005, The zinc-finger protein Zat12 plays a central role in reactive oxygen and abiotic stress signaling in Arabidopsis, Plant Physiol., 139(2): 847-856 doi:10.1104/pp.105.068254 PMid:16183833 PMCid:1256000
Doherty C.J., van Buskirk H.A., Myers S.J., and Thomashow M.F., 2009, Roles for Arabidopsis CAMTA transcription factors in cold-regulated gene expression and freezing tolerance, The Plant Cell, 21(3): 972-984 doi:10.1105/tpc.108.063958 PMid:19270186 PMCid:2671710
Dong C.H., Agarwal M., Zhang Y.Y., Xie Q., and Zhu J.K., 2006, The negative regulator of plant cold responses, HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1, Proc. Natl. Acad. Sci., USA, 103(21): 8281-8286 doi:10.1073/pnas.0602874103 PMid:16702557 PMCid:1472463
Fowler S., and Thomashow M.F., 2002, Arabidopsis transcriptome profiling indicates that multiple regulatory pathways are activated during cold acclimation in addition to the CBF cold response pathway, The Plant Cell, 14(8): 1675-1690 doi:10.1105/tpc.003483 PMCid:151458
Fursova O.V., Pogorelko G.V., and Tarasov V.A., 2009, Identification of ICE2, a gene involved in cold acclimation which determines freezing tolerance in Arabidopsis thaliana, Gene, 429(1-2): 98-103 PMid:19026725
Gilmour S.J., Zarka D.G., Stockinger E.J., Salazar M.P., Houghton J.M., and Thomashow M.F., 1998, Low temperature regulation of the Arabidopsis CBF family of AP2 transcriptional activators as an early step in cold-induced COR gene expression, The Plant Journal, 16(4): 433-442 doi:10.1046/j.1365-313x.1998.00310.x PMid:9881163
Haarke V., Cook D., Riechmann J.L., Pineda O., Thomashow M.F., and Zhang J.Z., 2002, Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis, Plant Physiol., 130(2): 639-648 doi:10.1104/pp.006478 PMid:12376631 PMCid:166593
Kagaya Y., Ohmiya K., and Hattori T., 1999, RAV1, a novel DNA-binding protein, binds to bipartite recognition sequence through two distinct DNA-binding domains uniquely found in higher plants, Nucleic Acids Res., 27(2): 470-478 doi:10.1093/nar/27.2.470 PMid:9862967 PMCid:148202
Knight H., Mugford S.G., Garton S.G., Ulker B., Gao D., Thorlby G., and Knight M.R., 2008, Identification of SFR6, a key component in cold acclimation acting post-translationally on CBF function, The Plant Journal, 58: 97-108 doi:10.1111/j.1365-313X.2008.03763.x PMid:19067974
Komatsu S., Yang G., Khan M., Onodera H., Toki S., and Yamaguchi M., 2007, Over-expression of calcium-dependent protein kinase 13 and calreticulin interacting protein 1 confers cold tolerance on rice plants, Molecular Genetics and Genomics, 227(6): 713-723 doi:10.1007/s00438-007-0220-6 PMid:17318583
Kreps J.A., Wu Y., Chang H.S., Zhu T., Wang X., and Harper J.F., 2002, Transcriptome changes for Arabidopsis in response to salt, osmotic, and cold stress, Plant Physiol., 130(4): 2129-2141 doi:10.1104/pp.008532 PMid:12481097 PMCid:166725
Lee B.H., Lee H., Xiong L., and Zhu J.K., 2002a, A mitochondrial complex I defect impairs cold regulated nuclear gene expression, The Plant Cell, 14(6): 1235-1251 doi:10.1105/tpc.010433 PMCid:150777
Lee H., Guo Y., Ohta M., Xiong L., Stevenson B., and Zhu J.K., 2002b, LOS2, a genetic locus required for cold-responsive gene transcription encodes a bi-functional enolase, EMBO J., 21(11): 2692-2702 doi:10.1093/emboj/21.11.2692 PMid:12032082 PMCid:126021
Lee H., Xiong L., Gong Z., Ishitani M., Stevenson B., and Zhu J.K., 2001, The Arabidopsis HOS1 gene negatively regulates cold signal transduction and encodes a RING finger protein that displays cold-regulated nucleo-cytoplasmic partitioning, Genes Dev., 15(7): 912-924 doi:10.1101/gad.866801
Liu Q., Kasuga M., Sakuma Y., Abe H., Miura S., Yamaguchi-Shinozaki K., and Shinozaki K., 1998, Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought- and low- temperature-responsive gene expression, respectively, in Arabidopsis, The Plant Cell, 10(8): 1391-1406 doi:10.2307/3870648 PMid:9707537 PMCid:144379
Los D.A., and Murata N., 2004, Membrane fluidity and its roles in the perception of environmental signals, Biochimica et Biophysica Acta (BBA), 1666(1-2): 142-157
Maruyama K., Sakuma Y., Kasuga M., Ito Y., Seki M., Goda H., Shimada Y., Yoshida S., Shinozaki K., and Yamaguchi-Shinozaki K., 2004, Identification of cold-inducible downstream genes of the Arabidopsis DREB1A/CBF3 transcriptional factor using two microarray systems, The Plant Journal, 38(6):982-993 doi:10.1111/j.1365-313X.2004.02100.x PMid:15165189
Miura K., Jin J.B., Lee J., Yoo C.Y., Stirm V., Miura T., Ashworth E.N., Bressan R.A., Yun D.J., and Hasegawa P.M., 2007, SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis, The Plant Cell, 19(4): 1403-1414 doi:10.1105/tpc.106.048397 PMid:17416732 PMCid:1913760
Novillo F., Alonso J.M., Ecker J.R., and Salinas J., 2004, CBF2/DREB1C is a negative regulator of CBF1/DREB1B and CBF3/DREB1A expression and plays a central role in stress tolerance in Arabidopsis, Proc. Natl. Acad. Sci., USA, 101(11): 3985-3990 doi:10.1073/pnas.0303029101 PMid:15004278 PMCid:374356
Okamuro J.K., Caster B., Villarroel R., van Montagu M., and Jofuku K.D., 1997, The AP2 domain of APETALA2 defines a large new family of DNA binding proteins in Arabidopsis, Proc. Natl. Acad. Sci., USA, 94(13): 7076-7081 doi:10.1073/pnas.94.13.7076
Orvar B.L., Sangwan V., Omann F., and Dhindsa R.S., 2000, Early steps in cold sensing by plant cells: The role of actin cytoskeleton and membrane fluidity, The Plant Journal, 23(6): 785-794 doi:10.1046/j.1365-313x.2000.00845.x PMid:10998189
Sangwan V., Foulds I., Singh J., and Dhindsa R.S., 2001, Cold-activation of Brassica napus BN115 promoter is mediated by structural changes in membranes and cytoskeleton, and requires Ca2+ influx, The Plant Journal, 27(1): 1-12 doi:10.1046/j.1365-313x.2001.01052.x PMid:11489178
Satoh R., Nakashima K., Seki M., Shinozaki K., and Yamaguchi-Shinozaki K., 2002, ACTCAT, a novel cis-acting element for proline- and hypoosmolarity-responsive expression of the ProDH gene encoding proline dehydrogenase in Arabidopsis, Plant Physiol., 130(2): 709-719 doi:10.1104/pp.009993 PMid:12376638 PMCid:166600
Stockinger E.J., Gilmour S.J., and Thomashow M.F., 1997, Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcription activator that binds to the C repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit, Proc. Natl. Acad. Sci., USA, 94(3): 1035-1040 doi:10.1073/pnas.94.3.1035
Vannini C., Locatelli F., Bracale M., Magnani E., Marsoni M., Osnato M., Mattana M., Baldoni E., and Coraggio I., 2004, Over expression of the rice Osmyb4 gene increases chilling and freezing tolerance of Arabidopsis thaliana plants, The Plant Journal, 37(1): 115-127 doi:10.1046/j.1365-313X.2003.01938.x PMid:14675437
Vaultier M.N., Cantrel C., Vergnolle C., Justin A.M., Demandre C., Benhassaine-Kesri G., Cicek D., Zachowski A., and Ruelland E., 2006, Desaturase mutants reveal that membrane rigidification acts as a cold perception mechanism up stream of the diacylglycerol kinase pathway in Arabidopsis cells, FEBS Lett., 580(17): 4218-4223 doi:10.1016/j.febslet.2006.06.083 PMid:16839551
Vergnolle C., Vaultier M.N., Taconnat L., Renou J.P., Kader J.C., Zachowski A., and Ruelland E., 2005, The cold-induced early activation of phospholipase C and D pathways determines the response of two distinct clusters of genes in Arabidopsis cell suspensions, Plant Physiol., 139(3): 1217-1233 doi:10.1104/pp.105.068171 PMid:16258011 PMCid:1283760
Vogel J.T., Zarka D.G., van Buskirk H.A., Fowler S.G., and Thomashow M.F., 2005, Roles of the CBF2 and ZAT12 transcription factors in configuring the low temperature transcriptome of Arabidopsis, The Plant Journal, 41(2): 195-211 doi:10.1111/j.1365-313X.2004.02288.x PMid:15634197
Xin Z., and Browse J., 1998, Eskimo1 mutants of Arabidopsis are constitutively freezing tolerant, Proc. Natl. Acad. Sci., USA, 95(13): 7799-7804 doi:10.1073/pnas.95.13.7799
Xin Z.G., Mandaokar A., Chen J.P., Last R.L., and Browse J., 2007, Arabidopsis ESK1 encodes a novel regulator of freezing tolerance, The Plant Journal, 49(5): 786-799 doi:10.1111/j.1365-313X.2006.02994.x PMid:17316173
Xiong L., Ishitani M., Lee H., and Zhu J.K., 2001, The Arabidopsis LOS5/ABA3 locus encodes a molybdenum cofactor sulfurase and modulates cold stress- and osmotic stress-responsive gene expression, The Plant Cell, 13(9): 2063-2083 doi:10.2307/3871428 PMid:11549764 PMCid:139452
Xiong L., Lee H., Huang R.F., and Zhu J.K., 2004, A single amino acid substitution in the Arabidopsis FIERY1/HOS2 protein confers cold signaling specificity and lithium tolerance, The Plant Journal, 40(4): 536-545 doi:10.1111/j.1365-313X.2004.02225.x PMid:15500469
Xiong L., Lee H., Ishitani M., Tanaka Y., Stevenson B., Koiwa H., Bressan R.A., Hasegawa P.M., and Zhu J.K., 2002, Repression of stress-responsive genes by FIERY2, a novel transcriptional regulator in Arabidopsis, Proc. Natl. Acad. Sci., USA, 99(16): 10899-10904 doi:10.1073/pnas.162111599 PMid:12149453 PMCid:125070
Yamaguchi-Shinozaki K., and Shinozaki K., 2006, Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses, Annu. Rev. Plant Biol., 57: 781-803 doi:10.1146/annurev.arplant.57.032905.105444 PMid:16669782
Zarka D.G., Vogel J.T., Cook D., and Thomashow M.F., 2003, Cold induction of Arabidopsis CBF genes involves multiple ICE (inducer of CBF expression) promoter elements and a cold-regulatory circuit that is desensitized by low temperature, Plant Physiol., 133(2): 910-918 doi:10.1104/pp.103.027169 PMid:14500791 PMCid:219064
Zhu J., Shi H., Lee B.H., Damsz B., Cheng S., Stirm V., Zhu J.K., Hasegawa P.M., and Bressan R.A., 2004, An Arabidopsis homeodomain transcription factor gene, HOS9, mediates cold tolerance through a CBF-independent pathway, Proc. Natl. Acad. Sci., USA, 101(26): 9873-9878 doi:10.1073/pnas.0403166101 PMid:15205481 PMCid:470766
Zhu J., Verslues P.E., Zheng X., Lee B.H., Zhan X., Manabe Y., Sokolchik I., Zhu Y., Dong C.H., Zhu J.K., Hasegawa P.M., and Bressan R.A., 2005, HOS10 encodes an R2R3-type MYB transcription factor essential for cold acclimation in plants, Proc. Natl. Acad. Sci., USA, 102(28): 9966-9971 doi:10.1073/pnas.0503960102 PMid:15994234 PMCid:1175003