 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 24 篇 doi: 10.5376/gab.cn.2011.30.0024
收稿日期: 2011年03月17日 接受日期: 2011年05月05日 发表日期: 2011年05月13日
引用格式(中文):
姜宝杰等, 2011,利用SMART法构建花生果针全长cDNA文库,基因组学与应用生物学(online), Vol.30 No.24 pp.1155-1161 (doi: 10.5376/gab.cn.2011.30.0024)
引用格式(英文):
Jiang et al., 2011, Construction of peanut gynophore full-length cDNA library with SMART method, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.24 pp.1155-1161 (doi: 10.5376/gab.cn.2011.30.0024)
果针是花生实现地上开花、地下结果的关键器官,果针的研究历来受到植物学家的重视。为进一步筛选、克隆和分析果针特异基因及研究花生果针发育的分子机理,我们以花生果针为材料,利用改良的CTAB法提取总RNA,根据SMARTTM cDNA文库构建试剂盒所示方法合成cDNA,限制性酶切消化后连接到质粒载体pDNR-LIB,构建花生果针全长cDNA文库。经鉴定,原始文库的滴度为1.3×106 cfu,重组率达100%,60%的插入片段在1.0~2.0 kb之间,插入片段平均大小在1.0 kb左右,扩增文库滴度达到了3.82×109 cfu。说明所构建的花生果针文库属高质量的全长cDNA,为筛选和克隆花生果皮特异表达基因和研究花生果针发育的分子机理提供了基础。
花生(Arachis hypogaea L.)是我国重要的油料和经济作物,果针是花生特有的组织结构,是实现花生地上开花、地下结荚的器官,果针能否入土,关系到花生能否正常结果。因此,果针生长发育的生物学特性引起许多学者的重视。目前对于果针的研究主要集中于栽培(张海燕等, 2004)及生长发育方面(贺立红等, 2006)。花生果针分子生物学方面的研究报道甚少,目前对于控制花生果针结构、生长发育以及其它特性的基因表达调控情况还是一片空白。
cDNA文库的构建和筛选是基因克隆的重要方法之一,是目前发现新基因和研究基因表达的基本工具(朱利军等, 2009, 海南大学学报自然科学版, 27(2): 185-190; 董志敏等, 2006, 作物杂志, (5): 1-4)。例如郭俣等(2010)人利用油鸡cDNA文库成功筛选并鉴定了purH基因的表达情况。全长cDNA文库的构建可以高效、大规模获得基因序列,能大幅度地加快计算机分析蛋白质表达和功能的进程,是进行基因组研究的一条重要途径(董志敏等, 2006)。目前,全长文库构建方法已比较成熟,报道的技术方法有Oligo-Capping法(Maruyama and Sugano, 1994)、CAPture法(Edery et al., 1995)、CAP-trapper法(Carninci et al., 1996)、SMART法(Chenchik et al., 1996)、Cap-Select法(Schmidt and Mueller, 1999)和CAP-jumping法(Fimove et al., 2001)等。近年来,许多研究者致力于全长文库的构建,拟南芥(Seki et al., 1998)、水稻(刘运华等, 2007)、花生(蔡宁波等, 2007)、甘蔗(许莉萍等, 2009)和鹿茸(郝丽等, 2009)等均有全长文库的报道,获得了大量的数据,完善了各种文库构建方法,极大的促进了功能基因组学的研究。然而,各种文库构建方法各有优缺点,综合比较而言,SMART法因其具有简单、快速的特点已广泛应用于一般质量cDNA文库的构建。Clontech公司改进的“SMART”法使构建文库所需的RNA量减少,大约只需50 ng总RNA即可构建完整的cDNA文库(储昭晖等, 2002, 科学通报, 47(21): 1656-1662)。
本文利用SMART技术成功构建了花生果针全长cDNA文库,该文库可用于克隆和鉴定一批功能基因全长cDNA序列,为开展果针重要基因的功能研究奠定了基础,将有利于进一步从分子水平深入阐明果针生长发育的分子机理。
1结果与分析
1.1总RNA的提取结果
利用本实验室改良的CTAB法提取的花生果针总RNA经1.0%的琼脂糖凝胶电泳结果(图1)显示,28S rRNA和18S rRNA条带清晰,说明提取的总RNA质量较高。紫外分光光度计检测所提RNA纯度,其OD260/OD280值为1.97,OD260/OD230值为2.32,获得了高纯度的RNA,满足文库构建的要求。
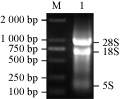 图1 花生果针总RNA电泳图 |
1.2 dscDNA的合成
3 μg总RNA起始第一链合成后采用LD-PCR法合成的ds cDNA经1.0%琼脂糖凝胶电泳检测,结果如图2所示,ds cDNA片段条带呈现分布在0.2~3.0 kb (图2)之间的弥散状条带,主要集中于750 bp以上,条带清晰、正常且产物的量足,说明合成了较高质量的双链cDNA,可用于cDNA文库的构建。
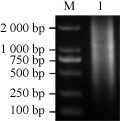 图2 花生果针dsDNA电泳图 |
1.3文库质量的鉴定
双链经蛋白酶K消化、Sfi Ⅰ酶切、连接载体、电击转化后涂板,计数培养板单克隆数,经计算原始文库得到1.23×106个重组子。随机挑取15个单菌落摇菌,进行菌液PCR扩增,电泳结果显示(图3),其中含有两个双插入,重组率达100%。随机挑取10个单克隆过夜摇菌后提取质粒,酶切后电泳(图4)显示,6个单克隆的插入片段大小在1.0~2.0 kb之间,所占比例为60%,插入片段平均大小在1.0 kb左右,达到文库质量要求。扩增文库经不同稀释倍数稀释后涂板过夜培养,经计算扩增文库滴度结果为3.82×109 cfu。以上数据表明该文库为较高质量的cDNA文库,可作为筛选和克隆果针特异表达基因基因、研究果针发育分子机理的平台。
图3 花生果针全长cDNA文库菌液PCR电泳图 |
 图4 花生果针全长cDNA文库单菌落质粒酶切电泳图 |
1.4文库随机测序及生物信息学分析结果
随机挑选文库30个单克隆进行双向测序,利用DNA序列常规分析软件BioXM 2.6分析测序结果,共有22个基因测通,且均具有开放阅读框结构。借助生物信息学技术判断序列是否为全长,结果有9个为全长序列,比率为40.9%。利用BLASTN和BLASTX程序(http://blast. ncbi nlm. nih. gov/blas.t cgi)进行同源性检索,初步确定基因的功能,所测序列包含结合类蛋白、氧化系统蛋白、结构蛋白、转录因子、抑制类蛋白、酶类蛋白和过敏原等基因(表1)。
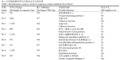 表1 文库随机测序序列生物信息学分析结果 |
2讨论
要成功构建一个全长cDNA文库,获得高质量的RNA是前提。而影响总RNA质量的因素主要有3个:一是否有降解;二是否有污染(DNA、蛋白质和小分子物质);三是否完整,即包括生命体所有基因的转录产物。花生富含酚和多糖等物质,使用RNA提取试剂盒或者其它通用RNA提取技术都很难获得高质量的RNA,本实验室改良的CTAB法提取花生RNA虽然耗时较长,但提取的RNA纯度较高。本实验所提取的果针RNA 28S、18S和5.8S 3条条带清晰,经紫外分光光度计检测,OD260/OD280=1.97,OD260/OD230=2.32说明所提取的RNA较纯,没有降解,没有蛋白质等分子污染,可成功应用于该文库的构建。
构建高质量的全长cDNA文库要求获得高质量的全长cDNA。本实验应用SMART方法,以3 μg总 RNA为模板起始第一链的合成,采用LD-PCR法合成双链可保证获得的cDNA序列的完整性。王淑红等经过大量测序证实了SMART法对cDNA序列的完整性的保证(王淑红等, 2008, 台湾海峡, 27(3): 278-285)。
cDNA文库的质量具体反映在两个方面:文库的库容量和插入cDNA片段的大小(易朝辉, 2010)。本实验所构建的原始cDNA文库具有的独立克隆数为1.3×106个,包括了大部分稀有的mRNA,可以满足大规模测序和筛选所用;插入片断均大于750 bp,60%的插入片段高于1 000 bp,平均长度为1.0 kb,保证了高比例全长cDNA的获得。
本实验利用SMART技术成功构建了高质量的花生果针全长cDNA文库,滴度测定、重组率、PCR鉴定结果均符合cDNA文库的质量要求。随机测序序列生物信息学分析表明本文库包含各类基因,全长率较高,可用于花生果针功能基因的筛选和测序。本文库的构建为下一步果针功能基因的研究以及筛选、克隆果针次生代谢产物合成途径相关基因奠定了基础。
3材料与方法
3.1材料
本研究选用福建农林大学油料作物所选育的优良花生品种闽花6号在大田种植入土前果针为材料,-70℃冰箱保存备用。
文库构建试剂盒CreatorTM SMARTTM cDNA Library Construction Kit购自Clontech公司。Primer Script Reverse Transcriptase、Ex Taq,dNTP,DL2000分子标准、DL15000分子标准和大肠杆菌DH5α感受态细胞均购自大连宝生物公司(TAKARA)。胶回收试剂盒购自杭州博日科技有限公司(BIOER)。其它试剂采用国产分析纯。
3.2花生果针总RNA的提取
取5 g花生果针于液氮预冷的研钵中研磨成粉状,转移至65℃含预热CTAB的离心管中,水浴20~30 min,氯仿/异戊醇抽提2次后,上层水相加入LiCl沉淀总RNA。4℃12 000 g离心30 min,将沉淀物溶于50 μL DEPC水即获得总RNA。总RNA经紫外分光光度计检测浓度及纯度,经1.0%琼脂糖凝胶电泳检测其完整性。
3.3 cDNA单链和双链的合成
单链cDNA的合成:取3 μg总RNA于DEPC处理离心管中,按照SMART文库构建试剂盒说明加入相应的引物和试剂合成cDNA第一链。采用LD-PCR法合成双链cDNA。按照SMART文库构建试剂盒说明添加单链模板、酶、引物及dNTP后用双蒸水补足至25 μL。反应在已预热到95℃的PCR仪上进行。反应程序:95℃预变性2 min;95℃变性15 s,68℃复性30 s,72℃延伸5 min,共8个循环;72℃延伸10 min。取2 μL反应产物进行琼脂糖凝胶电泳检测其质量。
3.4蛋白酶K与Sfi I限制性酶切消化及文库构建
50 μL双链cDNA的PCR产物经蛋白酶K消化后抽提两次,用80%乙醇室温沉淀。沉淀溶于79 μL去离子水中,加入15 μL Sfi Ⅰ酶于50℃水浴酶切。酶切产物经琼脂糖凝胶电泳后回收750 bp以上片段。回收产物经乙醇、糖原-20℃共沉淀后溶于7 μL去离子水,-70℃保存备用。
回收产物在T4 DNA连接酶作用下,与质粒载体pDNR - LIB(经 Sfi Ⅰ酶处理)连接,电击转化入感受态细胞(大肠杆菌DH5α)后迅速转移至LB培养基中,37℃ 225 r/min震荡培养1 h获得原始文库。大肠杆菌感受态细胞的制备和连接转化等操作参考分子克隆实验指南(萨姆布鲁克和拉塞尔, 2002)。
3.5原始文库质量的鉴定
取1 μL原始文库菌液至99 μL液体LB培养基中混匀,均匀地涂在含30 mg/L氯霉素的LB培养板上。计数过夜培养板单克隆数,原始文库库容(pfu)=生长克隆数×103。
随机挑取15个单菌落,过夜摇菌后做菌液PCR鉴定文库重组率。随机挑取11个单菌落过夜摇菌后提取质粒,质粒经SfiⅠ酶切后在1.0%的琼脂糖凝胶上电泳检测文库cDNA插入片段的大小。随机挑取30个单菌落做穿刺stock并送上海国家人类基因组南方研究中心进行双向测序。
3.6文库的扩增及滴度的检测
依据原始文库库容,将原始文库菌液均匀地涂在几个含30 mg/L氯霉素的LB培养板,37℃过夜培养。每个培养板用10 μL液体LB培养基洗脱后混匀,即为扩增文库。加入等量50%甘油混匀后分装,-80℃保存。
取1 μL扩增文库菌液,稀释103、104和105倍后吸取100 μL均匀地涂在含30 mg/L氯霉素的LB培养板,37℃过夜培养。文库的滴度公式(cfu)=(平均每个培养板中单菌落数×1 000 μL/mL)/涂布稀释的菌体体积(μL)。
3.7序列分析
30个随机挑取的单克隆测序后,依据引物序列去除载体序列,利用DNA序列常规分析软件BioXM 2.6进行拼接,分析其开放阅读框(open reading frame, ORF)和非编码区(untranslated regions, UTRs),最后利用BLASTN和BLASTX程序(http://www.ncbi.nlm.nih.gov/)进行同源性检索,初步确定基因的功能。
作者贡献
姜宝杰为本文章主要完成人,该文章所涉及的实验及文章写作是在在庄伟建老师的指导下完成的,同时陈华、邓烨、曾建斌、张冲及贺小彦等同学为实验提供了技术和操作上的帮助,在此一并表示感谢。
致谢
本实验得到了福建省科技厅:基因工程改良花生油亚比(O/L)的研究(2008I0002)的资金支持,再次表示感谢。同时感谢两位匿名同行评审人的评审意见和修改建议。
参考文献
Cai N.B., Huang X.W., and Zhuang W.J., 2007, Construction and identification of a full-length cDNA library from peanut seeds, Huasheng Xuebao (Journal of Peanut Science), 36(2):1-5 (蔡宁波, 黄湘文, 庄伟建, 2007, 花生种子全长cDNA文库的构建和鉴定, 花生学报, 36(2): 1-5)
Carninci P., Kvam C., Kitamura A., Ohsumi T., Okazaki Y., Itoh M., Kamiya M., Shibata K., Sasaki N., Izawa M., Muramatsu M., Hayashizaki Y., and Schneider C., 1996, High efficiency full length cDNA cloning by biotinylated CAP trapper, Genomics, 37(3): 327-336 doi:10.1006/geno.1996.0567 PMid:8938445
Chenchik A., Moqadam F., and Siebert P., 1996, A new method for full-length cDNA cloning by PCR, In: Krieg P.A. (ed.), A laboratory guide to RNA: Isolation, analysis, and synthesis, New York, pp.273-321
Dong Z.M., Zhang B.S., Guan R.X., Chang R.Z., and Qiu L.J., 2006, The methods of constructing the full length cDNA library, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 22(2): 51-55 (董志敏, 张宝石, 关荣霞, 常汝镇, 邱丽娟, 2006, 全长cDNA文库的构建方法, 中国农学通报, 22(2): 51-55)
Edery I., Chu L.L., Sonenberyg N., and Pelletier J., 1995, An efficient strategy to isolate full-length cDNAs based on an mRNA cap retention procedure (CAPture), Moecularl Cell Biology, 15(6): 3363-3371
Fimove V.A., Chakhmakhcheva O.G., Archdeacon J., Fernandez J.M., Fedorkin O.N., Dorokhov Y.L., and Atabekov J.G., 2001, Detection of the 5'-cap structure of messenger RNAs with the use of the CAP-jumping approach, Nucleic Acids Research, 29(22): 4751-4759 doi:10.1093/nar/29.22.4751 PMid:11713326 PMCid:92527
Guo Y., Liu C.Q., Lu T.F., Tong C.L., Xiu X.N., Guan W.J., and Ma Y.H., 2010, Construction and characterization of a cDNA library from Beijing fatty chicken and expression analysis of purH gene, Nanjing Nongye Daxue Xuebao (Journal of Nanjing Agricultural University), 33(4):93-99 (郭俣, 刘长青, 陆涛峰, 佟春玲, 修晓娜, 关伟军, 马月辉, 2010, 北京油鸡cDNA文库的构建及purH基因的表达, 南京农业大学学报, 33(4):93-99)
Hao L., Li H.P., and Yan L., 2009, Construction of full-length cDNA library for antler tip tissue of sika deer, Dongbei Linye Daxue Xuebao (Journal of Northeast Forestry University), 37(11):120-122 (郝丽, 李和平, 严厉, 2009, 东北梅花鹿鹿茸尖端组织全长cDNA文库的构建, 东北林业大学学报, 37(11):120-122)
He L.H., Cai M., He S.G., Lan X., Zheng Y.X., Xiong X.C., and Lan S.W., 2006, Effects of relevant enzymes on lignification of Arachis hypogaea gynophore, Huanan Nongye Daxue Xuebao (Journal of South China Agricultural University), 27(1): 76-78 (贺立红, 蔡马, 何生根, 兰霞, 郑奕雄, 熊兴嫦, 蓝石维, 2006, 相关酶对花生果针木质化的影响, 华南农业大学学报, 27(1): 76-78)
Liu Y.H., Liu Z.C., Zhou L.G., Yu S.W., Liu H.Y., and Luo L.J., 2007, Construction of full length cDNA library from drought-tolerant rice cv. Zhonghan No.3 cultivated under drought stress, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 5(3): 367-370 (刘运华, 刘灶长, 周立国, 余舜武, 刘鸿艳, 罗利军, 2007, 干旱胁迫下耐旱稻中旱3号全长cDNA文库的构建, 分子植物育种, 5(3): 367-370)
Maruyama K., and Sugano S., 1994, Oligo-capping: A simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides, Gene, 138(1-2): 171-174 doi:10.1016/0378-1119(94)90802-8
Sambrook J., and Russell D.W., eds., Huang P.T., trans., 2002, Molecular cloning: A laboratory manual, 3rd, China Science Press, Beijing, China, pp.93-96 (萨姆布鲁克, 拉塞尔, 主编, 黄培堂, 主译, 2002, 分子克隆实验指南, 第三版, 科学出版社, 中国, 北京, pp.93-96)
Schmidt W.M., and Mueller M.W., 1999, CapSelect: A highly sensitive method for 5’ CAP-dependent enrichment of full-length cDNA in PCR-mediated analysis of mRNAs, Nucleic Acids Research, 27(21): e31 doi:10.1093/nar/27.21.e31
Seki M., Carninci P., Nishiyama Y., Hayashizaki Y., and Shinozaki K., 1998, High-efficiency cloning of Arabidopsis full-length cDNA by biotinylated CAP trapper, The Plant Journal, 15(5): 707-720 doi:10.1046/j.1365-313x.1998.00237.x PMid:9778851
Xu L.P., Que Y.X., Liu J.X., Guo J.L., Zheng Y.F., Xu J.S., Yuan Z.N., Chen P.H., and Chen R.K., 2009, Construction of full-length cDNA library from sugarcane leaves and the corresponding EST analysis, Nongye Shengwu Jishu Xuebao (Journal of Agricultural Biotechnology), 17(5): 843-850 (许莉萍, 阙友雄, 刘金仙, 郭晋隆, 郑益凤, 徐景升, 袁照年, 陈平华, 陈如凯, 2009, 甘蔗叶片全长cDNA文库构建及EST序列分析, 农业生物技术学报, 17(5): 843-850)
Yi Z.H., 2010, Construction and identification of cDNA library of heart tissue from Yunan pig, Xumu Yu Siliao Kexue (Animal Husbandry and Feed Science), 31(5): 1-2 (易朝辉, 2010, 豫南黑猪心脏cDNA文库的构建与鉴定, 畜牧与饲料科学, 31(5): 1-2)
Zhang H.Y, Wang M.L., Liu Z.M., and Liang Q.S., 2004, Effects of AnM cultivate technique on bud differentiation and gynophore elongation of peanut, Laiyang Nongxueyuan Xuebao (Journal of Laiyang Agricultural College), 21(3): 203-205 (张海燕, 王铭伦, 刘赞谟, 梁全松, 2004, 花生控制下针(AnM)栽培法对花芽分化及果针生长的影响, 莱阳农学院学报, 21(3): 203-205)