 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 28 篇 doi: 10.5376/gab.cn.2011.30.0028
收稿日期: 2011年05月16日 接受日期: 2011年05月26日 发表日期: 2011年06月15日
引用格式(中文):
董迎辉等, 2011,5个缢蛏野生种群遗传变异的AFLP分析,基因组学与应用生物学(online), Vol.30 No.28 pp.1182-1188 (doi: 10.5376/gab.cn.2011.30.0028)
引用格式(英文):
Dong et al., 2011, Genetic variation of 5 wild populations of Sinonovaula constricta by AFLP analysis, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.28 pp.1182-1188 (doi: 10.5376/gab.cn.2011.30.0028)
为了深入了解我国缢蛏主产区浙、闽沿海缢蛏野生种群的遗传多样性和遗传分化水平,科学开展种质资源的评价、保护和持续利用。本研究利用9对引物组合对5个缢蛏野生种群(NH, WL, YQ, XP和DS) 150个个体的基因组DNA进行了AFLP扩增,共得到345个清晰的扩增位点,其中多态性位点312个,多态位点比例高达90.43%。Nei基因多样性指数和Shannon’s信息指数反映的遗传多样性分析表明,5个缢蛏种群的遗传多样性水平均较高,各种群的遗传多样性大小依次为XP>YQ>NH>WL>DS,相比之下XP和YQ种群的遗传多样性最高。基因分化系数Gst和AMOVA分析均表明,5个缢蛏种群间的遗传分化不明显,遗传变异主要源自种群内个体间。群体间遗传距离和聚类分析表明,福建的XP和DS群体间的遗传距离最近(0.006 3),聚为一支,而浙江的NH、WL和YQ群体间的遗传距离也较近(0.007 6~0.012 3),聚为另外一支,这说明2省的缢蛏种群已经发生了一定的遗传分化。 本研究为我国主产区缢蛏种群的种质资源评价提供参考,为缢蛏的合理保护和持续利用提供科学依据。
缢蛏(Sinonovaula constricta)属广温性滩涂埋栖型双壳贝类,在我国南北沿海均有分布,是我国传统4大养殖贝类之一,尤其在浙江和福建两省沿海滩涂及虾塘养殖最为广泛。近年来,缢蛏养殖业呈快速发展趋势,养殖区从浙江、福建向周边的江苏、山东和广东等省份迅速拓展。据不完全统计,2008年缢蛏全国总产量达到74.20万吨,养殖总面积约6.16万公顷,其中浙江省的养殖产量和养殖面积分别达到24.73万吨和1.78万公顷,位居全国第一,其次为山东和福建(刘增胜和柳正, 2009)。然而,伴随着缢蛏养殖业的快速发展,异地引种养殖给缢蛏种质资源保护和管理带来很大困难,这使得加强种群遗传结构分析显得尤为重要和迫切。
国内外有关缢蛏群体遗传学方面的研究报道已有一些,如王冬群等(2005)采用同工酶技术比较了象山缢蛏养殖群体和野生群体遗传多样性的差异;姜志勇等(2007)分析了福建缢蛏野生群体与养殖群体的ITS-1和ITS-2序列变异;李成华等(2004b)和于颖等(2007)利用RAPD (random amplified polymorphism DNA, 随机扩增多态性DNA)标记技术分别对缢蛏宁波长街群体和大连群体的遗传结构进行了分析;牛东红等(2007; 2008; 2009)采用线粒体16S rRNA、COI基因序列分析和ISSR分子标记技术对缢蛏多个群体进行了遗传多样性和遗传关系分析。与同工酶、RAPD和ISSR技术相比,本文采用的AFLP标记技术兼具RFLP的准确性和PCR的高效性,具有高效简便、灵敏度高、多态性丰富、结果稳定可靠、遗传方式符合孟德尔定律等优点,可在短时间内获得大量遗传信息,已被广泛用于遗传多样性分析、种质鉴定、遗传连锁图谱构建、目的基因的克隆和定位等诸多遗传学研究领域(张留所等, 2003)。在贝类方面,已有不少用AFLP技术研究不同地理群体或育种群体间遗传变异的报道(潘洁等, 2002; 张雯等, 2005; Liu et al, 2007;林志华等, 2008, 代悦等, 2010),但目前采用AFLP技术进行缢蛏群体遗传学研究的报道尚未见到。本研究对浙、闽沿海5个缢蛏野生地理种群的基因组DNA进行AFLP分析,旨在深入了解我国缢蛏主产区缢蛏种群的遗传多样性和遗传分化水平,评价种质资源现状,为其合理保护和持续利用提供科学依据。
1结果与分析
1.1 AFLP选择性扩增结果
利用9对多态性高的选择性引物组合,对5个缢蛏野生种群的150个个体的基因组DNA进行了AFLP扩增,结果得到345个清晰位点,多态性位点为312个,多态性位点比例高达90.43%。不同引物组合的扩增位点数(26~48)和多态位点比例(80.77%~94.87%)存在较大差别(表1),平均每对引物扩增出38.3个位点,其中34.7个为多态性位点。引物组合E45M54、E45M51和E35M54扩增出的多态性位点数较多(41~44),多态性位点比例也较高(91.67%~95.35%),是较好的扩增引物选择;而E32M54和E45M48的多态性条带和多态性位点比例均较低。
.png) 表1 9对AFLP选择性引物的扩增结果比较 |
1.2 5个缢蛏野生种群的AFLP扩增带谱和遗传多样性分析
图1是E35M54引物组合对5个缢蛏野生种群60个体的扩增电泳图谱。从图谱中可以看出,AFLP扩增片段大多在450 bp~1 500 bp之间,其中多态性位点比例很高;5个地理种群之间的扩增位点差异不大,未发现特征性位点。这表明,缢蛏种群内保持了较大的遗传变异,而种群间亲缘关系较近。
 图1 引物组合E35M54对5个缢蛏野生种群部分个体的扩增图谱 |
从表2可以看出,不同种群的扩增位点数差别不大,NH、WL、YQ、XP和DS分别为290、289、297、291和282,其对应的多态性比例依次为84.06%、83.77%、86.09%、84.35%和81.74%。其中YQ种群的多态性位点最多,多态性位点比例最高,而DS种群的多态性位点数最少,多态性位点比例最低。从Nei基因多样性指数和Shannon’s信息指数来看,各种群的遗传多样性由大到小依次为XP>YQ>NH>WL>DS。总体来讲,5个野生种群都保持了很高的遗传多样性,而相比之下XP和YQ种群的遗传多样性最高。
 表2 5个缢蛏种群的遗传结构和遗传多样性分析 |
1.3群体内和群体间遗传分化程度
如表3所示,群体间基因分化系数(Gst)结果显示,缢蛏群体内的遗传变异占总变异的95.61%,基因流(Nm)为10.885 9,可见5个地理种群间基因流动很大,遗传分化主要来源于种群内的个体间。应用AMOVA方法对缢蛏5个地理种群的遗传变异来源 进行分析,同样发现总的遗传分化系数Fst为0.022 9,2.29%的变异来自种群间,而97.71%的变异来自种群内个体间,这与上述Gst的变化趋势基本一致,这说明5个缢蛏野生群体间的变异不大,遗传分化不明显。
 表3 缢蛏5个地理种群的AMOVA分析 |
1.4不同缢蛏地理种群间的遗传相似性系数、相对遗传距离和聚类分析
利用Popgen 1.32软件,计算了5个缢蛏地理种群间的遗传相似性系数和无偏遗传距离(Nei, 1978),结果见表4。种群间的遗传相似性系数为0.979 5~0.993 7,遗传距离为0.006 3~0.020 8,其中福建的XP与DS种群间的遗传距离最近(0.006 3),其次WL与YQ群体间也较近(0.007 6)。而XP、DS与NH群体间的遗传距离均较大,分别达到0.020 8和0.015 9。
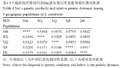 表4 5个缢蛏地理种群间的Nei遗传相似性系数和相对遗传距离 |
根据群体间Nei’s遗传距离,用MEGA 4.0软件构建了5个种群的UPGMA和NJ系统进化树(图2)。结果均显示,浙江的NH、WL和YQ群体聚为一支,而福建的XP和DS群体聚为另外一支,说明两省的缢蛏种群已经发生了一定的遗传分化。
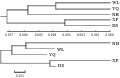 图2 根据群体间遗传距离用UPGMA法和NJ法构建的缢蛏5个种群的系统树 |
2讨论
2.1缢蛏群体的遗传多样性分析
遗传多样性是生物种内不同种群或同一种群不同个体之间遗传变异的总和,是生物生存适应和发展进化的重要基础。一般来讲,一个物种或种群的遗传多样性越丰富,它对环境变化的适应能力就越强。缢蛏是我国重要的滩涂经济贝类,有关其种群遗传多样性的研究已有不少报道,如邵艳卿等(2009)对浙江乐清湾缢蛏群体的AFLP分析发现,缢蛏的多态位点比例、Nei基因多样性指数和Shannon’s信息指数等遗传参数都比小荚蛏和大竹蛏大得多,而且个体间相对遗传距离最大,说明缢蛏种内核DNA遗传多样性水平很高;李成华等(2004b)、王冬群等(2005)和姜志勇等(2007)分别对比了宁波长街、象山、福建缢蛏野生群体和养殖群体间的遗传多样性差异,结果均表明野生群体的遗传多样性略高于养殖群体,而两群体间的遗传分化不明显;在缢蛏不同地理种群遗传多样性分析方面,我国学者已采用同工酶(王冬群等, 2005)、RAPD (李成华等, 2004a)、ISSR (牛东红等, 2009)和线粒体基因序列(牛东红等, 2007; 2008; 2010)等多种遗传标记技术,检测了不同种群的遗传多样性水平和群体间的遗传结构变异、遗传分化程度,为我国缢蛏种质资源管理和遗传育种研究提供了科学的理论依据。
本研究采用AFLP分子标记技术,对浙闽沿海5个缢蛏野生种群150个样品的遗传多样性进行了分析,5个种群扩增位点的多态位点比例为81.74%~86.09%,此参数比使用RAPD技术检测缢蛏4个野生群体(55.24%~72.38%)、养殖和自然群体(74.56%~75.42%)的多态位点比例高,也比用ISSR技术检测缢蛏6个野生群体(61.59%~72.46%)多态位点比例高,而与邵艳卿等(2009)用AFLP检测乐清湾群体多态位点比例(84.25%)的结果相近,这表明AFLP技术较RAPD和ISSR技术具有更高的多态性检出率,可提供丰富的遗传变异信息,更适合用于物种种质资源的遗传多样性分析。遗传多样性分析发现,缢蛏的Nei基因多样性指数平均值为0.269 9,高于海洋贝类的平均水平(0.147) (相建海, 1999),这说明缢蛏种群仍保持了较高的遗传多样性。5个野生种群的遗传多样性比较来看,霞浦和乐清群体的基因多样性指数较高,而各群体之间的遗传参数差异不大,这一方面与缢蛏物种在浙闽沿海的广泛连续分布、易于基因交流有关,另一方面与本实验采样群体的地理范围不大也有很大关系。
2.2 5个缢蛏种群间的遗传分化
群体间的遗传分化取决于地理隔离作用,距离越远分化就越明显(Wright, 1931)。本文为了考察我国缢蛏主产区浙江、福建种群的遗传多样性差异和遗传分化水平,分析了浙江的宁海、温岭、乐清和福建的霞浦、东山5个野生种群的遗传结构,Fst和Gst的结果均显示,种群内个体间的遗传变异占总变异的95.61%和97.71%,这说明5个缢蛏野生群体间的变异不大,遗传分化不明显。群体遗传学理论认为,基因流可以减少群体间的遗传分化,当群体间基因流大于1时,则能有效抑制由遗传漂变引起的遗传分化(Wright, 1931),而5个缢蛏种群间的基因流达到了10.885 9,说明种群间基因交流非常频繁,从而大大减少了群体间的遗传分化。
缢蛏为广温、广盐性滩涂贝类,在我国东海的浙江、福建天然资源丰富且连续分布,匍匐幼虫前的浮游幼虫期约5至7天,如遇风浪可作一定地理距离的迁移,这为近距离的基因交流创造了天然条件。加之,缢蛏是浙、闽沿海滩涂传统贝类养殖的主要对象,近年来随着缢蛏养殖业的快速发展,两省各地之间为解决苗种供应而进行的亲贝和苗种调运变得日益频繁,这种人工育苗和大规模人工养殖中逃逸的苗种势必会侵蚀当地野生种群的种质资源,造成种质的混杂和群体间的均质化现象,对野生种质资源的有效保护和可持续利用极为不利。因此,一方面要加强对养殖生产中外来苗种调运的监管和防止养殖群体累代人工繁殖造成的生产性状衰退,另一方面应尽快建立缢蛏的自然保护区和原种场,加强对当地野生种质资源的有效保护和合理利用。
3材料与方法
3.1实验材料
2010年8~10月,从浙江的宁波宁海、台州温岭、温州乐清和福建的宁德霞浦、漳州东山5个自然海域采集缢蛏(Sinonovaula constricta)野生群体样品。样品采集地、采样时间等基本信息列于表5。所取样品均活体运回实验室,每个群体随机取样30颗,解剖取足部肌肉,剪碎后用无水酒精固定2次,置于4℃冰箱中保存。
 表5 5个缢蛏野生群体的名称及来源 |
实验所用的限制性内切酶EcoRⅠ、Mse Ⅰ和T4 DNA连接酶购自Fermentas公司,PCR扩增体系购自大连宝生物工程有限公司,AFLP扩增所用接头和引物均由生工生物工程(上海)有限公司合成。
3.2实验方法
3.2.1基因组DNA的提取
取缢蛏的足部肌肉,采用传统的酚/氯仿法提取基因组DNA。获得的DNA样品用1%的琼脂糖电泳进行纯度和完整性检测,紫外分光光度仪(Varian Cary 50)定量,灭菌超纯水稀释至100 ng/μL。
3.2.2 AFLP标记的扩增与检测
AFLP实验流程参照Vos等(1995)的方法,略加改动。采用限制性内切酶EcoR I和Mse I进行双酶切,酶切产物用T4DNA连接酶连接人工接头(EcoR Ⅰ F: 5’-CTCGTAGACTGCGTACC-3’, EcoRⅠR: 5’-AAT TGGTACGCAGTC-3’; MseⅠF: 5’- GACGATGAGT CCTGAG-3’, MseⅠR: 5’-TACTCAGGACTCAT-3’),然后进行预扩增和选择性扩增两次PCR。选择扩增产物用4.5%变性聚丙烯酰胺胶电泳进行检测,60 W恒功率电泳2 h,银染法染色,胶版在室温下自然干燥,ScanMarker 9700XL扫描仪成像。
根据预试验结果,筛选出9对扩增带数适中、条带清晰、多态性高的选择性引物组合,对5个群体共150个个体进行了AFLP分析,各引物组合分别为E32M54、E32M58、E33M48、E33M58、E35M54、E42M48、E45M48、E45M51和E45M54,引物序列见表6。
 表6 AFLP扩增所用引物的序列 |
3.2.3数据处理与分析
根据缢蛏各种群AFLP标记的聚丙烯酰胺凝胶电泳检测结果,在相同的迁移位置上将图谱中每个扩增位点的有无转化为1、0 (出现记为“1”, 缺失记为“0”)数据矩阵,再用Popgen 1.32软件计算各种群的多态位点比例、Nei’s基因多样性指数(Nei, 1973)、Shannon’s信息指数、群体间基因分化系数和无偏相对遗传距离。利用Arlequin 3.0软件进行5个群体的分子方差分析(analysis of molecular variance,AMOVA),计算群体间遗传分化系数(Fst)。根据群体间相对遗传距离,用MEGA 4.0软件对5个缢蛏种群进行分子聚类分析,构建NJ和UPGMA系统进化树,分析群体间的亲缘关系。
作者贡献
董迎辉负责实验设计、样品采集、AFLP酶切和连接、实验结果分析及论文撰写;郑崇磊和姚定余完成了基因组DNA提取、PCR扩增和扩增产物的凝胶检测;姚韩韩负责实验数据的采集与处理;林志华负责实验指导、论文统稿。
致谢
本研究由国家现代贝类产业技术体系(nycytx-47)、宁波市自然科学基金(201001A6004021)和宁波市科技局农业择优委托项目(2010C10011)共同资助。在样品采集过程中,得到了台州学院孙长森副教授、浙江省海洋水产养殖研究所胡利华工程师和宁波市海洋与渔业研究院沈伟良助理研究员的热心帮助,在此一并表示感谢。同时感谢两位匿名的同行评审人的评审意见和修改建议。
参考文献
Dai Y., Yu Z. N., Zhao X. X., Wang Q. H., Du X. D., Wu Y. Z., 2010, Genetic structure of F1 among four shell color selective lines of pearl oyster Pinctada martensii based on AFLP analysis, Jiyinzuxue Yu Yingyong Shengwuxue (Genetics and Applied Biology), 29(2): 252-258 (代悦, 喻子牛, 赵晓霞, 王庆恒, 杜晓东, 吴燕忠, 2010, 马氏珠母贝4个壳色选育系F遗传结构的AFLP分析, 基因组学与应用生物学, 29(2): 252-258)
Jiang Z.Y., Niu D.H., Chen H., Shen H.D., and Li J.L., 2007, The genetic analysis of ITS2 and ITS2 between wild and cultured populations of Sinonovacula constricta in Fujian, Haiyang Yuye (Marine Fisheries), 29(4): 314-318 (姜志勇, 牛东红, 陈慧, 沈和定, 李家乐, 2007, 福建缢蛏野生群体与养殖群体的ITS-1和ITS-2分析, 海洋渔业, 29(4): 314-318)
Li C.H., Li T.W., Song L.S., and Su X.R., 2004b, Genetic variations among four populations of Sinonovacula constricta by using random amplified polymorphic DNA, Shuichan Kexue (Fisheries Science), 23(12): 26-28 (李成华, 李太武, 宋林生, 苏秀榕, 2004a, 4个缢蛏群体遗传结构的RAPD分析, 水产科学, 23(12): 26-28)
Li C.H., Li T.W., Su X.R., and Song L.S., 2004a, Genetic structure analysis of the natural and the cultural stocks of Sinonovacula constricta in Changjie, Ningbo, Haiyang Kexue (Marine Sciences), 28(10): 48-51 (李成华, 李太武, 苏秀榕, 宋林生, 2004b, 宁波长街缢蛏遗传结构的RAPD分析, 海洋科学, 28(10): 48-51)
Lin Z.H., Dong Y.H., Li N., Lu R.M., Xiao G.Q. and Liu B.Z., 2008, The genetic structure and diversity analysis of different geographical populations of Meretrix meretrix using morphological parameters and AFLP markers. Haiyang Yu Huzhao (Oceanologia Et Limnologia Sinica), 39(3): 245-251 (林志华, 董迎辉, 李宁, 陆荣茂, 肖国强, 刘保忠, 2008, 基于形态参数和AFLP标记的文蛤不同地理群体遗传变异分析, 海洋与湖沼, 39(3): 245-251)
Liu X.Q., Bao Z.M., Hu J.J., Wang S., Zhan A.B., Liu H., Fang J.G., and Wang R.C., 2007, AFLP analysis revealed differences in genetic diversity of four natural populations of Manila clam (Ruditapes philippinarum) in China, Acta Oceanologica Sinica, 26(1): 150-158
Liu Z.S., and Liu Z., 2009, China Fisheries Year book, China Agriculture Press, Beijing, China, pp.229-235 (刘增胜, 柳正, 2009, 中国渔业年鉴, 中国农业出版社, 中国, 北京, pp.229-235)
Nei M., 1973, Analysis of gene diversity in subdivided populations, Proceedings of the National Academy of Sciences of the USA, 70(12): 3321-3323 doi:10.1073/pnas.70.12.3321
Nei M., 1978, The theory of genetic distance and evolution of human races, Journal of Human Genetics, 23(4): 341-369
Niu D.H., Chen H., Wang S.L., Lin G.W., and Li J.L., 2010, Population genetic structure of Sinonovacula constricta along the coast of China. Dongwuxue Zazhi (Chinese Journal of Zoology), 45(2): 11-18 (牛东红, 陈慧, 王树亮, 林国文, 李家乐, 2010, 我国沿海缢蛏群体遗传结构的mtDNA-COI分析, 动物学杂志, 45(2): 11-18)
Niu D.H., Li J.L., Feng B.B., and Liu D.B, 2009, ISSR analysis on genetic structure of six Sinonovacula constricta populations, Yingyong Yu Huanjing Shengwu Xuebao (Chinese Journal of Applied & Environmental Biology), 15(3): 332-336 (牛东红, 李家乐, 冯冰冰, 刘达博, 2009, 缢蛏6个群体遗传结构的ISSR分析, 应用与环境生物学报, 15(3): 332-336)
Niu D.H., Li J.L., Shen H.D., and Jiang Z.Y., 2008, Sequence variability of mitochondrial DNA-COI gene fragment and population genetic structure of six Sinonovacula constricta populations, Haiyang Xuebao (Acta Oceanolo Sinica), 30(3): 109-116 (牛东红, 李家乐, 沈和定, 姜志勇, 2008, 缢蛏六群体线粒体DNA-COI基因序列变异, 海洋学报, 30(3): 109-116)
Niu D.H., Li J.L., Wang G.L., Jiang Z.Y., Zhang W.B., Shen Y.B., and Feng B.B., 2007, The genetic diversity of mitochondrial 16S rRNA gene fragment in six populations of Sinonovacula constricta, Shanghai Shuichan Daxue Xuebao (Journal of Shanghai Fisheries University), 16(1): 1-6 (牛东红, 李家乐, 汪桂玲, 姜志勇, 张文博, 沈玉帮, 冯冰冰, 2007, 缢蛏六群体16S rRNA基因片段序列的差异分析, 上海水产大学学报, 16(1): 1-6)
Pan J., Bao Z.M., Zhao Y., Hu X.L., Wan J.F., and Li Z., 2002, The genetic diversity of different geographical Zhikong Scallop (Chlamys farreri) populations, Gaojishu Tongxun (Chinese High Technology Letters), 12: 78-82 (潘洁, 包振民, 赵洋, 胡晓丽, 万俊芬, 李桢, 2002, 栉孔扇贝不同地理群体的遗传多样性分析, 高技术通讯, 12: 78-82)
Shao Y.Q., Lu R.M., Dong Y.H., Chai X.L., Xiao G.Q., and Lin Z.H., 2009, The genetic structure and diversity analysis of three species of razor clam using AFLP markers, Haiyang Kexue (Marine Sciences), 33(10): 26-30 (邵艳卿, 陆荣茂, 董迎辉, 柴雪良, 肖国强, 林志华, 2009, 三种蛏的遗传多样性分析, 海洋科学, 33(10): 26-30)
Vos P., Hogers R., Bleeker M., Reijans M., van de Lee T., Hornes M., Frijters J.P., Peleman J., and Kuiper M., 1995, AFLP: A new technique for DNA fingerprinting, Nucleic Acids Research, 23(21): 4407-4414 doi:10.1093/nar/23.21.4407 PMid:7501463 PMCid:307397
Wang D.Q., Li T.W., and Su X.R., 2005, Comparison of genetic diversity between hatchery stock and wild population of Sinonovacula constricta Lamarck in Xiangshan Bay, Zhongguo Shuichan Kexue (Journal of Fishery Science of China) 12(2): 138-143 (王冬群, 李太武, 苏秀榕, 2005, 象山缢蛏养殖群体和野生群体遗传多样性的比较, 中国水产科学, 12(2): 138-143)
Wright S., 1931, Evolution in Mendelian populations, Genetics, 16(2): 97-159 PMid:17246615 PMCid:1201091
Xiang J.H., ed, 1999, Studies on cell and population genetics of marine life, Shandong Science and Technology Press, Jinan, China, pp.112-123 (相建海, 主编, 1999, 海洋生物细胞和种群水平遗传研究, 山东科技出版社, 中国, 济南, pp.112-123)
Yu Y., Meng X.Y., Wang X.L., and Qiu X.M., 2007, Genetic diversity analysis of Razor Clam (Sinonovacula constricta) by RAPD, Shengwu Jishu Tongbao (Biotechnology Bulletin), (6): 138-140 (于颖, 孟祥盈, 王秀利, 仇雪梅, 2007, 缢蛏遗传多样性的RAPD分析, 生物技术通报, (6):138-140)
Zhang L.S., Kong X.Y., Yu Z.N., and Kong J., 2003, Application and prospects of AFLP in genetics studies of aquatic animals, Gaojishu Tongxun (Chinese High Technology Letters,) 13(4): 95-98, 104 (张留所, 孔晓瑜, 喻子牛, 孔杰, 2003, AFLP技术在水生动物遗传学研究中的应用及前景展望, 高技术通讯, 13(4): 95-98, 104)
Zhang W., Liu X., and Zhang G.F., 2005, AFLP analysis on genetic structure and differentiation of four different cultured populations of Argopecten irradians, Gaojishu Tongxun (Chinese High Technology Letters), 15(4): 84-88(张雯, 刘晓, 张国范, 2005, 利用AFLP技术研究海湾扇贝不同养殖群体的遗传结构及其分化, 高技术通讯, 15(4): 84-88)