 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 29 篇 doi: 10.5376/gab.cn.2011.30.0029
收稿日期: 2011年05月27日 接受日期: 2011年06月29日 发表日期: 2011年07月14日
引用格式(中文):
王艳等, 2011,花烛叶片总RNA提取方法的优化,基因组学与应用生物学(online), Vol.30 No.29 pp.1189-1193 (doi: 10.5376/gab.cn.2011.30.0029)
引用格式(英文):
Wang et al., 2011, Optimized of the methods on total RNA extraction from the leaf of Anthurium andraeanum, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.29 pp.1189-1193 (doi: 10.5376/gab.cn.2011.30.0029)
花烛叶片表面蜡质层较厚,组织中多糖、多酚等次生代谢物质含量较高,使用单纯的Trizol法难以提取高质量的总RNA,本试验比较了Trizol法、改良Trizol法和改良CTAB法提取花烛叶片总RNA的效果。结果表明,采用改良CTAB法所提取的花烛叶片总RNA中,28S rRNA亮度约为18S rRNA的两倍,OD260/OD280比值均为1.87~1.88,OD260/OD230比值为1.93~1.95,RNA产率为325.0~464.6 μg/mL,并以提取的RNA反转录得到cDNA,然后扩增GAPDH基因片段以验证RNA的质量,结果显示利用改良CTAB法从花烛叶片中提取的RNA质量好、产率高、完整性好,适用于RT-PCR等后续试验,能满足进一步分子生物学实验的要求。
花烛(Anthurium andraeanum Lind.),又名红掌、安祖花,为天南星科(Araceae)花烛属(Anthurium)多年生草本植物,原产南美洲热带雨林(袁秀波, 2005, 中国花卉园艺, (4): 16-19),因其苞片色泽鲜艳,造型奇特,瓶插期长,深受大众喜爱,已成为世界范围内广泛种植的切花和盆花之一(张正伟等, 2007,北京农业, (30): 100-103)。目前国内市场上的优良花烛品种均引自国外,培育具有自主知识产权的优良花烛新品种是提高我国花烛产业水平的关键之一,杂交育种、诱变育种等传统的花烛育种方法耗时较长,而通过基因工程,如转基因等途径可以克服固有生物种间限制,培育优良新品种是花烛育种的有效途径之一。
提取完整性好、纯度高的高质量花烛叶片总RNA,是开展花烛分子生物学方面研究及分子育种的前提和基础。而关于花烛RNA提取未见系统报道,本试验室前期采用Trizol法进行多次RNA提取效果均不理想,进一步比较了Trizol法、改良Trizol法和改良CTAB法的提取效果,针对花烛叶片富含多糖、多酚的特点,在RNA提取过程中加入去多糖步骤,提高了β-巯基乙醇的浓度,对CTAB法进行了改良(Clark et al., 1998; 李宏和王新力, 1999; 杜中军等, 2005),改良CTAB法成功地从花烛叶片中提取出了高质量的RNA,且所提取的RNA能够满足进一步分子生物学实验的需要。
1结果与分析
1.1花烛叶片总RNA凝胶电泳检测
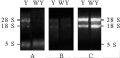
琼脂糖凝胶电泳是检验RNA质量的一种主要方法,从电泳凝胶上不仅可以判断RNA的完整性和降解程度,也可以判断有无DNA、蛋白质和多糖的污染。3种提取方法所提取的RNA采用1.2%的琼脂糖凝胶电泳,结果如图1所示。
|
|
由图1结果可以看到:Trizol法能提取出花烛黄化叶片总RNA,28S rRNA亮度约为18S rRNA的二倍,但野生型未能提出;采用改良Trizol法,野生型和黄化型叶片均能提出,但条带亮度较低且弥散,说明提取质量低且降解;采用改良CTAB法提取野生型和黄化型叶片总RNA均能提出且RNA普带清晰,亮度较高,且28S rRNA亮度约为18S rRNA的二倍,无5S小分子量RNA,说明RNA产率高、完整性好。
1.2提取的总RNA产量和纯度
采用3种方法所提取的野生型和黄化型花烛叶片总RNA产量因方法不同而有所不同(表1)。
|
|
表1所列结果为紫外分光光度计测定的各样品在波长230 nm、260 nm和280 nm的OD值,并据此计算出了RNA的浓度。OD230、OD260和OD280分别代表盐类、核酸及蛋白等有机物的含量。纯度较高的RNA OD260/OD280为1.7~2.0,OD260/OD230大于2.0。由表1可以看出,改良CTA法提取的野生型和黄化型花烛叶片RNA的OD260/OD280值分别 为1.88和1.88,OD260/OD230值分别为1.93和1.95,说明改良CTAB法提取的RNA样品纯度较高,没有蛋白质及多糖等杂质;Trizol法提取的黄化型花烛叶片RNA OD260/OD280、OD260/OD230值为1.77和1.41,说明杂质污染较严重;改良Trizol法提取黄化型和野生型花烛叶片RNA的OD260分别为0.061、0.026,说明核酸含量极低。
1.3 RT-PCR检测
为进一步确定改良CTAB法所提取RNA的质量和完整性,验证该RNA是否适宜RT-PCR、差异显示等分子生物学试验,我们以改良CTAB法提取的野生型和黄化型花烛叶片总RNA为模板,采用花烛3-磷酸甘油醛脱氢酶 (GAPDH)基因的特异性引物进行RT-PCR。结果表明,以改良CTAB法提取的野生型和黄化型花烛叶片总RNA为模板,均能获得接近400 bp的片段,且亮度较高;而以Trizol法提取的黄化型花烛叶片总RNA为模板,扩增产物条带亮度较低(图2)。改良Trizol法提取的野生型和黄化型花烛叶片总RNA浓度过低不能用来进行RT-PCR。试验结果表明,采用改良CTAB法提取的RNA完整性好,适用于基因扩增等试验。
|
|
2讨论
由于花烛叶片中酚类和多糖等次生代谢物含量较高,影响RNA的有效分离,前期试验采用RNA提取试剂盒所提取的RNA质量均较低,不能用于进一步的分子生物学研究。多糖的理化性质与RNA相似,因此在去除多糖的同时RNA容易被随之去除,导致RNA产量降低,而在沉淀RNA时,多糖亦随之沉淀,这样二者较难分离,进而造成一定程度的RNA降解和丢失(Wang and Vodkin, 1994)。此外,含有多糖的RNA沉淀难溶于水或溶解后的溶液较粘稠,而且会抑制许多酶的活性(Fang et al., 1992)。
本试验中,采用Trizol法能提取到花烛黄化型叶片的总RNA,但浓度较低,野生型则未能提取获得,而根据已测定的蛋白质和碳水化合物含量结果可知,野生型花烛叶片中糖分含量较黄化型叶片高(陈星旭, 2009),说明Trizol法不适于多糖多酚含量高的植物;改良Trizol法虽能提取到黄化型和野生型花烛叶片的总RNA,但浓度都均较低,且降解严重,不能用于RT-PCR及相关分子生物学试验;改良CTAB法以CTAB、高浓度β-巯基乙醇和NaCl作为裂解液的主要成分,较高浓度的β-巯基乙醇,可以有效防止酚的氧化,打断多酚氧化酶的二硫键而使之失活(Chang et al., 1993; Hu et al., 2002),采用24/1的氯仿/异戊醇混合液抽提2次去除了蛋白质污染,经过高浓度LiCl低温专一性沉淀,可将大部分多糖类物质保留在上清液中,用70%乙醇洗涤沉淀,可彻底去除多糖和蛋白质,在花烛绿色野生型及黄化型叶片中均能获得完整性好、纯度高的RNA。与Trizol法相比,本试验中所采用的改良CTAB法成本低,操作简单,所提取的RNA纯度高、完整性高,适用于RT-PCR及相关分子生物学试验。该方法可以为花烛分子生物学研究提供技术基础,同时也为其他富含多糖多酚成分的植物组织RNA提取提供了借鉴。
3材料与方法
3.1试验材料
以花烛品种‘Sonate’正常野生型和黄化突变体无菌苗叶片为材料进行RNA提取试验。
3.2主要试剂
Trizol试剂(Invitrogen公司),RT-PCR试剂(M-MLV、Buffer、dNTP、RNase Inhibitor、Oligo(dT)18、DEPC H2O、Tag酶)(Takara公司),CTAB(十六烷基三乙基溴化铵)、PVP(聚乙烯吡咯烷酮)、乙醇、氯仿、异丙醇、异戊醇、EDTA-Na2和LiCl等生化试剂均为国产分析纯。
2×CTAB提取缓冲液:100 mmol/L Tris-HCl (pH8.0),20 mmol/L EDTA-Na2,1.4 mol/L NaCl,2% CTAB (W/V), 1% PVP (W/W)。以上试剂均采用RNase Free水配置,122℃灭菌20 min;氯仿:异戊醇=24:1 (V/V);8 mol/L LiCl采用RNase Free水配置,并于122℃灭菌20 min。
3.3 Trizol试剂法
选取继代培养15 d后新形成的幼嫩叶片进行总RNA提取,称取约100 mg花烛幼嫩叶片,在液氮环境下研磨成灰白色粉末后,加入1 mL Trizol裂解液,室温放置5 min,待融化后迅速转至1.5 mL离心管中,4℃,12 000 rpm,离心10 min。取上清(约500 μL),转入另一干净的1.5 mL离心管中,加入200 μL氯仿,涡旋混匀,室温放置2 min。4℃,12 000 rpm,离心10 min。取上清(约600 μL),转入1.5 mL离心管中,加入等体积氯仿,涡旋混匀,4℃,12 000 rpm,离心10 min。取上清(约400 μL),转入1.5 mL离心管中,加入等体积冰的异丙醇,涡旋混匀,室温放置10 min。4℃,12 000 rpm,离心10 min。弃上清,加入1 mL 75%乙醇,震荡片刻。4℃,7 500 rpm,离心5 min。弃上清,4℃,7 500 rpm,高速离心1 min,用移液器移走残余溶液,室温倒置10 min,加入30 μL RNAse-free水溶解RNA,-70℃保存。
3.4改良Trizol试剂法
取1.5 mL离心管,加入1 mL Trizol裂解液、30 μL β-巯基乙醇、30 mg PVP,混匀,65℃水浴15分钟。称取100mg花烛幼嫩叶片,在液氮条件下研成灰白色粉末后,迅速转入Trizol裂解液的离心管中,涡旋混匀,65℃水浴15 min,4℃,12 000 rpm,离心10 min。其余步骤同Trizol试剂法。
3.5改良CTAB法
取2 mL离心管,加CTAB裂解液900 μL,β-巯基乙醇30 μL,65℃水浴10 min,称取100 mg幼嫩花烛叶片于液氮中研磨。将预热的CTAB裂解液加入到研钵中裂解,融化后迅速转入2 mL离心管,涡旋混匀30 s,65℃水浴5 min。加入1 mL氯仿/异戊醇,涡旋混匀,4℃,10 000 rpm,离心15 min。取上清至新的2 mL离心管,加入1 mL氯仿/异戊醇,涡旋混匀,4℃,10 000 rpm,离心15 min。取上清至1.5 mL离心管,加入1/3体积的8 mol/L LiCl,4℃沉淀过夜(不超过16 h)。4℃,12 000 rpm离心20 min,弃上清,加入500 μL70%乙醇悬浮沉淀,4℃,12 000 rpm离心5 min,加入500 μL 100%乙醇,弃上清,室温倒置1 min,加入30 μL RNAse-free水溶解RNA,-70℃保存。
3.6总RNA样品电泳分析
取2 μL RNA样品于1×TAE,1.2%琼脂糖凝胶上电泳,GelRed荧光染料(Biotium公司)染色,用凝胶成像系统(Tanon GIS2500)观察RNA条带,并照相记录,检测RNA的完整性。
3.7 RNA样品紫外分光光度计检测
采用核酸蛋白分析仪(Eppendorf Biophotometre 4G 22331 Hamburg)测定OD230、OD260、OD280以及RNA的浓度,用OD260/OD280,OD260/OD230检测RNA的纯度。
3.8 RT-PCR检测
取所提花烛叶片总RNA,按照Takara公司的M-MLV反转录试剂盒说明书进行反转录(3 μL RNA+1 μL oligo dT (18)+0.5 μL dNTP+2 μL 5×Buffer+0.25 μL RNase抑制剂+2 μL RNase free水+0.25 μL RTase M-MLV),得到10 μL花烛叶片cDNA。取1 μL花烛叶片cDNA进行PCR鉴定(20 μL反应体系):PCR扩增基因为花烛GAPDH基因片段(上游引物5’-ATCTGCCAAGGACTGGACA-3’,下游引物5’-ACGCGACGAGCTTAACAA-3’);cDNA模板经94℃变性5 min,按94℃ 30 s,58℃ 30 s,72℃ 30 s程序扩增32个循环,最后72℃继续延伸10 min。反应结束后,取10 μL PCR扩增产物于1%琼脂糖凝胶中进行电泳检测。
作者贡献
通讯作者王广东负责实验设计及安排,第一作者王艳负责实验具体实施和论文撰写,孙国胜负责溶液配制和RNA提取操作,王园园负责实验材料准备。
致谢
本研究得到国家自然科学基金课题(30871725)资助,特此感谢。同时感谢两位匿名的同行评审人的评审意见和修改建议。
参考文献
Chang S., Puryear J., and Caimey J., 1993, A simple and efficient method for isolating RNA from pine trees, Plant Molecular Biology Reporter, 11(2): 113-116 doi:10.1007/BF02670468
Chen X.X., 2009, Formative mechanism and maintenance characteristics of leaf color chimera and mutants of Anthurium andreaeanum ‘Sonate’, Thesis for M.S., Nanjing Agricultural University, Supervisor, Wang G.D., pp.28-29 (陈星旭, 2009, 花烛叶色嵌合体及突变体叶色形成机理及保持特征, 硕士学位论文, 南京农业大学, 导师: 王广东, pp.28-29)
Clark M.S., Gu H.Y., Qu L. J, Zheng H.H., Hu P., Zhou B.Y., Li D.H., Lin J.A., Wu X.M., and Sun S.Q., 1998, trans., Plant molecular biology, A laboratory manual, Higher Education Press, Beijing, China, pp.4-7
Du Z.J., Xu B.Q., Huang J.S., Wang J.B., and Xu L., 2005, An improved method for extracting intact total RNA from mango tissues rich in polysaccharides, Zhiwu Shenglixue Tongxun (Plant Physiology Communications), 41(2): 202-204 (杜中军, 徐兵强, 黄俊生, 王家保, 徐立, 2005, 一种改进的富含多糖的芒果组织中完整总RNA提取方法, 植物生理学通讯, 41(2): 202-204)
Fang G., Hammar S., and Grumet R., 1992, A quick and inexpensive method for removing polysaccharides from plant genomic DNA, Biotechniques, 13(1): 52-54, 56 PMid:1503775
Hu C.G., Honda C., Kita M., Zhang Z.L., Tsuda T., and Moriguchi T., 2002, A simple protocol for RNA isolation from fruit trees containing high levels of polysaccharides and polyphenol compounds, Plant Molecular Biology Reporter, 20(1): 69a-69g doi:10.1007/BF02801935
Li H., and Wang X.L., 1999, The difficulties in the isolation of RNA from plant tissues and their resolving strategies, Shengwu Jishu Tongbao (Biotechnology Bulletin), 15(1): 36-39 (李宏, 王新力, 1999, 植物组织RNA提取的难点及对策, 生物技术通报, 15(1): 36-39)
Wang C.S, and Vodkin L.O., 1994, Extraction of RNA from tissues containing high levels of procyanidins that bind RNA, Plant Molecular Biology Reporter, 12(2): 132-145 doi:10.1007/BF02668374
.png)
