2. 中科院上海有机化学研究所生命有机国家重点实验室, 上海, 200032
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 42 篇 doi: 10.5376/gab.cn.2011.30.0042
收稿日期: 2011年06月29日 接受日期: 2011年07月22日 发表日期: 2011年07月28日
引用格式(中文):
辛泽娜等, 2011,CoREST蛋白介导的组蛋白修饰与神经元发育机制,基因组学与应用生物学(online), Vol.30 No.42 pp.1272-1278 (doi: 10.5376/gab.cn.2011.30.0042)
引用格式(英文):
Xin et al., 2011, Histone modifications and mechanism of neuronal development mediated by CoREST protein, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.42 pp.1272-1278 (doi: 10.5376/gab.cn.2011.30.0042)
CoREST蛋白是在与REST蛋白C端相互作用时被发现。研究表明,CoREST蛋白参与了LSD1复合物和HDACs复合物介导组蛋白去甲基化与去乙酰化过程;作为REST的共抑制因子在REST蛋白发挥其特异性神经元基因抑制过程发挥辅助抑制因子作用;通过与其它DNA结合蛋白相互作用介导基因的转录过程。但是,CoREST蛋白结构和其它功能未知,为此,本文综述了近几年CoREST蛋白生物功能及相关可能的作用机制研究,为后期CoREST蛋白相关结构与功能研究奠定基础。
神经元限制性沉默因子(neuron-restrictive silencer factor, NRSF)抑制元素-1-沉默转录因子(repressor element-1-silencing transcription factor, REST)对基因表达的转录起着非常重要的负调控作用。神经元限制性沉默因子(NRSF/REST)是一种锌指蛋白转录因子,它能结合一个被称为神经限制性沉默元件(neuron-restrictive silencer element or repressor element-1, NRSE/RE1)的保守DNA片段上,从而在转录水平抑制许多和神经元发育及功能相关的基因在非神经元细胞中的表达。
1999年,人们在研究RE1沉默转录因子时发现了与NRSF/REST蛋白C-端相互作用的CoREST (corepressor of REST)蛋白,可以作为NRSF/REST蛋白发挥功能的辅助因子,在NRSF/REST蛋白与NRSE/RE1 dsDNA特异性结合时以抑制神经元基因表达的过程中发挥共抑制的作用(Andrés et al., 1999)。
对神经元特异性抑制基因所必需的多蛋白复合物BHC (BRAF-histone deacetylase complex)进行研究,结果显示神经元特异性抑制基因REST招募BHC多蛋白中的组份,从而调节REST蛋白对其特异性基因发挥抑制作用。BHC多蛋白复合物主要由具有两种酶活性的蛋白组成,即组蛋白去乙酰基化酶(HDAC1/2)和组蛋白去甲基化酶(BHC110, 又被称为LSD1或AOF2) (Hakimi et al., 2002; Shi et al., 2004; Hakimi et al., 2003)。BHC复合物包含6种蛋白:组蛋白去乙酰基酶HDAC1/2蛋白、包含SANT-结构域的CoREST蛋白、含PHD (plant homeo domain)结构域的BHC80蛋白、含HMG结构域的BRAF35蛋白和组蛋白去甲基化酶BHC110蛋白(即LSD1蛋白),BHC复合物中其它组份在CoREST蛋白介导的REST蛋白特异性抑制神经元基因转录过程中,不仅加强了组蛋白去甲基化的作用,同样促进了核小体基质的去乙酰基化作用,最终达到REST蛋白对神经元基因的转录抑制目的。为探索CoREST蛋白与REST蛋白相互作用提供了研究方向和基本依据。
CoREST蛋白主要以基因转录抑制REST蛋白的共抑制蛋白发挥作用,基因转录抑制作用常通过对组蛋白去甲基化过程或者组蛋白去乙酰基化过程来调节染色质结构变化而发生。研究表明,CoREST蛋白参与了LSD1复合物和HDACs复合物介导组蛋白去甲基化与去乙酰化过程;作为REST的共抑制因子在REST蛋白发挥其特异性神经元基因抑制过程发挥辅助抑制因子作用;通过与其它DNA结合蛋白相互作用介导基因的转录过程。但是,CoREST蛋白结构和其它功能未知,为此,本文综述了近几年CoREST蛋白生物功能及相关可能的作用机制研究,为后期CoREST蛋白相关结构与功能研究奠定基础。
1 CoREST蛋白的结构和序列的特征
CoREST蛋白包含3个结构域,其中有两个高度保守的SANT结构域,因为其中包含SWI3、ADA3、NCoR和TFIIIB4个转录因子而被命名为SANT,具有核受体的结构特征,ELM位于SANT结构域之前(图1)。(Yang et al., 2006)。在序列数据库里对CoREST中SANT段序列对比结果得出(图2),SANT结构域在很多蛋白中都存在,且主要是参与基因的转录调控。
图1人源CoREST结构域 注: ELM: aa105-182; SANT1: aa183-293; SANT2: aa381-450 Figure1.Structure of hCoREST Note: ELM: aa105-182; SANT1: aa183-293; SANT2: aa381-450 |
图2人源CoREST序列 注: 橙色区域为SANT1序列,蓝色区域为SANT2序列 Figure 2 hCoREST sequence Note: The range of the orange is SANT1 sequence and the range of the bule is SANT2 sequence |
2 CoREST在组蛋白去甲基化过程中的作用
核小体是构成染色质的基本单位。它由核心组蛋白八聚体和绕其约2周的146 bp双链DNA组成。组蛋白是一类碱性蛋白质,含精氨酸和赖氨酸等碱性氨基酸特别多,两者加起来约为其所有氨基酸残基的1/4,其有4个种类H2A,H2B,H3,H4。在染色体中核心组蛋白八聚体中央是由组蛋白H3和H4形成的异四聚体侧翼是组蛋白H2A 和H2B形成的异二聚体。每个.的氨基端约20~35个富含基本氨基酸的片段延伸到核小体的表面,这些突出于核小体的部分,称为“组蛋白尾(histone tail)。组蛋白尾在空间结构上相对可变,并与核内各种蛋白质或酶直接接触而被修饰。组蛋白尾的共价修饰,包括乙酰化、磷酸化、甲基化、泛素化等影响染色质结构的重构和DNA与蛋白质的相互作用,对染色质状态的形成和维持、基因转录表达调控、DNA损伤反应和修复、细胞记忆、以及多余DNA 的清除等多种细胞生命活动具有至关重要的作用。
突出于核小体颗粒外部的组蛋白N端尾部的转录后修饰,常发生在赖氨酸和精氨酸残基上,会影响染色质的结构和基因转录。而甲基化就代表了一种至关重要的组蛋白转录后修饰(Shi et al., 2004),甲基化修饰在很长一段时间里被认为是不可逆的,最近赖氨酸特异性去甲基化酶(lysine specific demethylation, LSD1)的发现对这一结论提出了新的观点。LSD1的发现,揭示了组蛋白的甲基化状态可以通过组蛋白甲基化酶和组蛋白去甲基化酶而进行动态调节(Shi et al., 2004)。LSD1是一种氨基氧化剂,通过依赖FAD (flavin adenosine dinucleotide)的氧化反应介导组蛋白去甲基化过程(Ballas et al., 2001) (图3)。继而发现,LSD1是通过对组蛋白H3K4的去甲基化作用来实现其作为转录共抑制蛋白的作用(Liang et al., 2004)。而H3K4上的甲基化与转录激活密切相关(Nakamura et al., 2002)。同时,在组蛋白H3K4特异性甲基化酶的超复合物中也发现了LSD1的存在(Yang et al., 2006),说明LSD1去甲基化活性在体外可以被调节。在对LSD1体外被调节的时间和方式的研究中发现了LSD1去甲基化的机制。
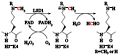 图3 FAD参与的LSD1去甲基化过程(Hakimi et al., 2003) Figure 3 FAD participate in the process of Histone demethylation by LSD1 (Hakimi et al., 2003) |
2.1 CoREST赋予重组LSD1对核小体基质去甲基化的作用
在之前的研究中提到,生物体内的LSD1可以对组蛋白肽段H3K4和体外组蛋白自由态H3K4去单甲基化和去二甲基化,但是重组的LSD1却不能使核小体基质去甲基化。研究发现,LSD1通过复合物来实现去甲基化的生物功能,LSD1复合物中包含HDAC1/2、CtBP1 (transcriptional corepressor C-terminal binding protein 1)、CoREST、BHC80和BRAF35,LSD1复合物中的组分会介导LSD1对更为复杂的基质进行修饰(Boyer et al., 2004)。
在对重组LSD1去甲基化作用的研究中发现,当加入CoREST蛋白后,重组LSD1重新恢复了去甲基化作用,使LSD1可以对核小体基质发挥去甲基化作用。对CoREST蛋白分析,其中SANT结构域非常保守,在许多染色质和转录调节子中都有发现。SANT结构域被认为作用在组蛋白尾部(Yu et al., 2003)。SMRT蛋白中含有SANT同源结构域,首先发现其与低度乙酰化的组蛋白尾部相互作用(Yu et al., 2003)。因此对CoREST中SANT结构域的功能提出了假设:CoREST蛋白作为一个连接核小体基质和去甲基化酶LSD1的桥梁,从而使去甲基化酶LSD1在核小体基质发挥去甲基化作用,CoREST在其中扮演桥梁蛋白的角色。
为了证实这样的一个假说,从事CoREST研究人员对CoREST蛋白的C端和N端分别进行了敲除和突变实验,如图4所示,以确定CoREST中分别与LSD1去甲基化酶和核小体之间相互作用的片段。研究结果表明,del5(aa293-381)与LSD1之间有物理的相互作用,但是不会使LSD1具有组蛋白去甲基化的活性;del2(aa293-482)与LSD1之间不仅仅有物理相互作用,而且还可以使LSD1发挥其组蛋白去甲基化作用。del5和del2之间所不同的是del2中包含了SANT2结构域,而del2中不包含SANT2结构域,这一发现支持了CoREST作为LSD1和核小体之间桥梁蛋白,而使LSD1发挥其去甲基化功能的说法。以此可以推测,SANT2可能有介导CoREST蛋白结合核小体基质的作用。在同样的条件下,CoREST蛋白N端结构域,即del1(aa1-293),其中包含了ELM结构域和SANT1结构域,并不与LSD1相互结合,而del1(aa1-293)在LSD1去甲基化过程中的具体作用尚不清楚,有人认为SANT1的作用也是介导蛋白-核小体之间相互作用,对于SANT1和ELM结构域,还需后续进一步的研究。
总之在LSD1的复合物中,CoREST主要是将LSD1连接到核小体基质上使其发挥组蛋白去甲基化的功能(Brown et al., 2005)。LSD1复合物去甲基化的机制,目前已有所研究。
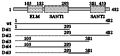 图4 鼠源CoREST蛋白敲除片段(Shi et al., 2005) Figure 4 Diagrams of mCoREST deletion mutant (Shi et al., 2005) |
2.2 在体内CoREST调节LSD1的稳定性
既然CoREST在LSD1的核小体基质去甲基化作用中有这么重要的作用,那么如果LSD1复合物中CoREST缺失的话会是什么情况呢?接下来就对CoREST的缺失对体内LSD1介导转录抑制过程的影响进行研究。研究结果显示,当CoREST蛋白不存在或者较大量缺失的情况下,大量的LSD1将被蛋白酶降解,表明了CoREST可以保护LSD1,使其不被蛋白酶降解而失去活性,维持了LSD1的稳定性;同样情况下,还会使LSD1靶基因发生去抑制而得以表达,同时组蛋白H3K4的二甲基化增加、LSD1在靶基因启动区数量锐减。在靶基因启动区LSD1数量的减少可能是由于LSD1本身降解所致。这暗示了一个可能存在却还未被认识的机制,可以通过调节CoREST蛋白表达量和CoREST/LSD1复合物之间的相互作用,最终影响到LSD1介导的组蛋白H3K4的去甲基化过程和转录抑制过程。
LSD1介导的组蛋白去甲基化过程,是受很多与LSD1相关的因素调节的。CoREST保护LSD1不被蛋白酶所降解,并且在LSD1介导的核小体基质去甲基化过程中扮演不可或缺的角色。这就说明COEST是LSD1正常发挥其功能的关键要素。
3 CoREST在组蛋白去乙酰基化过程中的作用
可逆的组蛋白乙酰基化,被认为是一种主要的真核基因转录调节因子,通过乙酰基转移酶(histone acetyltransferases, HATs)将组蛋白尾部的赖氨酸残基乙酰基化,可以活化基因转录(Kouzarides, 1999; Struhl et al., 1998; Urnov and Wolffe, 2001)。组蛋白的乙酰基化削弱了组蛋白N端尾部与DNA之间的相互作用,使染色质结构更少的受到限制,通常有利于基因的转录活性(Kuo et al., 1998)。组蛋白的乙酰基化状态依赖组蛋白乙酰基转移酶(HATs)和组蛋白去乙酰基酶(histone deacetylations, HDACs)的协调来维持(Strahl et al., 2000)。大部分的HDACs (Histone Deacetylases)属于锌金属酶的超大家族,在催化结构域有380个保守的残基(Finnin et al., 1999)。
组蛋白去乙酰基酶HDACs,与HATs的酶催化反应相反,能够使染色质结构到达抑制转录的状态(Pazin and Kadonaga, 1997; Leipe et al., 1997)。HDACs被发现在体内作为很大的一个多蛋白复合物,可以被DNA结合蛋白锚定从而活化基因转录抑制过程(Humphrey et al., 2001)。NRSF/REST蛋白作为DNA结合蛋白,C端招募CoREST/HDACs复合物,从而能正常发挥神经元特异性基因的转录抑制功能。
组蛋白去乙酰基酶复合物,又称为CoREST/HDACs复合物。通过研究复合物组分,分析了CoREST与HDACs的相关性,确定CoREST为HDACs复合物的成份。HDAC1 (482个残基)和HDAC2 (488个残基)为人类同源蛋白,同源性高达84%。HDAC1和HDAC2功能是否不同尚不清楚,但是他们在基因转录抑制过程中扮演相互补充一起发挥作用的角色。研究发现,CoREST-HDACs复合物显示出组蛋白去乙酰基化活性,而重组的HDACs却没有去乙酰基的活性。但CoREST具体活化机制尚不明确。
3.1 CoREST与组蛋白去乙酰基酶HDACs的相互作用
进一步研究表明,CoREST与HDAC1/2之间存在相互作用(You et al., 2001)。为了确定CoREST中与HDAC1/2相互作用的区域,对CoREST进行片段敲除和突变进行了实验,结果显示,CoREST中只SANT结构域与HDAC1相互作用,CoREST蛋白N端179个氨基酸包含的SANT1结构域的多肽,对CoREST与HDAC1之间的相互作用至关重要。与全长CoREST对比发现,这个区域被敲除后,将会导致HDAC1与CoREST之间失去相互作用,表明这段结构域对CoREST和HDACs之间的相互作用很重要;同时发现,删除该区域后,也会使HDAC失去组蛋白去乙酰基酶的活性,从而影响到基因的转录抑制功能。CoREST在HDAC去乙酰基化过程中,可能也是连接HDACs到核小体上,而使HDACs可以发挥其组蛋白去乙酰基化作用,为不可缺少的共抑制因子。
3.2 组蛋白去乙酰基化酶HDACs和组蛋白去甲基化酶LSD1之间的相互作用
在前述中,CoREST可以使LSD1和HDAC发挥组蛋白去甲基化和去乙酰基化的作用,CoREST/LSD1对高度乙酰化的核小体不识别,只对低度乙酰化的核小体有去甲基化作用。于此一致的发现是,HDAC1可以通过与CoREST的相互作用,使高度乙酰化的核小体去乙酰化,从而使核小体低度乙酰化。表明在LSD1复合物中,HDACs在CoREST/LSD1上游发挥作用,产生一种低度乙酰化的核小体基质,可以更好的被CoREST/LSD1所识别。将HDACs用TSA(HDAC抑制剂)处理发现,LSD1的靶基因发生去抑制而得以表达。这样的发现可以启发我们,HDACs在LSD/CoREST介导体内基因转录抑制过程中扮演重要角色,HDACs与LSD/CoREST相互合作一起抑制REST靶基因的表达(Shi et al., 2005)。
4 CoREST参与REST蛋白特异性抑制神经元基因的表达
NRSF/REST属于Gli-Kruppel样转录因子家族,NRSF/REST在非神经元细胞中表达比较高,而在神经元细胞中不表达,在一些特定的成熟神经元中REST也会有表达(Shimojo et al., 2004; Driffith et al., 2001),暗示REST可能在某些神经元中能够重新表达或者持续表达。REST在生物体中的作用非常复杂,如在海马神经细胞中就能检测到REST,随着缺血和癫痫损害,NRSF/REST表达量随之升高(Palm et al., 1998; Calderone et al., 2003),证实了REST在神经元中作为重要抑制子的功能,另外,NRSF/REST在与亨廷顿舞蹈症相关的神经退化当中也起着重要作用(Zuccato et al., 2003)。NRSF/REST还能介导胎儿心血管基因在心脏和血管平滑肌的程序表达(Kuwabara et al., 2004; Cheong et al., 2005),也能与经典的抑癌基因相互作用(Westbrook et al., 2005)。
NRSF/REST能与神经限制性沉默元件NRSE/RE1结合,在转录水平抑制许多与神经元发育及功能相关的基因在非神经细胞中的表达。RE1最早在电压门控Ⅱ型钠离子通道(NaChⅡ)和颈上神经基因10 (SCG10)的5'非编码区发现的一个基因转录沉默子,是长度为21~23 bp的DNA反应元件。其功能主要是作为沉默子介导了神经元特异性基因在神经干细胞和非神经细胞中的转录抑制作用,从而赋予神经元的细胞特异性。
NRSF/REST全长1 069个氨基酸,含9个Cys2/His2型锌指结构,具有一个DNA结合结构域、赖氨酸富含区、脯氨酸富含区和两个转录抑制结构域和N端抑制结构域以及C端抑制结构域(含一个C2H2型锌指结构域, 即ZnF9) (Leichter and Thiel, 1999; 居家莹等, 2009)。REST N-端结构域招募mSin3HDAC复合物,C-端结构域招募CoREST。N-端结构域招募的mSin3HDAC复合物与NRSF/REST靶基因即RE1短时抑制相关。研究表明将缺失N端或C端抑制结构域的NRSF/REST在多种细胞株中过量表达后,发现只有部分启动子区域携带RE1序列的基因表达得到了抑制(Grimes et al., 2000),暗示部分受NRSF/REST调节的基因可能需要两种抑制方式的同时存在。
4.1 CoREST与REST的相互作用
CoREST并不与NRSF/REST中八锌指结构域相互作用,当将REST C-端单锌指结构域ZnF9敲除或突变时,NRSF/REST将失去抑制活性,也不再与CoREST相互作用(Tapia-Ramirez et al., 1997)。NRSF/REST C端抑制结构域包含单锌指结构ZnF9,位于终止密码子上游40个氨基酸的位置。将这段结构域敲除或者突变对于维持锌指结构比较关键的残基时,会使NRSF/REST蛋白失去抑制活性,因为单锌指结构经常介导蛋白与蛋白相互作用,表明NRSF/REST中ZnF9单锌指在参与与CoREST共抑制因子相互作用中是关键结构域。
对CoREST蛋白逐段敲除的研究(图5)显示,CoREST中包含部分SANT1在内的结构域即CoREST (aa102~225)显示与REST相互作用(Ballas et al., 2001)。
 图5 鉴定hCoREST中功能片段 (Ballas et al., 2001) Figure 5 Idenrification of Function Domains in hCoREST (Ballas et al., 2001) |
4.2 CoREST与REST蛋白相互作用的初步模型
CoREST介导NRSF/REST蛋C端抑制结构域的抑制过程中,如前所述,CoREST在组蛋白去甲基化作用过程和组蛋白去乙酰基化过程中都有非常重要的作用。其HDACs和LSD1作为CoREST的复合物,参与介导NRSF/REST蛋白对神经特异性基因的抑制作用。RE1会将REST蛋白招募至其基因启动区,REST蛋白通过C端招募CoREST复合物和N端招募mSin3HDAC复合物,CoREST招募HDACs和LSD1来实现组蛋白的去乙酰基和去甲基从而使染色质结构到达抑制状态,进行神经元特异性基因的抑制(图6)。
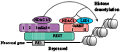 图6 RE-1依赖REST/CoREST复合物特异性抑制基因模型 Figure 6 Model of the RE-1 dependent REST/CoREST Complex |
可能存在的作用机制是,NRSF/REST通过与CoREST N-端含SANT1结构域的相互作用,使CoREST在NRSF/REST上固定,在CoREST N端包含SANT1的区域里,与HDAC1发生相互作用,SANT2结构域则招募LSD1,HDAC1使高度乙酰化的核小体去发生乙酰基作用,LSD1则对低度乙酰化的核小体发生去甲基化作用,使染色质到达一个抑制转录的状态,从而抑制了NRSF/REST靶基因的表达。
5 展望
CoREST作为转录抑制辅助因子,与CoREST复合物一起介导基因转录过程。CoREST功能研究能很好促进CoREST复合物以及这些复合物与DNA结合蛋白间的对转录过程调控机制的研究,为以后探究转录过程调控机制奠定良好的基础。CoREST在成人细胞中的广泛存在和表达,表明CoREST不仅仅只作为NRSF/REST的共抑制因子而发挥神经特异性基因抑制作用,也可以与其它DNA结合蛋白相互作用发挥类似的功能。但是CoREST蛋白结构和其它功能未知,在探求CoREST蛋白结构中存在多种困难,CoREST蛋白不易得到,且其确切的功能模式尚不明确。为此,本文综述了近几年CoREST蛋白生物功能及相关可能的作用机制研究,为后期CoREST蛋白相关结构与功能研究奠定基础。
作者贡献
辛泽娜调阅文献,撰写文章。张岩协助调阅文献,修改文章。童孝田修改文章,协助解决文献中的难点问题。杨桂秋为本文通讯作者。
致谢
感谢中科院上海有机所生命有机国家重点实验室曹春阳课题组成员在本文撰写中的指导与支持,同时感谢国家自然科学青年基金(30800176)与科技部“蛋白质重大研究计划”(2009CB918600)对本文工作的资助。感谢两位匿名的同行评审人的评审意见与修改建议。
参考文献
Andrés M.E., Burger C., Peral-Rubio M.J., Battaglioli E., Anderson M.E., Grimes J., Dallman J., Ballas N., and Mandel G., 1999, CoREST: A functional corepressor required for regulation of neural-specific gene expression, Proceedings of the National Academy of Science of the United States of America, 96(17): 9873-9878
http://dx.doi.org/10.1073/pnas.96.17.9873
Ballas N., Battaglioli E., Atouf F., Andres M.E., Chenoweth J., Anderson M.E., Burger C., Moniwa M., Davie J.R., Bowers W.J., Federoff H.J., Rose D.W., Rosenfeld M.G., Brehm P., and Mandel G., 2001, Regulation of neuronal traits by a novel transcriptional complex, Neuron, 31(3): 353-365
http://dx.doi.org/10.1016/S0896-6273(01)00371-3
Boyer L.A, Latek R.R., and Peterson C.L., 2004, The SANT domain: A unique histone-tail-binding module? Nat. Rev. Mol. Cell Biol., 5: 158-163
http://dx.doi.org/10.1038/nrm1314
PMid:15040448
Brown C.E., Lechner T., Howe L., and Workman J.L., 2005, The many HATs of transcription coactivators, Trends Biochem. Sci., 25(1): 15-19
http://dx.doi.org/10.1016/S0968-0004(99)01516-9
Calderone A., Jover T., Noh K., Tanaka H., Yokota H., Lin Y., Grooms S.Y., Regis R., Bennett M.V.L., and Zukin R.S., 2003, Ischemic insults derepress the gene silencer REST in neurons destined to die, The Journal of Neurosicence, 23(6): 2112-2121
PMid:12657670
Cheong A., Bingham AJ., Li J., Kumar B., Sukumar P., Munsch C., Buckley N.J., Neylon C.B., Porter K.E., Beech D.J., and Wood I.C., 2005, Downregulate REST transcription factor is a switch enabling critical potassium channel expression and cell proliferation, Molecular Cell, 20(1): 45-52
http://dx.doi.org/10.1016/j.molcel.2005.08.030
PMid:16209944
Driffith E.C., Cpwan C.W., and Greenberg M.E., 2001, REST acts through multiple deacetylase complexs, Neuron, 31(3): 339-340
http://dx.doi.org/10.1016/S0896-6273(01)00386-5
Finnin M.S., Donigian J.R., Cohen A, Richon V.M., Rifkind R.A., Marks P.A., Breslow R., and Pavletich N.P., 1999, Strcture of a histone deacetylase homologue bound to the TSA and SAHA inhibitors, Nature, 401(6749): 188-193
http://dx.doi.org/10.1038/43710
PMid:10490031
Grimes J.A., Nielsen S.J., Battaglioli E., Miskai E.A., Speh J.C., Berry D.L., Atouf F., Holdener B.C., Mandel G., and Kouzarides T., 2000, The co-repressor mSin3A is a functional component of the REST-CoREST repressor complex, J. Bio. Chem., 275(13): 9461-9467
Hakimi M.A., Bochar D.A., Chenoweth J., Lane W.S., Mandel G., and Shiekhattar R., 2002, A core-BRAF35 complex containing histone deacetylase mediates repression of neuronal-specific genes, Proc. Natl. Acad. Sci. USA, 99(11): 7420-7425
http://dx.doi.org/10.1073/pnas.112008599
PMid:12032298 PMCid:124246
Hakimi M.A., Dong Y.S., Lane W.S., Speicher D.W., and Shiekhattar R., 2003, A candidate X-linked mental retardation gene is a component of a new family of histone deacetylase-containing complexes, J. Biol. Chem., 278(9): 7234-7239
http://dx.doi.org/10.1074/jbc.M208992200
PMid:12493763
Humphrey G.W., Wang Y.H., Russanova V.R., Hirai T., Qin J., Nakatani Y., and Howard B.H., 2001, Stablge histone deacetylase complexes distinguished by the presence of SANT domain proteins CoREST/kiaa0071 and Mta-L1, J. Biol. Chem., 276(9): 6817-6824
http://dx.doi.org/10.1074/jbc.M007372200
PMid:11102443
Ju J.Y., Yang Z.Z., Wu H.M., Wang Z.F., and Cao C.Y., 2009, Solution structure determination of zinc finger 9 in NRSF/REST by NMR, Huaxue Xuebao (Acta Chimic. Sinica), 67(7): 705-708 (居家莹, 杨中正, 吴厚铭, 王仲孚, 曹春阳, 2009, NRSF/REST蛋白结构域锌指9核磁共振溶液结构确定, 化学学报, 67(7): 705-708)
Kouzarides T., 1999, Histone acetylase and deacetylases in cell proliferation, Curr. Opin. Genet. Dev., 9(1): 40-48
http://dx.doi.org/10.1016/S0959-437X(99)80006-9
Kuo M.H., and Allis C.D., 1998, Roles of histone acetyltransferases and deacetylases in gene regulation, Biocessays, 20(8): 615-626
http://dx.doi.org/10.1002/(SICI)1521-1878(199808)20:8<615::AID-BIES4>3.0.CO;2-H
Kuwabara T., Hsieh J., Nakashima K., Taira K., and Gage F.H., 2004, A small modulatory dsRNA specifies the fate of adult neural stem cells, Cell, 116(16): 779-793
http://dx.doi.org/10.1016/S0092-8674(04)00248-X
Leichter M., and Thiel G., 1999, Transcriptional repression by the zinc finger protein REST is mediated by titratable nuclear factors, Eur. J. Neurosci., 11(6): 1937-1946
http://dx.doi.org/10.1046/j.1460-9568.1999.00617.x
PMid:10336662
Leipe D.D., and Landsman D., 1997, Histone deacetylases, acetoin utilization proteins and acetylpolyamine amidohydrolases are members of an ancient protein superfamily, Nucleic Acids Research, 25(18): 3693-3697
http://dx.doi.org/10.1093/nar/25.18.3693
PMid:9278492 PMCid:146955
Liang G., Lin J.C., Wei V., Yoo C., Cheng J.C., Nguyen C.T., Weisenberger D.J., Egger G., Takai D., Gonzales F.A., and Jones P.A., 2004, Distinct localization of histone H3 acetylation and H3-K4 methylation to the transcription start sites in the human genome, Proceeding of the National Academy of Sciences of the United States of America, 101(19): 7357-7362
Nakamura T., Mori T., Tada S., Krajewski W., Rozovskaia T., Wassell R., Dubois G., Mazo A., Croce C.M., and Canaani E., 2002, ALL-1 is a histone methyltransferase that assembles a supercomplex of proteins involved in transcriptional regulation, Molecular Cell, 10(5): 1119-1128
http://dx.doi.org/10.1016/S1097-2765(02)00740-2
Palm K., Belluardo N., Metsis M., and Timmusk T., 1998, Neuronal expression of zinc finger transcription factor REST/TRB gene, J. Neurosci., 18(4): 1280-1296
PMid:9454838
Pazin M.J., and Kadonaga J.T., 1997, What's up and down with histone deacetylation and transcription? Cell, 89(3): 325-328
http://dx.doi.org/10.1016/S0092-8674(00)80211-1
Shi Y., Lan F., Matson C., Mulliqan P., Whetstine J.R., Cole P.A., Casero R.A., and Shi Y., 2004, Histone demethylation mediated by the nuclear amine oxidase homolog LSD1, Cell, 119(7): 941-953
http://dx.doi.org/10.1016/j.cell.2004.12.012
PMid:15620353
Shi Y.J., Matson C., Lan F., Iwase S., Baba T., and Shi Y., 2005, Regulation of LSD1 histone demethylase activity by its associated factors, Molecular Cell, 19(6): 857-864
http://dx.doi.org/10.1016/j.molcel.2005.08.027
PMid:16140033
Shimojo M., and Hersh L.B., 2004, Regulation of the cholinergic gene locus by the repressor element-1 silencing transcription factor/neuron restrictive silencer factor (REST/NRSF), Life sciences, 74(18): 2213-2225
http://dx.doi.org/10.1016/j.lfs.2003.08.045
PMid:15017977
Strahl B.D., and Allis C.D., 2000, The language of covalent histone modifications, Nature, 403: 41-45
http://dx.doi.org/10.1038/47412
PMid:10638745
Struhl K., 1998, Histone acetylation and transcriptional regulatory mechanisms, Genes and Development, 12(5): 599-606
http://dx.doi.org/10.1101/gad.12.5.599
Tapia-Ramirez J., Egen B.J.L., Peral-Rubio M.J., Toledo-Aral J.J., and Mandel G., 1997, A single zinc finger motif in the silencing factor REST represses the neural-specific type II sodium channel promoter, Proc. Natl. Acad. Sci. USA, 94(4): 1177-1182
http://dx.doi.org/10.1073/pnas.94.4.1177
Urnov F.D., and Wolffe A.P., 2001, A necessary good: Nuclear hormone receptors and their chromatin templates, Mol. Endocrinol., 15(1): 1-16
http://dx.doi.org/10.1210/me.15.1.1
PMid:11145735
Westbrook T.F., Martin E.S., Schlabach M.R., Leng Y., Liang A.C., Feng B., Zhao J.J., Roberts T.M., Mandel G., Hannon G.J., DePinho R.A., Chin L.D., and Elledge S.J., 2005, A genetic screen for candidate tumor supressors identifies REST, Cell, 121(6): 837-848
http://dx.doi.org/10.1016/j.cell.2005.03.033
PMid:15960972
Yang M.J., Gocke C.B., Luo X.L., Borek D., Tomchick D.R., Machius M., Otwinowski Z., and Yu H.T., 2006, Structural basis for CoREST-dependent demethylation of nucleosomes by the human LSD1 histone demethylase, Molecular Cell, 23(3): 377-387
http://dx.doi.org/10.1016/j.molcel.2006.07.012
PMid:16885027
You A., Tong J.K., Grozinger C.M., and Schreiber S.L., 2001, CoREST is an integral component of the CoREST human histone deacetylase complex, PNAS, 98(4): 1454-1458
http://dx.doi.org/10.1073/pnas.98.4.1454
PMid:11171972
Yu J., Li Y., Ishizuka T., Guenther M.G., and Lazar M.A., 2003, A SANT motif in the SMRT corepressor interprets the histone code and promotes histone deacetylation, Embo J., 22(13): 3403-3410
http://dx.doi.org/10.1093/emboj/cdg326
PMid:12840002 PMCid:165650
Zuccato C., Tartari M., Crotti A., Goffredo D., Valenza M., Conti L., Cataudella T., Leavitt B.R., Hayden M.R., Timmusk T., Rigamonti D., and Cattaneo E., 2003, Huntingtin interacts with REST to modulate the transcription of RE1-controlled neuronal genes, Nat. Genet, 35(1): 76-83
http://dx.doi.org/10.1038/ng1219
PMid:12881722