 作者
作者  通讯作者
通讯作者
豆科基因组学与遗传学, 2010 年, 第 1 卷, 第 1 篇 doi: 10.5376/lgg.cn.2010.01.0001
收稿日期: 2010年05月14日 接受日期: 2010年06月12日 发表日期: 2010年09月27日
Liu et al., 2010, Diversity Analysis of Faba Bean Germplasms in Qinghai Based on AFLP and
Construction of Their Core Gerplasms, Douke Jiyinzuxue Yu Yichuanxue (online) (Legume Genomics and Genetics) Vol.1 No.1 (doi: 10.5376/lgg.cn.2010.01.0001) (刘玉皎等, 2010, 青海蚕豆种质资源AFLP 多样性分析和核心资源构建, 豆科基因组学与遗传学(online), Vol.1 No.1 (doi: 10.5376/lgg.cn.2010.01.0001))
采用AFLP分子标记技术分析了青海蚕豆种质资源遗传多样性,结果表明:地方品种资源、国外种质和选育品种的有效等位变异分别为1.0 655±0.1 263、1.0 634±0.1 430和1.0 659±0.1 651;Shannon’s信息指数分别为0.0 954±0.1 449、0.0 843±0.1 518和0.0 742±0.1 679;地方品种资源有效等位变异比例最小(为73.73%),而多态性比例最高(为44.51%);选育品种的有效等位变异比例最大(为90.39%),多态性比例最低(为17.92%);地方品种间的遗传距离较大;国外种质间的遗传距离较小。遗传结构分析表明青海蚕豆种质资源可分为7个组群。本研究初步构建了40份核心种质资源,基本包含了全部种质80%以上的遗传信息,群体总基因多样性和Nei’s基因多样性指数明显提高,可作为杂交育种的优异亲本材料。
AFLP (Amplified Fragment Length Polymorphism)是1992年由荷兰科学家Zabeau和Vos发展起来的一种检测DNA多态性的新方法:对基因组总DNA酶切后经PCR进行选择性扩增,随后将特定的接头连接在这些DNA片段的两端。接头序列和邻近的限制性酶切位点序列作为引物结合位点,然后用特定引物进行PCR扩增,最后通过聚丙烯酰胺凝胶电泳分离扩增的特异限制片段(周自清, 2005)。它是在RFLP和RAPD的基础上发展起来的,是RFLP和RAPD的结合,既有RFLP的可靠性,也有RAPD的方便性。
青海属典型的高原大陆性气候,而且海拔高具有明显垂直分布特点。蚕豆是青海的传统农作物,种植历史悠久,分布区域比较广,种植面积28 230 hm2 (青海省统计年鉴定, 2009),在海拔1 500~3 000 m的地区均有蚕豆种植,在独特的生态环境中栽培和驯化后形成了当地丰富的蚕豆种质资源。遗传多样性是资源利用和作物品种改良的基础,评估具有普遍适应性和特异性的青海蚕豆主要种质资源的遗传多样性,可以预测纯系品种分离后代的变异度。国内外同行已采用多种标记方法研究了蚕豆种质资源的多样性,如采用形态标记分析蚕豆种质资源多样性(王晓娟等, 2009; 张小田和刘玉皎, 2009; 刘玉皎和宗绪晓, 2003);利用AFLP技术对79个蚕豆近交系的遗传多样性进行了分析,结果共产生了477个多态性片段(Zeid M et al, 2001)。应用RAPD技术以28个杂交品系为研究材料,研究欧洲小粒种和大粒种与地中海资源的遗传多样性(Link et al, 1995)。运用7个同工酶对来自5个不同地理来源和5个不同类型的26份蚕豆资源进行主成分分析,结果表明,埃索比亚地区(Ethiopia)和亚洲地区的蚕豆资源可以单独的分为一个组群(M. Serradilla et al., 1993);采用草酰乙酸转氨酶(GOD)、过氧化物歧化酶(SOD)和苹果酸酶(ME)对33份蚕豆资源进行研究,判别分析表明,33份蚕豆资源可以清楚的划分为5个组群(Polignano et ai.,1998)。基于形态标记构建了青海蚕豆种质资源的初步核心资源(杨菁等, 2009)。
本文采用AFLP标记技术分析青海蚕豆种质资源的遗传多样性,并以此构建青海蚕豆种质资源的核心资源,为更好地掌握青海蚕豆种质资源的遗传基础,提高种质的有效利用提供理论依据。
1 结果分析
1.1 AFLP引物的多样性
10对AFLP引物扩增的多态性带在3~10之间,其中E1/M6、E2/M2、E4/M4和E7/M7等4对引物组合扩增出的多态性条带最多,为10条,多态性比例分别为52.63%、52.63%、50.00%和52.63%,有效等位变异占等位变异的比例分别为67.80%、68.46%、73.35%和68.59%;E6/M6扩增出的多态性条带只有3条,其它引物组合扩增出的多态性条带数在为7~8条,见表1。
 表1 10对AFLP引物组合扩增的等位变异数、有效变异数与Shannon’s信息指数 Table 1 The AFLP primer combination amplified products and their polymorphism in Qinghai faba bean |
1.2青海蚕豆种质资源的AFLP多样性
10对AFLP引物在149份蚕豆资源中检测到平均有效等位变异为1.0 647,有效等位变异所占比例为72.52%,共扩增出多态性条带81条,多态性比例为46.82%,Shannon's信息指数为0.0 946,其中从选育品种、引进国外资源、地方品种中检测到平均有效等位变异所占比例依次为90.39%、81.76%和72.52%,地方品种的多态性较高,多态性比例和多样性指数分别为44.51%和0.0512,选育品种的多态性较低,多态性比例和多样性指数分别为17.92%和0.0455,见表2。
表2 10对AFLP引物组合扩增的不同资源群等位变异数、有效变异数与Shannon's信息指数 Table 2 The distribution of AFLP amplified products and their polymorphism in groups of Qinghai faba bean |
1.3 Structure遗传结构分组分析
利用Structure 2.2软件对蚕豆种质资源进行遗传结构分析,根据分析结果绘制K值拐点图(图1),K值为7时出现了第一个明显的拐点,因此,青海蚕豆种质资源最佳分组应为7个组群(图2)。
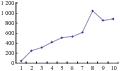 图1 K值与Var[lnP(D)]的关系图 Figure 1 The map of relation between K and Var[lnP(D)] |
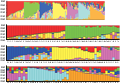 图2 青海蚕豆种质资源群体遗传结构图 Figure 2 Estimated population structure for faba bean gene resources in Qinghai |
149份种质资源的群体遗传分为7个组群,且不同来源的种质资源出现在不同的组群中,说明资源的遗传差异相对较大,且有一定的遗传相似性。第一组群中包含了26个群体,其中50%的选育品种、16.67%的引进资源和12.87%的地方品种,有效等位变异数所在比例最高为92.96%,信息指数为0.027 8,各群体间的平均遗传距离较小;第二组群包含24个群体,其中18.81%的地方品种和13.89%的引进资源,有效等位变异所占比例为89.58%,信息指数为0.065 9;第三组群包含20个群体,其中16.83%的地方品种、5.56%的引进资源和8.33%的选育品种,有效等位变异所占比例为84.47%,信息指数为0.124 0,各群体间的平均遗传距离最大,第四组群包含22个群体,其中14.85%的地方品种、16.67%的引进资源和8.33%的选育品种,有效等位变异所占比例为92.50%,信息指数为0.033 2,第五组群包含17个群体,其中8.91%的地方品种、16.67%的引进资源和16.67%的选育品种,有效等位变异所占比例为89.08%,信息指数为0.064 5;第六组群包含18个群体,其中8.91%的地方品种、19.44%的引进资源和16.67%的选育品种,有效等位变异所占比例为91.39%,信息指数为0.077 2;第七组群包含23个群体,其中17.82%的地方品种和11.11%的引进资源,有效等位变异所占比例为92.74%,信息指数为0.059 8,见表3。
表3 不同组群的遗传信息特征 Table 3 The Gene information of Groups
|
1.4 青海蚕豆种质资源的遗传相似性分析
149份青海蚕豆种质资源中,地方品种、国外种质以及选育品种中各群体之间的遗传相似系数分别为0.948 2 (0.867 1~1.000 0)、0.951 3 (0.884 4~1.000 0)、0.950 3 (0.907 5~0.994 2);遗传距离分别为0.053 5 (0~0.142 7)、0.050 3 (0~0.122 9)、0.051 1 (0.005 8~0.097 0)(图3)。地方品种间遗传距离较大,国外资源的遗传距离较小,这与种群数量有关。
 图3 不同来源青海蚕豆种质资源的遗传相似系数和遗传距离 Figure 3 The gene coefficience and distance of faba bean gerplasm in Qinghai |
1.5 基于AFLP的核心资源构建
以来源为单元,Popgen V1.32结合MEGA 3.0分析蚕豆种质资源的亲缘关系进化树,以进化树为基础进行亲缘关系较近资源的按比例取舍,结果见表4、图4。从149份种质资源中,初步构建出40份核心资源,其等位变异数、有效等位变异数、多态性条带数、遗传相似系数、遗传距离以及基因多样性指数等遗传信息的符合率均在80%以上,经显著性测验后,差异不显著,表明初步构建的青海蚕豆种质资源的核心种质能代表全部种质的遗传信息,群体的总基因多样性和Nei’s基因多样性指数分别为0.058 3、0.103 2,分别提高了89.3%和104.0%,核心种质资源包含青海地方品种20份,国外种质11份,选育品种9份。
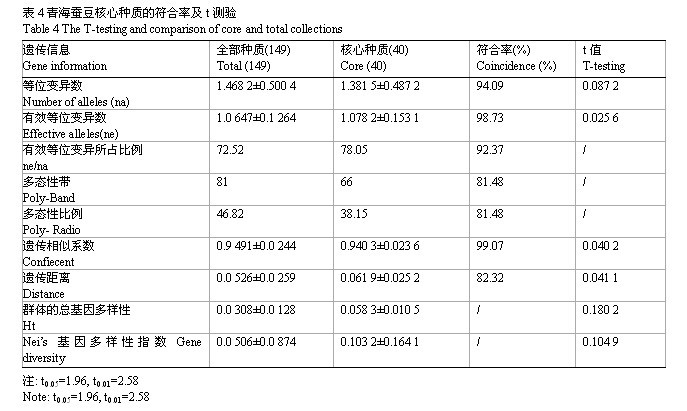 表4 青海蚕豆核心种质的符合率及t测验 Table 4 The T-testing and comparison of core and total collections |
.png) 图4 基于AFLP的核心资源聚类图 Figure 4 The cluster mapping of core resources based AFLP |
2 讨论
2.1 多样性分析与种质分类
青海蚕豆种质资源的分子多样性较丰富,其中地方品种的遗传多样性最丰富,与刘玉皎等选用相同种质资源采用形态标记分析遗传多样性的结果一致(刘玉皎和宗绪晓, 2008)。但种质资源分类的结果却不同,应用Structure2.2软件,分析青海蚕豆种质资源的遗传结构后,确定149份资源可分为7个组群,结果比较可靠,而后者只是根据聚类图将其则分为3个组群。种质资源分类结果除与标记方法有关外,与分类方法或分类依据紧密相关。种质资源分类结果表明,种质资源分类并完全是按来源进行聚类的,同一类型的种质资源也具有遗传差异,不同类型的种质资源也同样具有一定的遗传相似性。在青海蚕豆种质资源中,国外种质资源间的遗传距离相对较小,要加强国外优异种质的引进,以丰富青海蚕豆育种的遗传基础。
2.2 核心种质的构建
基于AFLP初步构建了40份青海蚕豆种质资源的核心种质,包含了149份种质资源的80%以上的遗传信息,而杨菁等基于形态标记从153份青海蚕豆种质资源初步构建了21份核心种质,各主要性状核心种质与全部种质资源的符合率在90%以上(杨菁等, 2009),而且两者构建的初步核心种质不尽一致。文中试验材料是杨菁等选用的153份蚕豆种质资源中的149份,两者选用的材料相同,采用的方法不同,结果不同。形态标记中充分考虑了具有特异性状的资源,AFLP标记中考虑了来源,按地方品种、引进种质和育成品种分别构建核心资源。因此,为了不使种质资源的基因漂移,使核心种质资源遗在品种改良中得到有效利用,多种方法结合构建核心种质是必要的。
2.3 AFLP引物
本文采用了10对EcorI/MseI引物对149份青海蚕豆种质资源进行扩增,共扩增出81条多态性条带,多态性比例为46.82%,Shannon's信息指数为0.0946。基于AFLP的遗传相似系数在0.9以上,相似性较高,多态性比较低,为了更好地反映种质资源的遗传多样性,要增加引物数量进一步分析。
3 材料与方法
3.1 材料
研究材料选用从青海省海东地区、海北州、海南州和黄南州等生态条件差异较大的生态区收集的地方品种(101份),为了比较说明,引入国外种质资源(36份)和青海当地选育品种(12份)。引物序列由上海生物工程公司合成,见表5。
表5 AFLP扩增引物序列表 Table 5 The primer sequence of AFLP |
3.2 DNA提取及AFLP反应体系方法
蚕豆DNA提取采用CTAB改良法(刘玉皎等, 2007),采用刘秀菊等的AFLP反应体系(刘秀菊等, 2007)。
3.3 数据统计与分析
采用“0-1”数据统计条带,有带为“1”,无带为“0”。利用Structure 2.2,采用混合模型和等位变异发生频率相关模型对蚕豆种质进行群体遗传结构分析。所设置的Structure参数Burnin,Period和after Burnin为10,000次,K值为1-10,每个K值运行10次。计算每个K值对应的Var[lnP(D)的均值,[LnP(D)]值的均值,然后作出折线图选择最佳K值,即得群体遗传结构的群体数(刘金等, 2009)。用Popgen1.32分析有效等位变异及多态性比例和Shannon-weaver多样性指数、遗传距离等参数(宗绪晓等, 2008);聚类法构建核心种质资源(杨菁等, 2009; 王述民等, 2002)。
作者贡献
刘玉皎是本研究的实验设计、执行以及稿件编写的主要执行人。侯万伟参与部分实验工作。全体作者都阅读并同意最终的文本。
致谢
本研究由国家科技支撑计划(2007BAD59B05)、现代农业产业技术建设专项(nycytx-18-G2)和青海省重点科技攻关计划(2006-G-126)资助。
参考文献
Link W, Dixkens C and Singh M, 1996, Genetic diversity in European and Mediterranean faba germplasm revealed by RAPD markers, Theor. Appl. Genet., (92): 637-643
Liu J. Guan J.P. Xu D.X, Zhang X.Y. Gu J. and Zong X.X., 2008, Phenotypic Diversity of Lentil (Lens culinaris Medik.) Germplasm Resources, Zhiwu Yichuan Ziyuan Xuebao (Journal of Plant Genetic Resources ), 9(2): 173-179 (刘金, 关建平, 徐东旭, 张晓艳, 宗绪晓, 2008, 小扁豆种质资源形态标记遗传多样性分析, 植物遗传资源学报, 9(2): 173-179)
Liu X.J., Ren J.Y., Zong X.X., Guan J.P. and Zhang X.Y., 2007, Establishment and Optimization of AFLP for Faba Bean, Zhiwu Yichuan Ziyuan Xuebao (Journal of Plant Genetic Resources ), 8(2): 153-158(刘秀菊,任俊云, 宗绪晓, 2007, 蚕豆AFLP技术体系的建立与优化, 植物遗传资源学报, 8(2):153-158)
Liu Y.J. and Zong X.X., 2008, Morphological Diversity Analysis of Faba Bean (Vicia. Faba L.) Germplasm Resources from Qinghai, Zhiwu Yichuan Ziyuan Xuebao (Journal of Plant Genetic Resources ), 9(2): 79-83 (刘玉皎, 宗绪晓, 2008, 青海蚕豆种质资源形态多样性分析, 植物遗传资源学报, 9(2): 79-83)
Liu Y.J., Yang J., and Cui Z.S., 2007, Modified DNA Extraction for AFLP analysis in Faba Bean,Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 5(5): 747-750 (刘玉皎, 杨菁, 崔志松, 2007, 适于AFLP分析的蚕豆DNA提取方法的改良, 分子植物育种, 5(5): 747-750)
Polignano G.B., Quintano G., and Bisignano V.,1998, Enzyme polymorphism in faba bean (Vicia faba L. minor) accessions, Genetic interpretation and value for classification,Euphytica 102(2) :169-176 doi:10.1023/A:1018340228500
Polignano G.B., Uggenti P., and Scippa G., 1993, The pattern of genetic diversity in faba bean collections from Ethiopia and Afghanistan, Genetic Resources and Crop Evolution, 40(2) : 71-75 doi:10.1007/BF00052637
Serradilla M., De Mora M.T., and Moreno T., 1993, Geographic dispersion and varietal diversity in Vicia faba L., Genetic Resources and Crop Evolution, 40(3): 143-151 doi:10.1007/BF00051119
Wang S.M., Cao Y.S., and Hu J.P, 2002, Preliminary Establishment of Chinese Adzuki Bean Germplasm Resource Core Collection with Agroecology and Characterization Data, Huabei Nongxuebao (Acta Agriculturae Boreali-Sinica), 17(1): 35-40 (王述民, 曹永生, 胡家蓬, 2002, 中国小豆种质资源核心样品的初步建立, 华北农学报, 17(1): 35-40)
Wang X.J.,Qi X.S. and Wang X.R, 2009, Genetic Diversity Analysis of Main Agronomic Characters in Faba Bean Germplasm, Nongye Xiandaihai Yanjiu (Research of Agricultural Modernization), 30(4): 633-636(王晓娟, 祁旭升, 王兴荣, 2009, 蚕豆种质资源主要性状的遗传多样性分析,农业现代化研究, 30(4):633-636)
Yang J.Chi D.Z. and Liu Y.J., 2009, Preliminary Construction of Core Collection of Broad Bean in Qinghai Based on Morphological Traits, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding),7(3): 599-606 (杨菁, 迟德钊, 刘玉皎, 2009, 基于形态性状的青海蚕豆核心种质的初步构建, 分子植物育种, 7(3): 599-606)
Zeid M, Schön CC, and Link W, 2001, Genetic diversity in a group of recent elite faba bean lines, Czech J Gene Plant Breed,(37): 34-40
Zhang X.T. and Liu Y.J., 2009, Morphological Diversity Analysis of Faba Bean [Vicia. Faba L.] Germplasm Resources, Qinghai Keji (Qinghai Science Technology), 5: 17-19 (张小田, 刘玉皎, 2009, 蚕豆种质资源形态标记遗传多样性分析, 青海科技, 5: 17-19)
Zhou Y.Q, 2005, The Application of DNA marker for Researched Plant, Chemical Industry Press,Beijing,China,pp.162-165 (周延清, 2005, DNA分子标记技术在植物研究中的应用, 化学工业出版社, 中国, 北京, pp.162-165)
Zong X.X., Guan J.P., Wang S.M and Liu Q.C, 2008, Genetic Diversity Chinese Pea (Pisum Sativum L.) Landrace Reveal by SSR Markers, Zhuwu Xuebao ( Acta Agronomic Sinica), 34(8): 1330-1338 (宗绪晓, 关建平, 王述民, 2008, 中国豌豆地方品种种质资源SSR多样性分析, 作物学报, 34(8): 1330-1338)