 作者
作者  通讯作者
通讯作者
计算分子生物学, 2015 年, 第 4 卷, 第 2 篇
收稿日期: 2015年02月26日 接受日期: 2015年02月26日 发表日期: 2015年02月26日
引用格式(中文):
李旭娟, 刘洪博, 林秀琴, 吴转娣, 徐超华, 刘新龙, 2015, 甘蔗KNOX基因 (Sckn1)的电子克隆及生物信息学分析, 基因组学与应用生物学, 34(1): 136-142
引用格式(英文):
Li X.J., Liu H.b., Lin X.Q., Wu Z.D., Xu C.H., and Liu X.L., 2015, In Silico Cloning and Bioinformatics Analysis of KNOX Gene in Sugarcane (Sckn1), Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 34(1): 136-142
KNOX基因对植物顶端分生组织的形成和维持有至关重要的作用。本文选择功能已知的水稻KNOX基因(OSH1, Oskn1)为参考序列,采用电子克隆技术克隆到甘蔗中与OSH1同源的一个KNOX基因Sckn1,并通过生物信息学分析预测该基因的结构、功能。结果表明:该基因包含一个1 071 bp的开放阅读框,编码 356个氨基酸,分子量为 39.36 kD,理论等电点为 6.47。该基因很可能定位于细胞核,起转录调控作用,序列比对发现该基因编码蛋白与高粱、玉米、小米草等 KN1 蛋白高度相似。以上分析结果为进一步 研究 Sckn1基因的功能奠定了坚实的基础。
甘蔗是一种重要的糖料作物和能源植物,其有效部位主要是其地上部分。研究表明,有花植物地上部分器官形成依赖于其顶端分生组织(shoot apical meristem, SAM),而顶端分生组织的形成和维护又受KNOX转录因子调控(Tsuda et al., 2014)。研究者已获得水稻同源异型盒基因OSH1,并证实该基因对水稻顶端分生组织的形成和维护具有重要作用。OSH1属于KNOX家族玉类,与Oskn1是同一基因,参与水稻茎端分生组织形成的调节和维持(Tsuda et al., 2011)。然而,甘蔗中是否也存在该类基因,其功能又如何还不为所知,鉴于此,我们以水稻OSH1为探针来探索甘蔗中相关基因。
电子克隆(in silico cloning)技术是基于生物学数据库中的蛋白质、核酸序列和和表达序列标签(ex-pressed sequence tags, ESTs)等信息,利用生物信息学工具和计算机技术快速获得功能基因的方法(苏亚春等, 2011)。其中基于EST的电子克隆技术是近几年发展起来的一种新的基因快速克隆方法,该方法是利用相近物种的核酸或蛋白为探针,在数据库中搜 索感兴趣基因的 ESTs,再利用生物信息学技术组装延伸ESTs序列,最终获得基因的部分乃至全长 cDNA 序列并进一步利用 RT-PCR 的方法进行克隆分析、验证(胡皝和萧浪涛, 2007)。该技术具有效率高、成本低、针对性强等优点(孟超敏等, 2014)。本研究以水稻OSH1基因序列为种子序列,在NCBI中Blast搜索甘蔗中与种子序列相似性较高的ESTs,然后利用Vector NTI、DNAMAN等软件进行拼接,获得EST重叠群(contig),再以此contig为种子序列,重复上面步骤,直到没有可供延长的EST序列为止。然后将拼接得到的序列在NCBI比对,初步确定正确后,利用在线工具ORF Finder搜寻其正确的开放阅读框(open reading frame, ORF),即获得甘蔗中OSH1的同源基因的cDNA序列,取名Sckn1 (NCBI GenBank: KM880186),再借助相关生物信息学知识及软件分析预测该基因编码蛋白的理化性质、结构、功能等,以期为后续Sckn1的功能研究奠定基础。
1 结果与分析
1.1甘蔗KNOX基因相关EST序列 Vector NTI 11.5 Advance 的拼接
通过水稻OSH1基因(NCBI GenBank: AK107637) 序列在NCBI中Blast搜索到甘蔗中相似性较高的25条序列(NCBI GenBank: CA234507.1, CA275017.1, CA147421.1, CA229919.1, CA184947.1, CA072428.1, CA205078.1, CA148829.1, CF571207.1, CA235280.1, CA242709.1, CA160039.1, CA220958.1, CA200242.1, CA184991.1, CA253948.1, CA212204.1, CA110552.1, CA229847.1, CA242304.1, CA261229.1, CA227414.1, BQ537310.1, CA199909.1, CA225630.1),利用 Vector软件将这些序列拼接,得到 1 591 bp 的 contig。Blast比对初步认为该电子克隆序列就是甘蔗的 KNOX基因(Sckn1)。
1.2 ROC22生长点总 RNA的提取及Sckn1基因扩增
按照试剂盒方法提取 ROC22生长点总 RNA后取 3 滋L 经 1.2%的琼脂糖凝胶电泳检测可见 3 条完整的条带(图1 A),经Bio Drop Lite PC超微量可见紫外分光度计检测RNA OD260/280在2.0左右,说明提取RNA质量较好,符合实验要求。将提取的 RNA反转录成cDNA,以此为模板进行PCR得到Sckn1,PCR产物电泳并进行胶回收(图1 B; 图1 C)。
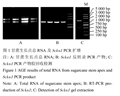 图1 甘蔗生长点总 RNA 及 Sckn1 PCR 扩增 |
1.3 ScKN1 生物信息学分析
1.3.1 ScKN1 的氨基酸理化性质分析
利用 ExPASy服务器中的ProtParam和 Com-pute pI/Wm程序 ,预 测 ScKN1的理化性质( 章文贤等, 2013),结果表1和图2。由表1可知,该蛋白的不稳定系数为 51.45,大于40,说明该蛋白不稳定;平均疏水性为-0.562,参考付海辉等(2010)报道初步推测ScKN1是亲水性氨基酸。由图2可知ScKN1氨基酸组成中含量较多的是Leu和Ser,推测它们可能与ScKN1空间结构的维持有关。采用 Kyte & Doolittle 标度计算,得到ScKN1的亲 / 疏水信号图(图 3)。图3中峰值分布在-0.5以下的比分布在 0.5以上的多,再次表明该蛋白为亲水性蛋白。
 |
 图3 ScKN1氨基酸序列的疏 / 亲水性预测 |
 表1 ScKN1一级结构预测 |
1.3.2 ScKN1蛋白亚细胞定位预测
基因编码的蛋白质需要处于细胞中特定的亚细胞位置上才能行使其功能,因而蛋白的分布与其功能紧密相关,蛋白的亚细胞定位也在一定程度上反映了其对应基因的功能。研究采用PSPORT程序预测ScKN1的可能分布位置,从表2来看,该蛋白分布在细胞核中的可能性最大。
 |
1.3.3 ScKN1的二级结构预测
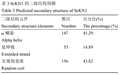
蛋白质的活性和功能与其结构密切相关,因此,研究蛋白空间结构对其功能探讨有重要指导作用,本文参考原晓龙等(2013)的方法,利用SOPMA预测ScKN1的二级结构。由表3可知,ScKN1由琢螺旋、延伸链和无规则卷曲构成,其中无规则卷曲和琢螺旋的比例较高,分别为43.82%和41.29%。图4也更直观地表明该蛋白的二级结构组成中包含无规则卷曲、琢螺旋和延伸链,其中前两者所占比例多。
 |
 |
1.3.4 ScKN1的功能预测
基因功能分类是基因注释过程中的重要环节之一。为了加强对Sckn1的认识,增加对该基因功能了解,本研究对该基因进行了功能预测,功能分类结果(表4)表明,Sckn1主要起转录调控作用,推测其可能是转录因子。
 |
1.3.5 ScKN1的蛋白质结构域与亲缘进化关系分析
以ScKN1为探针,在NCBI中进行Blast比对,结果显示ScKN1包含一个转录调控DNA结合域Homeodomain (HD),KNOX1区域和KNOX2区域,此外还有一个核定位区域ELK(图5),这些区域对植物的形态建成起重要作用,水稻OSH1突变体osh1中缺失HD区域(Tsuda et al., 2011),ELK是核定位区域,KNOX2和HD对诱导转基因作物异常表型起重要作用,KNOX1和ELK可抑制转基因作物中靶基因的表达(Nagasaki et al., 2011)。下载与该探针蛋白相似性较高的蛋白序列通过DNAMAN进行序列比对(图6),发现它们在保守结构域上氨基酸相似度很高,非保守结构域上存在一定变化。利用软件MEGA6.06构建ScKN1与高粱假定蛋白(GenBank: XP_002463952.1)、高粱KN1 (ABC71525.1)、玉米KN1 (NP_001266662.1)、玉米KN1同源蛋白(NP_001105436.1)、小米草KN1 (ABC71528.1)、小米草KN1同源蛋白(XP_004981913.1)、小盼草KN1 (ABC71529.1)、粟米KN1 (ABC71526.1)其它几种植物KN1氨基酸序列进化树(图7),可知ScKN1与小米草同源异型结构域蛋白亲缘关系最近。综上所述,虽然Sckn1的具体功能还有待进一步验证,但本文的分析预测结果为后续实验奠定了基础。
 |
 |
 图7 ScKN1与其它几种植物KN1氨基酸序列进化树 |
2讨论
OSH1基因属于KNOX(knotted1-like homeobox)家族,该家族基因编码同源异型结构域(homeodomain, HD)转录因子。研究发现,HD转录因子作用于赤霉素(GA)氧化酶基因(Bolduc and Hake, 2009)、GA生物合成酶基因(Sakamoto et al., 2001)和细胞分裂素(CK)生物合成酶等基因(Sakamoto et al., 2006),进而调控这两类激素水平,从而影响植物SAM细胞分化。
电子克隆(in silico cloning)是基因克隆的新策略,也是一项伴随着基因组计划和EST计划发展起来的快速克隆基因的方法(Gill and Sanseau, 2000)。随着基因高通量测序技术的迅速发展,数据库中表达序列标签(expressed sequence tags, EST)序列数量与日俱增,利用基于EST的电子克隆获取目标基因的技术应用愈加广泛,目前已运用该方法成功从多种作物中获得了许多新基因,如武安泉等(2013)克隆了小麦Cyp450基因,徐婉莉等(2014)克隆了辣椒actin基因,郝爱平和王婷婷(2014)克隆了高粱SAMDC基因,方静平等(2012)克隆了甘蔗β-1,3-葡聚糖酶基因以及巩元勇等(2013)克隆的陆地棉GhNIP5.J基因等。生物信息学也是一项新兴技术,广泛用于基因功能研究中新基因序列及其所编码的蛋白质结构和功能的详细预测和分析,对新基因序列的分析有很大的指导作用。相信随着基因组序列信息的丰富和生物信息学技术的发展,基因电子克隆技术也将更加精准、高效且备受青睐。
本研究利用基于EST的电子克隆技术从甘蔗中克隆了一个新基因Sckn1,生物信息学分析表明该基因包含一个1 071 bp的开放阅读框,编码356个氨基酸,其蛋白定位于细胞核,可能作为转录因子参与转录调控作用,蛋白二级结构主要由琢螺旋和无规则卷曲和延伸链构成。序列分析发现该蛋白与高粱、玉米和小米草等植物KN1蛋白高度保守,这些为Sckn1的功能研究奠定了良好的基础。
3材料与方法
3.1材料
3.1.1植物材料
本实验所用材料保存于国家甘蔗种质资源圃,选取甘蔗当家品种ROC22的新鲜幼嫩分蘖芽,剥开其生长点剪碎后液氮速冻并用于后续RNA提取。
3.1.2主要试剂
RNA提取试剂盒Trans ZolTM Plant、反转录试剂盒Trans Script One-step gDNA Removal and cDNA Synthesis SuperMix、Trans Taq DNA Polymerase High Fidelity(HiFi)、DNA Marker、克隆载体 PEASY-T5 Zero Cloning Kit 等购于北京全式金生物技术有限公司。
3.1.3生物信息学软件及网上资源
文中所用生物信息学软件及网上资源归纳如表5所示。
 |
3.2方法
3.2.1 ROC22生长点总RNA的提取及反转录
将液氮速冻准备好的植物材料于液氮中迅速研磨成粉末并 按 Trans ZolTM Plant 试 剂盒方 法提取RNA,用1.2%的琼脂糖凝胶和 Bio Drop Lite PC 超微量可见紫外分光度计检测 RNA质量和浓度,然后根据TransScript One-step gDNA Removal and cDNA Synthesis SuperMix试剂盒操作要求反转录成cDNA后于-20℃保存备用。
3.2.2 Sckn1的电子克隆
以水稻OSH1 (NCBIGenBank:AK107637, D16507)为基础,在NCBI (http://blast.ncbi.nlm.nih.gov/Blast. cgi) blast甘蔗的相关EST序列,用 DNAMAN软件对搜索到的EST序列进行拼接,NCBI在线分析初步确定所拼接序列就是甘蔗的KNOX基因序列。利用NCBI中ORF Finder (http://www.ncbi.nlm.nih.gov/p- ro jects/gorf/orfig.cgi)搜索Sckn1的开放阅读框(open read- ingframe, ORF),得到Sckn1的cDNA全长的电子克 隆序列。
3.2.3 RT-PCR扩增Sckn1
以电子克隆序列为模板,利用软件Primer Pre- mier 5.0设计引物如下,以保存的ROC22生长点RNA反转录cDNA为模板,利用引物Sckn1-43-F:5'-AGCCTTTTCCCTGCTGTGC-3';Sckn1-1348-R:5'-GCCCTGCCTTCAGTAGTTC-3'进行PCR扩增,反应程序为:94℃预变性5 min;94℃变性45 s,56℃退火45 s,72℃延伸 1 min,35 个循环;72℃最后延伸10 min,反应结束后,用1.2%的琼脂糖凝胶电泳检测并胶回收纯化,克隆测序分析。50 μL反应体系如表6所示。
 |
3.2.4 ScKN1的生物信息学分析
(1) ScKN1氨基酸的理化性质分析,利用Ex- PASy服务器中的ProtScale和ProtParam 软件预测ScKN1的理化性质。
(2) ScKN1的亚细胞定位:利用在线软件 PSORT (http://psort.hgc.jp/form. html)预测ScKN1 在细胞中的存在部位。
(3)利用 SOP-MA分析预测 ScKN1的二级结构。
(4)通过ProtFun软件(http://www.cbs.dtu.dk/ser-vices/ ProtFun/)预测ScKN1的功能。
作者贡献
刘新龙负责相关实验设计及论文进行修改;李旭娟主要负责实验具体实施、数据结果分析以及论文的撰写;刘洪博、林秀琴和吴转娣协助实验取材以及RNA提取;徐超华负责文献资料的收集与整理工作。
致谢
本研究由国家自然科学基金(31360359)项目资助;感谢国家甘蔗种质资源圃提供实验材料。
Bolduc N., and Ha ke S., 2009, The maize transcription factor KNOTTED1 directly regulates the gibberellin catabolism gene ga2ox1, The Plant Cell, 21(6): 1647-1658
Fu H.H., Xin P.Y., Xu Y.L., Liu Y., Wei Y.J., Dong J., Cao Y.L., and Zhou J., 2011, Bioinformatics analysis of UFGT gene from several economic plants, Jiyinzuxue Yu Yingyong Sheng- wuxue (Genomics and Applied Biology), 30(1): 92-102 (付海辉, 辛培尧, 许玉兰, 刘岩, 韦援教, 董娇, 曹有龙, 周军, 2011, 几种经济植物 UFGT 基因的生物信息学分析, 基因 组学与应用生物学, 30(1): 92-102)
Fang J.P., Su Y.C., You Q., Que Y.X., Xu L.P., and Chen R.K., 2012, In silico cloning and bioinformatic analysis of β-1,3-glucanase gene (ScBG) from sugarcane (Saccharum spp.), Shengwu Xinxixue (Chinese Journalof Bioinformatics), 10(3): 199-207 (方静平, 苏亚春, 游倩, 阙友雄, 许莉萍, 陈如凯, 2012, 甘蔗β-1,3- 葡聚糖酶基因的电子克隆与分析, 生物信息学,10(3): 199-207)
Gill R.W., and Sanseau P., 2000, Rapid in silico cloning of genes using expressed sequence tags (ESTs), Biotechnology Annual Review, 5: 25-44
Gong Y.Y., Guo S.Q., Shu H.M., He L.C., and Ni W.C., 2013, In silico cloning and bioinformatics analysis of aquaporin gene GhNIP5.1 from upland cotton (Gossypium hirsutum), Jiangsu Nongye Xuebao (Jiangsu Journal of Agricultural Sciences), 29 (3): 682-684 (巩元勇, 郭书巧, 束红梅, 何林池, 倪万潮, 2013, 陆地棉 GhNIP5.1 基因的电子克隆及生物信息学分析, 江苏农业学报, 29(3): 682-684)
Hao A.P., and Wang T.T., 2014, In silico cloning and bioinfor- matic analysis of SAMDC gene in sorghum, Xinan Nongye Xuebao (Southwest China Journal of Agricultural Sciences), 27(2): 507-511 (郝爱平, 王婷婷, 2014, 高粱 SAMDC 基因的电子克隆与生物信息学分析, 西南农业学报, 27(2): 507-511)
Hu H., and Xiao L.T., 2007, Application of bioinformatics in full-length cDNA sequence in silicon cloning of novel genes, Shengwu Jishu Tongbao (Biotechnology Bulletin), 4: 93-96 (胡皝, 萧浪涛, 2007, 生物信息学在新基因全长cDNA 电子克隆中的应用, 生物技术通报, 4: 93-96)
Meng C.M., Ji J.H., Li X.L., and Shang J., 2014, Electronic clone and bioinformatics analysis of CPP transcription factor genes from wheat, Shengwu Jishu (Biotechnology), 24(4): 39-42 ( 孟超敏, 姬俊华, 李雪林, 尚家, 2014, 小麦 CPP 转录因子基因 的电子克隆及生物信息学分析, 生物技术, 24(4): 39-42)
Nagasaki H., Sakamoto T., Sato Y., and Matsuoka M., 2001, Functional analysis of the conserved domains of a rice KNOX homeodomain protein, OSH15, The Plant Cell, 13 (9): 2085-2098
Sakamoto T., Kamiya N., Ueguchi-Tanaka M., Iwahori S., and Matsuoka M., 2001, KNOX homeodomain protein directly suppresses the expression of a gibberellin biosynthetic gene in the tobacco shoot apical meristem, Genes & Development, 15(5): 581-590
Sakamoto T., Sakakibara H., Kojima M., Yamamoto Y., Nagasa- ki H., Inukai Y., Sato, Y., and Matsuoka M., 2006, Ectopic expression of KNOTTED1-like homeobox protein induces expression of cytokinin biosynthesis genes in rice, Plant Physiology, 142(1): 54-62
Su Y.C., Li G.Y., Que Y.X., Guo J.L., Xu J.S., and Xu L.P., 2011, In silico cloning and characterization of chitinase gene from sugarcane using bioinformatics tools, Shengwu Xinxixue (China Journal of Bioinformatics), 9(4): 322-330 ( 苏亚春, 李国印, 阙友雄, 郭晋隆, 徐景升, 许莉萍, 2011, 甘蔗几丁 质酶基因的电子克隆与生物信息学分析, 生物信息学, 9(4): 322-330)
Tsuda K., Ito Y., Sato Y., and Kurata N., 2011, Positive autoreg- ulation of a KNOX gene is essential for shoot apical meris- tem maintenance in rice, The Plant Cell, 23(12): 4368-4381
Tsuda K., Kurata N., Ohyanagi H., and Hake S., 2014, Genome-wide study of KNOX regulatory network reveals brassinosteroid catabolic genes important for shoot meristem function in rice, The Plant Cell, 26(9): 3488-3500
Wu A.Q., Wang J.S., Zhang Y.X., and Yin G.H., 2013, Analysis of electronic clone and bioinformatics on the cytochrome P450 gene in wheat(Triticum aestivum L.), Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 29(18): 38-44 (武安泉, 王俊生, 张玉玺, 殷贵鸿, 2013, 小麦 Cyp450 基因的电子克隆与生物信息学分析, 中国农学通报, 29(18):38-44)
Xu W.L., Pei X.L., Jing Z.G., and Xiong Z.L., 2014, Analysis of electronic clone and bioinformatics on the actin gene in pepper (Capsicum annum L.), Jiangsu Nongye Kexue (Jiangsu Agricultural Sciences), 42(5): 46-48 (徐婉莉, 裴徐梨, 荆赞革, 熊自立, 2014, 辣椒 actin 基因电子克隆与生物信息学分析, 江苏农业科学, 42(5): 46-48)
Yuan X.L., Zhou J., Xin P.Y., Dong J., Meng F.X., and Fu H.H., 2013, Bioinformatics analysis of CHI genes in eight plant species, Xinan Linye Daxue Xuebao (Journal of Southwest Forestry University), 33(2): 88-95 ( 原晓龙, 周军, 辛培尧, 董娇, 孟富宣, 付海辉, 2013, 8 种植物 CHI 基因的生物信 息学分析, 西南林业大学学报, 33(2): 88-95)
Zhang W.X., Jiang Y.M., He W.X., Guo W.Y., Huang X.J., and Zheng S.Y., 2013, In silico cloning and expression analysis of 14-3-3 gene in Ganoderma lucidum, Huabei Nongxuebao (Acta Agriculturae Boreali-Sinica), 28(5): 15-22 (章文贤, 蒋咏梅, 贺望兴, 郭文燕, 黄晓菊, 郑素云, 2013, 灵芝 14-3-3 蛋 白基因的电子克隆与表达分析, 华北农学报, 28(5): 15-22)