2 东北林业大学生命学院, 哈尔滨, 150040; 3 东北农业大学农学院, 哈尔滨, 150030
* 同等贡献作者
 作者
作者  通讯作者
通讯作者
计算分子生物学, 2012 年, 第 1 卷, 第 12 篇
收稿日期: 2012年12月21日 接受日期: 2012年12月21日 发表日期: 2012年12月22日
Wang J., Zhang L.W., Liu C.Y., Li Y.H., Chen Q.S., and Hu G.H., 2011, Prediction of pathogenesis related protein in Soybean using HMMER and Blast, Jiyin Zuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 30(6): 649-656 (王晶, 张丽伟, 刘春燕, 李玉花, 陈庆山, 胡国华, 2011, HMMER及同源比对预测大豆病程相关蛋白, 基因组学与应用生物学, 30(6): 649-656)
病程相关蛋白(pathogenesis related proteins, PRs)是病理或病理相关环境下诱导产生的一类蛋白,它 的产生与积累是植物体应答生物或非生物胁迫的主要特征之一。近年来大量 PR 蛋白被鉴定,根据它们的结 构特征,生物功能以及进化关系等将 PR 蛋白分为 14 个家族。然而,在重要的粮食和油料作物的大豆中发现 的 PR 蛋白却很少,本文通过搜索拟南芥、水稻、玉米以及豆科植物所有的已有的 PR 蛋白,根据其保守结构 域利用 BLAST 程序和 HMMER 程序同时预测大豆中可能存在的 PR 蛋白,通过两种方法的预测和比较整 合,共得到大豆 9 个家族的 36 个 PR 蛋白序列。并对它们的连锁群分布、基因结构、基因长度及进化关系进 行了详细的分析。发现 PR 家族成簇分布于 Gm05、Gm10、Gm13、Gm15、Gm17、Gm19 和 Gm20 等几个连锁 群,基因普遍存在序列较短,大部分都小于 1 000 bp,且内含子数目较少,结构相对简单的特点。在 PR4 家族 中,其家族成员亲缘关系都非常相近,而 PR1-4 和 PR1-3 等与该家族其它成员亲缘关系较远的情况。本研究 结果预测的 PR 蛋白为大豆抗病育种以及抗病基因工程研究提供了良好的基础,同时为大豆中其它家族基 因预测研究以及其它物种基因家族研究提供参考方法。
病程相关蛋白(pathogenesis related proteins, PRs)是存在于许多种植物中受病原菌侵染或一些特定化合物处理后新产生的一种或几种蛋白质,后来发现这些蛋白质都与病原菌侵染有关,称为病程相关蛋白。它们可以通过在侵染部位大量产生,形成抵御病原菌的保护屏障,来降低植物的敏感性,形成抗真菌或细菌的活性蛋白(Edreva, 2005)。病程相关蛋白在健康的植物中也有发现,根、衰老的叶子和植物开花期间都发现有病程相关蛋白的表达。功能主要包括:攻击病原物、降解细胞壁大分子释放二级(内源)激发子、分解毒素、结合或抑制病毒外壳蛋白等。最早是在烟草花叶病毒(tobacco mosaic virus, TMV)侵染烟草叶片时检测到PR蛋白的,起初被称为b蛋白,后来人们将其命名为病程相关蛋白(van Loon and van Kammen, 1970)。同一家族PR蛋白同源性较高且功能相近,不同家族的PR蛋白功能不同,大多为酶类,如几丁质酶等(温韵洁等, 2008)。PR蛋白最初分为五大组(PR-1~PR-5),是在烟草中通过分子遗传技术研究来分类,按照电泳迁移率来排序的。每组的成员都有相似的组成(Bol et al., 1990)。PR-1组最丰富,达到叶片总蛋白的1%~2%。PR-5组为类甜蛋白(thaumatin-like protein, TLP)。可降解真菌细胞膜,对真菌,尤其是卵菌纲有很强的抵抗能力(Batalia et al., 1996)。可激活对丝氨酸肽链内切酶有抗性的蛋白质的活性。
根据PR蛋白的结构特点,可以将其分为14个家族(表1) (van Loon et al., 1994; van Loon and van Strien, 1999)。
|
表 1 PR 蛋白识别组成及家族分类(van Loon and van Strien, 1999) Table 1 Recognized and proposed families of pathogenesis-related proteins (van Loon and van Strien, 1999) |
然而,随着研究的进一步深入与完善,又将PR蛋白分为17个家族(王钧, 1995, 植物生理学通讯, 31(4): 312-317, 320),其中PR-15和PR-16为萌发或萌发类似蛋白。目前分别在辣椒(李惠霞等, 2006)、马铃薯(田振东等, 2003)、豌豆(刘红霞等, 2010)、白毛杨(雷杨等, 2011, http://www.paper.edu.cn/index.php/default/eleasepaper/content/201102-239)和小麦(张岗等, 2009)中均有大量的PR蛋白基因的研究,而在大豆中PR蛋白研究较少,仅5个,而有文献报道的研究仅有关于PR1和类甜蛋白两个(Graham, 2005)。因此大量地开发大豆中存在的PR蛋白为大豆抗病功能深入研究和大豆抗病育种都有非常重要的意义。
HMMER是可以用来搜索使用统计模型或概要文件“隐马尔可夫模型”(HMM)的基因序列数据库的一个应用程序包。HMMER3可以从http://hmmer.wustl.edu/下载 HMMER应用程序包。如果尚不知道可信的比对,则可以训练HMM来识别不一致的基因序列中的模式,并将它们大量应用于整个基因组或“表达序列标记”(EST)分析(Finn et al., 2011)。BLAST程序是通过比对未知序列与数据库中的短序列来发现最佳匹配序列的。最初进行扫描就是确定匹配片段,序列的匹配程序由短序列的联配得分总和来决定。打分高的序列被认为是高度的同源的序列。
从功能基因研究的角度来讲,相关的搜索,比如从序列数据库中,找同源的序列,或者对一个个新的基因功能进行鉴定,使用HMMER比使用BLAST有着更高的灵敏度以及更高的搜索速度,但由于二者比对原理的不同结果也不尽相同。
本研究通过收集不同物种的PR蛋白序列,利用HMMER和BLAST的方法预测了大豆中可能存在的PR蛋白序列。并对他们的连锁群分布、基因结构、 基因长度和进化关系进行了详细的分析。
1结果与分析
1.1同源比对获得候选PR蛋白序列
通过同源比对的方法我们获得了许多PR蛋白家族同源的序列,如在PR-1中我们共获得了79条,对这些PR蛋白进行多序列连配,寻找到具有典型同源的蛋白序列。如图1所示为部分PR-1蛋白序列联配后得到的保守结构域部分。
|
图 1 部分 PR-1 蛋白序列联配 注: 豌豆: gi|76873802; Glycine max: gi|82408517, gi|4928711; 水稻: gi|47497165, gi|47497750, gi|34395064; 玉米: gi|260080581, gi|195636216, gi|195606898, gi|226492447, gi|195606704, gi|226503055; 其余全为拟南芥序列 Figure 1 Sequence aligment of partial PR-1 protein Note: Pisum sativum: gi|76873802; Glycine max: gi|82408517, gi|4928711; Oryza sativa: gi|47497165, gi|47497750, gi|34395064; Zea mays: gi|260080581, gi|195636216, gi|195606898, gi|226492447, gi|195606704, gi|226503055; Others were all Arabidopsis thaliana sequences |
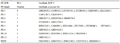
利用同源比对的方法我们可以获得较详细的基 因的信息。表2为通过同源比对预测得到的PR蛋白,其中PR-1和PR-5家族都预测到了6个同源的PR蛋白,而原数据库中PR-13和PR-14家族由于序列较少或同源匹配打分较低而被舍去,所以没有 新的成员被预测到。
|
表 2 同源比对获得大豆候选 PR 蛋白序列及登陆号 Table 2 Sequence and accession number of candidate PR protein |
1.2 HMMER预测获得候选PR蛋白的CDS序列
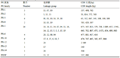
由于是对基因组的CDS进行预测多肽序列构建的蛋白数据库,因此利用HMMER方法在该数据库中预测获得候选PR蛋白仅仅能得到相应的多肽序列、CDS序列及其连锁群分布和CDS长度(表3)。其中PR5家族中预测到的成员较多,有16个,PR-12、PR-13和PR-14家族成员有1个,且在这些家族中的成员普遍序列较短,大部分都小于1 000 bp,只有PR-12家族中预测到了2 589 bp的序列。
|
表 3 HMMER预测获得候选 PR蛋白的CDS序列 Table 3 CDS of candidate PR protein by HMMER |
1.3大豆PR蛋白序列预测及分析
将BLAST和HMMER两种方法预测得到的序列进行比对去掉其中重复的序列,并将剩余序列进行拼接延伸,与NCBI进行预测注释,得到确定的CDS。其中,PR-1、PR-3、PR-5、PR-6、PR-10及PRNF数量上均有减少,PR4家族无重复,而PR-2和PR-12由于存在与其它家族的重复而被去除,可能是由于PR-2与PR-2和PR-1、PR-3家族有相似性,PR-12与PR-13家族存在相似性,而使重复的预测结果被去除。
PR蛋白相对分子质量较低(6~43 kD),在低pH<3下稳定,对蛋白酶有较高的抗性(van Loon and van Strien, 1999),可以在胞内和胞间较好地积累。PR蛋白在进化上相对保守,不同植物的同类型PR蛋白在分子结构和氨基酸组成等方面高度相似,因此我们以E-value小于e-100的同源序列为基因拷贝,为其进行基因定位、序列拷贝数基因数分析,以及基因长度和外显子数(表4),发现PR家族基因普遍存在序列较短,大部分都小于1 000 bp,且内含子数目较少,结构相对简单的特点。
|
表 4 PR蛋白家族成员信息 Table 4 The information of the members in PR family |
对所有的PR蛋白家族成员及拷贝基因进行连锁群分布研究发现:PR蛋白基因主要集中分布于Gm05、Gm10、Gm13、Gm15、Gm17、Gm19和Gm20等几个连锁群,而其它连锁群相对较少。说明PR蛋白基因之间存在着成簇分布的现象特别是同一家族成员之间成簇现象更为严重,如图3所示的大部分的PR5家族成员分布于Gm10连锁群上(图2; 图3)。
|
图 2 PRs 基因在连锁群上的分布 Figure 2 Distribution of genes in PRs families on the LGs |
|
图 3 PRs 家族分布的连锁群 Figure 3 Distribution of PRs families on the LGs |
利用MEGA4对获得的这9个家族36个成员进行进化分析,同一家族成员间大部分进化起源较为相近,如PR4家族亲缘关系都非常相近,同时也存在相同家族进化关系较远的情况,如PR1-4和PR1-3等与该家族其它成员亲缘关系较远。而未分 类家族可能由某些家族进化而来(图4)。
|
图 4 PR蛋白基因的聚类分析 Figure 4 The cluster analysis of PR protein gene |
2讨论
2.1 PR蛋白基因预测的意义和可行性
PR蛋白基因的表达受病原菌侵染、植物发育阶段、激素和胁迫等因素的调节,参与植物的局部和系统诱导抗性。然而,目前对PR蛋白基因的表达调控机理及引起PR蛋白基因表达的信号传导途径知之甚少。所以,PR蛋白基因的预测对PR蛋白基因深入研究抗病调节过程有着重要的作用。而基于PR蛋白序列的保守性和同一家族功能结构域的同一性,使得同样基于相同原理的同源比对和特征分析的HMMER成为有力的预测方法,同时通过二者结合,重复序列的发现相互验证了彼此预测的准确性。但这两种预测方法同时存在着预测结果较少的现象,可能是由于3种原因:(1)PR蛋白家族成员间同源性很高,就使得我们要预测的源序列减少;(2)由于对同源性要求较高,打分低的序列就被排除在外;(3)HMMER的预测原理是整合序列之间的共同特征,再根据这一特征来搜索序列,这也就又使得源序列减少。
2.2 PR蛋白特征分析
PR蛋白基因表达的基本机制是转录活化。同一家族成员间的大部分具有相同的内含子数,同一拷贝之间内含子数也完全相同。不同长度的内含子,内含子的长短与是否接受某种信号有关。大部分成员之间比较集中地分布于几个连锁群上,成簇分布,可能利于在接受刺激信号后的强烈表达或在病原菌侵染后的不断连续产生大量的PR蛋白。并且不同基因家族可接受同一刺激信号而激活表达,可能是由于不同家族之间也存在着同源性,导致有些家族在预测中由于与其它家族预测的重复而被去除;而对同一种刺激信号,不同家族的激活表达可是同步的和协调的,也可是相互抑制的。靶位序列对不同刺激信号表现出特异性,例如番茄中有3种不同的PR蛋白对氨基丁酸的3种异构体反应差异达86%,说明不同家族间的差异序列可能决定着PR蛋白的特异性 (赵淑清和郭剑波, 2003)。
3材料和方法
3.1 PR蛋白序列信息收集和整理
按家族分类分别从NCBI (http://www.ncbi.nlm.nih.gov/)上下载拟南芥(Arabidopsis thaliana)、玉米(Zea mays)、水稻(Oryza sativa)以及豆科植物(Fabaceae)的PR蛋白序列。
共下载拟南芥PR蛋白114个、水稻83个、玉米23个及豆科植物46个,其中大豆的PR蛋白仅有4个。将他们按家族名称分类,共得到PR-1、PR-2、PR-3、PR-4、PR-5、PR-6、PR-10、PR-14及未定家族(pathogenesis-related protein in no family, PRNF)PR蛋白等9类266个PR蛋白序列(表5)。
|
表 5 PR家族分类及成员数目 注: PRNF 为 pathogenisis related protein in no family
Table 5 Classification and numbers of PR families |
3.2大豆蛋白数据库及软件的准备
从NCBI(http://www.ncbi.nlm.nih.gov/)上下载大豆基因组数据库,运用GENSCAN对大豆基因组进行开放阅读框(ORF)的预测,并将得到全基因组的编码序列预测其蛋白序列,建立蛋白质数据库。同时下载大豆的EST 数据库。
从NCBI下载用于进行本地比对的BLAST2.2.16软件包,并下载HMMER3.0软件进行安装。
3.3 PR蛋白通过同源比对预测
将下载的PR蛋白序列进行去重复,对于E值小于0.01的序列可以认定为同源序列,将其去除,按照与大豆亲缘关系由近及远的选择方式仅保留一条非重复序列。将去重复的PR蛋白序列利用tblastn程序与大豆EST 数据进行比对,获得与PR蛋白序列同源的大豆EST序列,即得到候选大豆PR蛋白EST序列。
3.4 PR蛋白的HMMER预测
将下载的拟南芥、玉米、水稻以及豆科植物的PR蛋白序列按家族分别作多序列联配,得到ALIGN文件,并转换成HMMER可识别的文件,并分别保存为seed和align文件。对于成员较少或多序列联配后同源性较差的家族,则通过NCBI同源搜索找出网络数据库中其它同源基因,通过对其它同源基因的多序列联配,得到seed文件。
通过HMMbuild将align文件和seed文件转换成隐马尔科夫模型文件seed.hmm和align.hmm,建立PR蛋白各家族家族的隐马尔科夫模型。
程序命令为“# hmmbuild PR.hmm PR.msf”。
通过HMMsearch用已建立的HMM文件对先前预测的大豆蛋白数据进行比对,E 值设定为HMMER默认值E-value 0.01,得出.out文件。程序命令为“#hmmsearch PR.hmm soybeandatabase>PR.out”。
再根据输出的out文件返回构建的本地蛋白数据库寻找到预测的PR蛋白的多肽序列和CDS序列,作为候选大豆PR蛋白。
3.5大豆PR蛋白序列预测及分析
将两种方法预测的候选大豆PR蛋白EST序列进行整合,去掉重复预测的序列。再对其候选序列进行多轮的拼接电子延伸,并用GENSCAN(http://genes.mit.edu/GENSCAN.html)预测全长ORF。将预测的全长ORF与NCBI进行同源比对,进行基因功能注释以及确定真正的CDS,并将其按家族和顺序进行分类,命名为大豆PR蛋白。
利用Phytozome (http://www.phytozome.net/)对得到的大豆PR蛋白在大豆基因组上进行定位,同时确定其在基因组上的分配情况、拷贝数、外显子内含 子数,以及多拷贝基因间结构变异和进化。
作者贡献
陈庆山老师负责实验设计和指导;王晶和张丽伟负责了软件分析、数据整理和论文写作;刘春燕、李玉花和胡国华老师帮助论文修改。
致谢
本研究由国家自然科学基金项目(30971809)资助,且得到朱命喜同学的指导和大力支持,特此致谢!
Batalia M.A., Monzingo A.F., Roberts W., and Robertus J.D., 1996, The crystal structure of the antifungal protein zea-matin, a member of the thaumatin-like, PR-5 protein family, Nature Struct. Biol., 3(1): 19-23
Bol J.F., Linthorst H.J.M., and Cornelissen B.J.C., 1990, Plant pathogenesis—related proteins induced by virus infection, Annu. Rev. Phytopathol., 28: 113-138
Edreva A., 2005, Pathogenesis-related proteins: Research progress in the last 15 years, Gen. Appl. Plant Physiology, 31 (1-2): 105-124
Finn R.D., Clements J., and Eddy S.R., 2011, HMMER web server: Interactive sequence similarity searching, Nucleic Acids Research, 39(Web Server Issue): W29-W37
Graham M.Y., 2005, The diphenylether herbicide lactofen induces cell death and expression of defense-related genes in soybean, Plant Physiol., 139(4): 1784-1794
Li H.X., Xie B.Y., and Feng L.X., 2006, Accumulation of pathogenesis-related proteins and their activities of pepper plants induced by β-aminonbutyric acid, Yuanyi Xuebao (Acta Horticulturae Sinica), 33(6): 1335-1337 (李惠霞, 谢丙炎, 冯兰香, 2006, β-氨基丁酸诱导辣椒产生PR蛋白及其酶活性的变化, 园艺学报, 33(6): 1335-1337)
Liu H.X., Zhao X., Bi Y., Zhang Z.Y., Chen B.H., and An C.C., 2010, A proteomic approach to study defense-related proteins responses to GSH treatment in pea (Pisum sativum), Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 43 (22): 4746-4753 (刘红霞, 赵鑫, 毕阳, 张增艳, 陈佰鸿, 安成才, 2010, 豌豆病程相关蛋白应答GSH的蛋白质组学分析, 中国农业科学, 43(22): 4746-4753)
Tian Z.D., Liu J., and Xie C.H., 2003, Cloning of a pathogenesis-related protein gene cDNA of potato using RACE methods combined with cDNA library, Yichuan Xuebao (Acta Genetica Sinica), 30(11): 996-1002 (田振东, 柳俊, 谢从华, 2003, cDNA文库与RACE方法结合克隆一个马铃薯病程相关蛋白基因cDNA, 遗传学报, 30(11): 996-1002) (Chinese journal in English)
van Loon L.C., Pierpoint W.S., Boller T., and Conejero V., 1994, Recommendations for naming plant pathogenesis-related proteins, Plant Mol. Biol. Rep., 12(3): 245-264
van Loon L.C., and van Kammen A., 1970, Polyacrylamide discelectrophoresis of the soluble leaf proteins from Nicotiana tabacum var. Samsun and Samsun NN. Ⅱ. changes in pro- tein constitution after infection with tobacco mosaic virus, Virology, 40(2): 190-211
van Loon L.C., and van Strien E.A., 1999, The families of pathogenesis-related proteins, their activities, and comparative analysis of PR-1 type proteins, Physiol. Mol. Plant Pathol., 55(2): 85-97
Wen Y.J., He H.W., Huang Q.S., Liang S., and Bin J.H., 2008, Roles of pathogenesis-relative protein 10 in plant defense response, Zhiwu Shenglixue Tongxun (Plant Physiology Communications), 44(3): 585-592 (温韵洁, 何红卫, 黄群生, 梁山, 宾金华, 2008, 病程相关蛋白10在植物防御反应中的作用, 植物生理学通讯, 44(3): 585-592)
Zhang G., Li Y.M., Zhang Y., Dong Y.L., Wang X.J., Wei G.R., Huang L.L., and Kang Z.S., 2009, Cloning and characterization of a pathogenesis related protein gene TaPR10 from wheat induced by stripe rust pathogen, Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 42(1): 110-116 (张岗, 李依民, 张毅, 董艳玲, 王晓杰, 魏国荣, 黄丽丽, 康振生, 2009, 条锈菌诱导的小麦病程相关蛋白TaPR10基因的克隆及特征分析, 中国农业科学, 42(1): 110-116)
Zhao S.Q., and Guo J.B., 2003, Systemic acquired resistance and signal transduction in plant, Zhongguo Nongye Kexue (Science Agriculture Sinica), 36(7): 781-787 (赵淑清, 郭剑波, 2003, 植物系统性获得抗性及其信号转导途径, 中国农业科学, 36(7): 781-787)
.png)
.png)
.png)
.png)
.png)
.png)
