2. 国家花卉工程技术研究中心, 北京, 100083
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2013 年, 第 11 卷, 第 8 篇 doi: 10.5376/mpb.cn.2013.11.0008
收稿日期: 2013年04月08日 接受日期: 2013年04月28日 发表日期: 2013年05月15日
引用格式(中文):
张杨等, 2013, iTRAQ技术在植物蛋白质组学中的应用, 分子植物育种(online), 11(8): 1047-1051 (doi: 10.5376/mpb.cn.2013.11.0008)
引用格式(英文):
Zhang et al., 2013, Application of iTRAQ Technology in Plant Proteomics, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding), 11(8): 1047-1051 (doi: 10.5376/mpb.cn.2013.11.0008)
随着蛋白质组学的迅速发展,定量研究蛋白质的变化已成为蛋白质组学研究的重要内容之一,相对和绝对定量同位素标记(iTRAQ)技术结合串联质谱及多维液相色谱联用技术作为目前最新、效果较好的蛋白质定性和定量研究的工具,已广泛应用于医学、动植物的蛋白质组学研究中。通过对iTRAQ技术的原理、实验流程、优缺点及近几年在植物蛋白质组学中的研究进展进行详细介绍,以期iTRAQ技术能为植物蛋白质组学研究提供借鉴。
随着拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、杨树(Populustrichocarpa)、番木瓜(carica papaya)等植物全基因组序列的测定,植物基因组学得到不断深入地研究,科学家们发现仅从基因组学水平根本无法完整、系统地揭示生物体的功能。而蛋白质组学直接从蛋白质水平进行研究与转录组和代谢组相互补充,能显著地加深对生命现象与本质的认识和研究,因而越来越受到人们的关注。因蛋白质的种类和浓度对于功能的表达都有着重要的作用,准确识别和定量一个蛋白质组已成为目前研究的重点之一。目前,蛋白质组学定量最常用的技术就是荧光双向差异凝胶电泳技术和iTRAQ技术。荧光双向差异凝胶电泳因其大大提高了传统双向电泳的精确度和可靠性,在前些年已受到广泛应用。但因本身作为凝胶电泳,还是存在很多缺点如受到蛋白丰度、等电点等影响分离等。而iTRAQ技术作为最近几年新开发的技术,不仅能克服传统凝胶电泳的缺点,还能进一步提高蛋白质定量的精确度和可靠性,在动物、人类的研究中已得到广泛的应用,而在植物上只有在拟南芥、水稻等模式植物中有涉及(Sui et al., 2013; Shetty et al., 2012; Fan et al., 2011)。为了使iTRAQ技术更好在植物蛋白质组学中得到应用,本研究对iTRAQ原理,实验流程、优缺点及近几年在植物蛋白质组学中的研究进展进行了详细介绍,以期iTRAQ技术能为植物蛋白质组学研究提供借鉴。
1 iTRAQ技术原理
同位素标记相对和绝对定量(isobaric tags for relative and absolute quantification, iTRAQ)是美国应用生物系统公司ABI研发出的一种多肽体外标记技术(王林纤等, 2010),因其具有精确度高、重复性好、能鉴定低丰度蛋白等特点,在拟南芥、水稻等植物的蛋白质组学中已被广泛使用。iTRAQ试剂(罗治文等, 2006)是一种小分子同重元素化学物质,包括三个部分:即报告基团(reporter group)、平衡基团(balance group)和肽反应基团(peptide reactive group)。其中,报告基团是相对分子质量相差1的同位素,平衡基团是用来补充质量之差,使各个报告基团和平衡基团组合在一起后的相对分子质量相等,肽反应基团的相对分子质量均相等。根据同位素个数的多少,目前有两种试剂盒:一种含有4种同位素标记试剂,一种含有8种同位素标记试剂。可分别进行多达4种和8种不同样品的检测。
iTRAQ试剂几乎可以标记所有蛋白因其中的肽反应基团能连接iTRAQ试剂与肽段的N端及赖氨酸侧链。同时,iTRAQ试剂的质量是相等的,即各标签在标记同一肽段后分子量也相等,所以,在第一级质谱检测结果中,不同样本中的相同肽段在质谱图上都表现为同一个质荷比(罗治文等, 2006)。接着通过串联质谱中的第二级质谱,将上一级分离出的前体离子(precursor ion)碰撞诱导解离,使iTRAQ试剂三部分之间的键断裂,并丢失平衡基团,从而产生低质荷比(m/z)的报告离子产物,即诊断离子(diagnostic ions) (罗治文等, 2006)。诊断离子的相对分子质量比较低,在二级质谱图中更容易与其他离子区别开来,而多肽的相对丰度就是通过比较诊断离子峰的峰高及面积来获得,使结果更加可靠。与此同时,多肽内的酰胺键会发生断裂,产生b离子和y离子,通过在蛋白质数据库中查询和比较对相应的离子片段,就可以鉴定出其对应的蛋白质。
2 iTRAQ技术实验流程
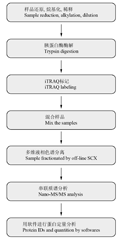
一般情况下,实验流程为:将样品还原、用半胱氨酸封闭;与胰蛋白酶结合酶解蛋白;将酶解获得的肽段进行iTRAQ试剂标记;将标记过的样品混合并通过多维液相色谱分离;将得到的馏分进行MS/MS串联质谱检测及分析(谢秀枝等, 2011) (图1)。质谱检测目前最高级的是用AB SCIEX Triple TOF™ 5600系统,然后用Proteinpilot等软件分析。
因植物多含有多糖、酚等杂质,相比于动物提取蛋白质来说,含量较少、纯度较低,因此在提取时会添加类似硫脲、SDS、DTT等物质来增加蛋白含量,但这些物质会对iTRAQ技术产生影响,因此在进行iTRAQ一般实验流程前,要对样品进行预处理,以消除或减少对iTRAQ试剂的影响。可通过多次丙酮沉淀、PD-10脱盐法等处理。除此,在iTRAQ标记实验中要注意两点:一是不能使用含有伯胺基的试剂,因其能与iTRAQ试剂反应,使试剂失效;二是在标记前要保证水相不能大于30%,因为iTRAQ试剂会发生水解,所以水相的含量要低。
3 iTRAQ技术的优缺点
目前,对复杂体系中的蛋白质进行相对定量主要有以下方法:传统的双向凝胶电泳(two-dimensional electrophoresis, 2-DE)、荧光双向差异凝胶电泳(DIGE)。这两种方法是先通过进行跑电泳然后通过切胶选择差异变化点进行质谱分析来获得数据。还有一类是通过对样品进行同位素标记,经过液相色谱分离后,进行串联质谱得到结果,如ICAT、iTRAQ技术等。其中,目前最常用的就是荧光双向差异凝胶电泳和iTRAQ技术。
荧光双向差异凝胶电泳是首先将不同样品分别进行不同的荧光染料标记,然后将各试验样品在同一块凝胶上进行双向电泳,因此避免了使用不同凝胶在操作上的偶然性和不平行性,大大提高了实验的可靠性、重复性和结果的可信度。但其还是存在很多缺点:一是由于凝胶电泳本身存在缺点,因为蛋白质的分离受到蛋白丰度、等电点、分子量和疏水性等的限制,对于低丰度蛋白、疏水性蛋白等都难以进行有效分离;二是从电泳到质谱不能直接联用,在人工酶切差异点时也会造成较大误差,费时费力;三是所用荧光染料Cy3、Cy5对于鉴定含赖氨酸少的蛋白质有一定的困难。
而iTRAQ技术作为一种新的蛋白定量技术,能克服DIGE中的所有缺点,且相对于其他同位素标记技术有以下优点(王林纤等, 2010):(1)因iTRAQ试剂本身是等量的,同一多肽在被不同同位素的iTRAQ试剂标记后,相对分子质量还保持不变,具有相同质荷比的肽段通过叠加会增加相对丰度,因此提高了检测的效率;(2) iTRAQ试剂能标记所有蛋白,增加了可分析的肽段数量,也提高了蛋白质鉴定的覆盖率和可信度;(3) 质谱检测中,低分子量区域的质谱定性和定量相对更加准确,而iTRAQ中报告基团的相对分子质量较低(113~121),提高了质谱检测的灵敏度和可靠性;(4)通过串联质谱,可在一次串联质谱过程中获得报告基团所对应的多肽丰度和蛋白质前体序列,达到同时比较、鉴定蛋白质的目的,操作步骤更加简单,结果更加可靠;(5)能同时处理高达8个的样品,在实验样品多的情况下能减少实验次数、降低误差,使结果更可靠,同时该试剂的标记效率更高,过程更简单;(6)试剂兼容性好,目前用于蛋白质组学研究的材料均能用iTRAQ技术。Wu等(2006)比较了三种蛋白定量方法(即DIGE, ICAT, iTRAQ)的优劣,结果表明相对来说iTRAQ技术的灵敏度要高于其他方法。但有些情况下,不同方法是可以相互补充的。
4 iTRAQ技术在植物蛋白质组学中的应用
iTRAQ技术在医学、动物学上的应用很是广泛,用于研究不同病理的差异蛋白。而在植物研究上还相对较少,主要集中在拟南芥、水稻、盐芥等模式植物上。
Jones等(2006)在应用iTRAQ技术来比较拟南芥正常与3种不同细菌侵染下的磷酸化作用相关蛋白的变化,结果鉴定出了5个相关蛋白(脱水蛋白, p23分子伴侣, 热激蛋白81, 质粒相关蛋白/原纤蛋白和二磷酸核酮糖羧化酶Rubisco)。
Fukao等(2011)为揭示拟南芥在锌过量的条件下的生长机制,通过应用iTRAQ技术对拟南芥根进行蛋白质组学定量实验,鉴定出了521个蛋白,其中还包括几种质膜相关蛋白。结果还显示出了蛋白定量结果,如铁螯合还原酶量增加,部分V-ATPase下降等。通过研究还发现,过量的锌会导致根毛的分枝和变形。
Lan等(2011)也用iTRAQ技术研究了拟南芥在缺铁条件下蛋白质的变化情况,通过比较缺铁3 d和对照的蛋白质,鉴定出了4 454个蛋白,其中2 882个蛋白被定量,找出了101个差异蛋白。这些蛋白与呼吸作用、S腺苷甲硫胺酸的合成与再生、氧胁迫反应等有关。
Owiti等(2011)也用iTRAQ技术比较了采后0 h到96 h后木薯蛋白的变化情况,分别在采后前期鉴定了67个蛋白,后期鉴定了170个差异蛋白。这些差异蛋白包含有与生理衰败不是十分相关的蛋白,如木薯亚麻苦甙水解酶、谷氨酸蛋白、甘氨酸RNA结合蛋白、果胶甲酯酶、脱氢抗坏血酸还原酶、丙二烯氧化物环氧化酶等。为了确定这些蛋白的可靠性,还选取了一些酶做活性鉴定。
Alvarez等(2009)为了全面研究钙离子对芥菜根的影响,同时用2-DIGE和iTRAQ技术对芥菜根进行蛋白质组学定量实验,结果显示硫同化、氧化平衡和异生物质解毒等相关蛋白含量升高,蛋白合成等蛋白含量下降。虽然两种不同的方法鉴定的蛋白质种类不一样,但是相关功能很类似。
Fan等(2011)为了解黄龙病对甜橙的影响,利用iTRAQ技术比较对照组、感病品种、抗病品种的蛋白变化情况,总共鉴定出了686个蛋白,其中与对照相比,感病品种有20个差异蛋白,抗病品种有10个差异蛋白,与胁迫相关的蛋白含量提高。此实验结果中鉴定出的差异蛋白相对较少,除了与蛋白本身的变化差异小有关系外,与物种的蛋白数据库的建立完善程度也有很大关系。
Marsh等(2010)利用iTRAQ技术比较葡萄在被白粉病侵染不同时间(0, 24 h, 36 h, 48 h, 72 h)后叶片的蛋白质组学定量变化,鉴定出了63个差异蛋白,通过功能分类,这些蛋白与光合作用、代谢、疾病防御、蛋白质合成等相关,表明了葡萄在受疾病侵害时有一定的防御措施。
Wei等(2009)用iTRAQ技术比较了水稻在受褐飞虱取食后的叶片蛋白质表达量的变化,通过比较鉴定出了3个JA合成蛋白,7个氧胁迫应答蛋白、3个蛋白激酶等总共37个差异蛋白。
Shingaki-Wells等(2011)也用iTRAQ技术比较了水稻和小麦胚芽鞘在缺氧条件下蛋白质表达情况,结果表明耐缺氧的水稻胚芽鞘能很快的对缺氧环境作出反应,蛋白质合成和代谢水平都有了很快的变化,与蛋白翻译、抗氧化防御等相关的蛋白含量都增加了,丝氨酸、甘氨酸、丙氨酸等与氨基酸的积累也增加,而不耐缺氧的小麦变化相对就比较小。同时发现,通过外部施加丝氨酸、甘氨酸、丙氨酸等缺氧下积累的氨基酸,不耐缺氧的小麦的耐性也有所提高,说明这些物质的合成有利于提高耐缺氧力。
Han等(2010)为了研究从花粉粒转化成花粉管的作用机理,通过对百合的花粉粒和花粉管进行双水相分区技术获得质膜蛋白,并结合iTRAQ技术鉴定了223个质膜相关蛋白。结果显示超过68%的蛋白与跨膜区和脂质相关,信号转导、膜交易和运输相关蛋白占主要作用。还发现了网格蛋白吞食途径中的很多蛋白种类。
5 iTRAQ技术应用前景及展望
随着基因组计划的完成,基因组学的不断完善,蛋白质组学也已成为当今生命科学研究的一个重点。iTRAQ技术作为最近兴起的一种蛋白定量技术,因其精确度高、重复性好、能鉴定低丰度蛋白等特点,已经在医学、动物学、微生物学等研究中有了较多的应用,相比而言,目前在植物上的应用还比较少,除了几种模式植物的数据库比较全外,其他种类的数据库还很欠缺,在很大程度上阻碍了蛋白质组学的研究。而且目前利用iTRAQ技术主要是研究植物在逆境条件下蛋白表达量的变化,在提高观赏性等方面的蛋白质组学研究还没有见报导,如不同花色、花型、株高、叶色、不同表现型等相关的差异蛋白,在提高果实产量、果实含糖量等方面均还未涉及。因此随着基因组和植物蛋白数据库的不断完善,iTRAQ技术在植物中的应用会有更多空间。
作者贡献
张杨是本综述的执行人,负责相关资料的收集与整理,并完成论文初稿的写作;孙明是项目的构思者及负责人,指导论文的写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由重要花卉种质资源发掘与创新利用(2013BAD01B07)和中央高校基本科研业务费专项资金(YX2011-32)资助。感谢两位匿名的同行评审人的评审建议和修改建议。
参考文献
Alvarez S., Berla B.M., Sheffield J., Cahoon R.E., Jez J.M., and Hicks L.M., 2009, Comprehensive analysis of the Brassica juncea root proteome in response to cadmium exposure by complementary proteomic approaches, Proteomics, 9(9): 2419-2431
http://dx.doi.org/10.1002/pmic.200800478
PMid:19343712
Fan J., Chen C.X.,Yu Q.B., Brlansky R.H.,Li Z.G., and Gmitter F.G., 2011, Comparative iTRAQ proteome and transcriptome analyses of sweet orange infected by "Candidatus Liberibacter asiaticus", Physiologia Plant., 143(3): 235-245
http://dx.doi.org/10.1111/j.1399-3054.2011.01502.x
PMid:21838733
Fukao Y., Ferjani A., Tomioka R., Nagasaki N., Kurata R., Nishimori Y., Fujiwara M., and Maeshima M., 2011, iTRAQ analysis reveals mechanisms of growth defects due to excess zinc in Arabidopsis, Plant Physiol., 155(4): 1893-1907
http://dx.doi.org/10.1104/pp.110.169730
PMid:21325567 PMCid:3091079
Han B., Chen S.X., Dai S.J.,Yang N., and Wang T., 2010, Isobaric tags for relative and absolute quantification- based comparative proteomics reveals the features of plasma membrane-associated proteomes of pollen grains and pollen tubes from Lilium davidii, J. Integr. Plant Biol., 52(12): 1043-1058
http://dx.doi.org/10.1111/j.1744-7909.2010.00996.x
PMid:21106004
Jones A.M., Bennett M.H., Mansfield J.W., and Grant M., 2006, Analysis of the defence phosphoproteome of Arabidopsis thaliana using differential mass tagging, Proteomics, 6(14): 4155-4165
http://dx.doi.org/10.1002/pmic.200500172
PMid:16850419
Lan P., Li W.F., Wen T.N., Shiau J.Y., Wu Y.C., Lin W., and Schmidt W., 2011, iTRAQ protein profile analysis of arabidopsis roots reveals new aspects critical for iron homeostasis, Plant Physiol.,155(2): 821-834
http://dx.doi.org/10.1104/pp.110.169508
PMid:21173025 PMCid:3032469
Luo Z.W.,Zhu L., and Xie W.F.,2006,Advances in isobaric tags for relative and absolute quantitation techiques research, Zhongguo Shengwu Gongcheng Zazhi (China Biotechnology), 26(10): 83-87(罗治文, 朱樑, 谢谓芬, 2006, 同位素标记相对与绝对定量技术研究进展, 中国生物工程杂志, 26(10): 83-87)
Marsh E., Alvarez S., Hicks L.M., Barbazuk W.B., Qiu W.P., Kovacs L., and Schachtman D., 2010, Changes in protein abundance during powdery mildew infection of leaf tissues of Cabernet Sauvignon grapevine (Vitis vinifera L.), Proteomics,10(10): 2057-2064
http://dx.doi.org/10.1002/pmic.200900712
PMid:20232356
Owiti J., Grossmann J., Gehrig P., Dessimoz C., Laloi C.,Hansen M.B., Gruissem W., and Vandrschuren H., 2011, iTRAQ-based analysis of changes in the cassava root proteome reveals pathways associated with post-harvest physiological deterioration, Plant J., 67(1): 145-156
http://dx.doi.org/10.1111/j.1365-313X.2011.04582.x
PMid:21435052
Shetty V., Nickens Z., Testa J., Hafner J., Sinnathamby G., and Philip R.,2012, Quantitative immunoproteomics analysis reveals novel MHC class I presented peptides in cisplatin-resistant ovarian cancer cells, J. Proteomics, 75(11): 3270-3290
http://dx.doi.org/10.1016/j.jprot.2012.03.044
PMid:22504797
Shingaki-Wells R.N., Huang S., Taylor N.L., Carroll A.J., Zhou W., Millar A.H., 2011, Differential molecular responses of rice and wheat coleoptiles to anoxia reveal vovel metabolic Adaptations in amino acid metabolism for tissue tolerance, Plant Physiol., 156(4): 1706-1724
http://dx.doi.org/10.1104/pp.111.175570
PMid:21622811 PMCid:3149938
Sui W.Q., Tang D.Q., Zou T.X., Zou G.M., Chen J.J., Li H., Li L.P., Hou Y.W., Li H.Y., and Dai Y., 2013, Differential proteomic analysis of renal tissue in mesangial proliferative glomerulonephritis using iTRAQ technology, J.Nephrol., 26(1): 191-198
http://dx.doi.org/10.5301/jn.5000124
PMid:22641580
Wang L.Q., Dai Y., and Tu Z.G., 2010, Itraq labeling and biomarker discovery in comparative proteomic studies, Shengming De Huaxue (Chemistry of Life), 30(1): 135-140(王林纤, 戴勇, 涂植光, 2010, iTRAQ标记技术与差异蛋白质组学的生物标志物研究, 生命的化学, 30(1): 135-140)
Wei Z., Hu W., Lin Q.S., Cheng X.Y., Tong M.J., Zhu L.L., Chen R.Z., and He G.C., 2009, Understanding rice plant resistance to the brown planthopper (Nilaparvatalugens) : a proteomic approach, Proteomics, 9(10): 2798-2808
http://dx.doi.org/10.1002/pmic.200800840
PMid:19405033
Wu W.W., Wang G.H., Baek S.J., and Shen R.F., 2006, Comparative study of three proteomic quantitative methods, DIGE, cICAT, and iTRAQ, using 2D-Gel or LC/MALDI TOF/TOF, J. Proteome Res., 5(3): 651-658
http://dx.doi.org/10.1021/pr050405o
PMid:16512681
Xie X.Z., Wang X., Liu L.H., Dong S.L., Pi X.E., and Liu W., 2011, ITRAQ technology and its application in proteomics, Zhongguo Shengwu Huaxue Yu Fenzi Shengwu Xuebao (Chinese Journal of Biochemistry and Molecular Biology), 27(7): 616-621 (谢秀枝, 王欣, 刘丽华, 董世雷, 皮雄娥, 刘伟, 2011, iTRAQ技术及其在蛋白质组学中的应用, 中国生物化学与分子生物学报, 27(7): 616-621)