 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2013 年, 第 11 卷, 第 12 篇 doi: 10.5376/mpb.cn.2013.11.0012
收稿日期: 2013年05月05日 接受日期: 2013年05月07日 发表日期: 2013年05月23日
张安等, 2013, 三浅裂野牵牛(Ipomoea trifida)(2X)谷胱甘肽过氧化物酶(GPX)基因的克隆与生物信息学分析, 分子植物育种(online), 11(12): 1082-1088 (doi: 10.5376/mpb.cn.2013.11.0012)
引用格式(英文):
Zhang et al., 2013, Cloning and Bioinformatical Analysis of Glutathione Peroxidase from Ipomoea trifida (2X), Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding), 11(12): 1082-1088 (doi: 10.5376/mpb.cn.2013.11.0012)
为了研究三浅裂野牵牛抗逆相关基因,根据转录组拼接结果设计特异引物,进行RT-PCR扩增,首次获得了三浅裂野牵牛谷胱甘肽过氧化物酶编码区序列,并进行生物信息学分析。经测序发现,此序列包含510 bp的完整开放阅读框,软件预测可以编码169个氨基酸残基的多肽,预测多肽分子量大小约为18.7 kDa,等电点为6.32,在GenBank中登录,登录号是KC961953,并命名为ItGPX。ItGPX有大段亲水区,推测在细胞核中起到抗氧化作用。系统发育分析表明ItGPX和甘薯IbGPX最为接近,使用PSIPRED和3D-JIGSAW分别预测了ItGPX的二级和三级结构。三浅裂野牵牛谷胱甘肽过氧化物酶基因的克隆为甘薯抗逆育种提供了新的基因资源。
植物在生长过程中经常面对各种逆境,如高温、干旱、低温、盐碱等,这些胁迫会使植物产生大量活性氧(reactive oxygen species, ROS),造成氧化胁迫(Bartels and Sunkar, 2005; Mahajan and Tuteja, 2005)。如果不能及时清除这些活性氧,就会造成植物细胞损伤,最终导致植物生长发育受阻,甚至死亡(Mittler et al., 2004)。谷胱甘肽过氧化物酶(glutathione peroxidase, GPX)是广泛存在于生物体内的一种活性氧清除剂,它可以利用谷胱甘肽(glutathione, GSH)清除活性氧来保护植物免受氧化伤害(Noctor et al., 2002)。目前对动物GPX的研究比较多,对植物GPX研究相对少一些。近年来人们陆续从多种植物中分离出GPX基因,如柑橘(Holland et al., 1993)、拟南芥(Sugimoto and Sakamoto, 1997)、番茄(Depège et al., 1998)等。通过转基因方法过表达GPX提高了植物的抗逆性(Chen et al., 2004; Miao et al., 2006)。相反,降低GPX的表达使得植物的抗逆性降低(Chang et al., 2009),说明GPX在作物抗逆育种中具有潜在应用价值。
甘薯近缘野生种中含有非常丰富的抗性基因资源(Chappell and Rausher, 2011; Liu et al., 2012; Meira et al., 2012)。从野生种中克隆相关基因,不仅丰富了对这些基因的理论认识,而且可以为甘薯育种提供优异基因资源。利用野生种培育甘薯新品种是解决甘薯遗传背景狭窄、育种进程缓慢的策略之一(Liu, 2011)。甘薯种植大部分集中在山区,生长条件相对恶劣,甘薯抗逆育种显得相对重要。
三浅裂野牵牛(Ipomoea trifida)(2X)是甘薯近缘野生种,也是甘薯祖先种之一(Srisuwan et al., 2006)。三浅裂野牵牛不仅具有优异的抗性基因资源(Komiyama et al., 2006),而且能够和甘薯栽培种杂交,产生可育后代(Orjeda et al., 1991),因此三浅裂野牵牛在甘薯育种中有重要价值。为了分离三浅裂野牵牛中的抗性基因,本实验室利用RNA-Seq研究三浅裂野牵牛转录组,从拼接结果中获得一条序列(comp355_c0_seq1),经BLAST搜索发现这条序列是GPX的同源序列。本文根据这条序列设计特异引物,提取三浅裂野牵牛根、茎、叶、花混合样品总RNA,经反转录获取第一链cDNA,后经PCR扩增获得了三浅裂野牵牛GPX基因的编码区,并做生物信息学分析,为进一步构建植物表达载体、研究基因功能提供方便,为培育抗逆甘薯新品种提供基因资源。
1结果与分析
1.1 ItGPX基因克隆与序列分析
从转录组拼接结果中获得一条序列(comp355_c0_seq1)(图1),经过BLAST比对发现这条序列与甘薯IbGPX (GenBank登录号: JQ906089)、杨树PtGPX (XM_002299500)、烟草NtGPX (AB041518)的核苷酸一致性分别到达99%、80%和79%,氨基酸一致性分别达到99%、81%和78%。
|
图1 ItGPX cDNA核苷酸序列和预测编码氨基酸序列 Figure 1 cDNA nucleotide sequence and amino acid residue sequence of ItGPX |
使用LaserGene分析发现这段序列包括一段510 bp的开放阅读框(open reading frame, ORF)(图1灰色部分所示),根据ORF两端序列设计特异引物,使用RT-PCR扩增获得大小约为500 bp的片段(图2)。经测序发现PCR所得序列与转录组拼接序列完全一致,将这段序列命名为ItGPX (GenBank登录号: KC961953)。从NCBI获取18个物种的GPX氨基酸序列进行进化发育分析,结果显示ItGPX与甘薯IbGPX关系最近(图3)。
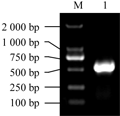 图2 ItGPX基因cDNA扩增结果 Figure 2 PCR amplification of ItAPX gene from cDNA first strand |
 图3 GPX进化发育分析 Figure 3 Phylogenetic analysis of GPX |
1.2 ItGPX蛋白理化性质的软件分析
用软件预测ItGPX的ORF区域编码169个氨基酸,分子量约为18 696.2,理论等电点约为6.32,负电荷残基(Asp+Glu)为21,正电荷残基(Arg+Lys)为21,不稳定系数(Instability Index)为32.69,故属于稳定蛋白,以赖氨酸(Lys)含量最高,达到10.7%,并含有3个半胱氨酸(Cys43, Cys72和Cys91)。存在大段亲水区(Met1-Gly31, Gly44-Leu65, Gln74-Arg89, Cys91-Ala96, Asp105-Pro113, Phe137-Leu165)(图4A),推测ItGPX属于亲水蛋白,且不含有信号肽序列(图4B)和跨膜区(图4C)。亚细胞定位预测结果表明ItGPX很可能位于细胞核中(nucl: 7.0, chlo: 4.0, extr: 1.0, pero: 1.0),与亲水性和跨膜预测结果相符合。磷酸化修饰位点分析显示ItGPX有3个可修饰位点Ser6、Tyr50和Ser154。
1.3 ItGPX高级结构预测
保守结构域分析表明ItGPX属于谷胱甘肽过氧化物酶家族(图4D)。经PSIPRED预测表明ItGPX包括5个α-螺旋、6个β-折叠和多个无规则卷曲(图5A),使用3D-JIGSAW同源建模,结果如图5B所示。
 图4 ItGPX理化性质分析及结构域预测 Figure 4 Physical and chemical property analysis and domain prediction of ItGPX |
 图5 ItGPX二级结构和三级结构预测 Figure 5 Secondary structure and 3D structure prediction of ItGPX |
2讨论
RNA-Seq是近年来发展起来的一种测序技术,广泛应用于转录组测序、新基因挖掘、可变剪接等研究领域(Grabherr et al., 2011)。得益于RNA-Seq的出现,使得甘薯研究进入一个新的阶段(Tao et al., 2012; Wang et al., 2010; Xie et al., 2012; Zhou et al., 2012)。本实验室也开始尝试使用这种方法研究甘薯野生资源,希望从中得到一些基因以供甘薯育种利用。
本文根据转录组拼接结果设计特异引物,使用RT-PCR扩增得到了ItGPX序列,经测序发现两者序列完全一致,说明RNA-seq拼接结果的正确性和可靠性,也表明使用RNA-seq拼接序列为基因克隆提供诸多便利。
我们对ItGPX编码的蛋白进行了分析和预测。植物GPX是一个包括多种同工酶的家族,这些同工酶在亚细胞位置、亚基结构、一级结构和酶学特点上显著不同,但都具有高度保守区域(Faltin et al., 2010)。与动物GPX活性中心的重要构成成分是硒代半胱氨酸不同,植物GPX 的氨基酸序列携带的是一个半胱氨酸残基,即植物中的GPX酶蛋白不含有硒(Faltin et al., 2010)。本文所获得的ItGPX也在Cys43处替代了动物GPX的硒代半胱氨酸,并且具有3个标志性Cys,说明ItGPX属于谷胱甘肽过氧化物酶家族。
分析结果还显示ItGPX有多个磷酸化修饰位点,定位于细胞核内,推测ItGPX可能在细胞核内发挥作用,可能会保护DNA免受氧化损伤。GPX一般只作为胁迫抗氧化的指标之一,近年来发现GPX不仅有抗氧化的功能,还能和其他蛋白相互作用(Holmes-Davis et al., 2005)。因此,ItGPX的确切功能值得深入研究。
ItGPX和甘薯IbGPX核苷酸序列和氨基酸序列高度一致性(99%)以及进化发育分析结果表明两者的亲缘关系十分密切,与前人分析结果基本一致(Gao et al., 2011; Srisuwan et al., 2006),在一定程度上佐证了甘薯起源于三浅裂野牵牛的假设。甘薯是一种重要的粮食作物,同时也是重要的饲料和工业原料。但是近年来发现甘薯育种亲本有限,导致遗传背景狭窄、育种进程缓慢,人们再次考虑从野生资源中引入优异基因来提高甘薯某些方面的特性(Liu, 2011)。本文首次从甘薯祖先种三浅裂野牵牛中克隆了一个具有完整编码区的GPX基因,并附加了酶切位点,可直接用于构建表达载体,为下一步研究该基因的功能和甘薯抗逆转基因育种提供了基础。
3材料与方法
3.1实验材料
实验材料为三浅裂野牵牛二倍体,保存于江苏徐州甘薯研究中心资源圃内。收集根、茎、叶和花速冻于液氮中备用。
3.2实验方法
3.2.1转录组测序和拼接
转录组测序和拼接等工作由上海美吉生物技术公司完成。
3.2.2总RNA的提取及cDNA第一链的合成
总RNA提取方法参照高纯总RNA快速提取试剂盒(Trizol-离心柱型)(上海捷瑞)说明书。经核酸测定仪和琼脂糖凝胶电泳检测产量和质量后,保存于-80℃超低温冰箱中备用。cDNA第一链的合成方法参照HiScriptTM 1st Strand cDNA Synthesis Kit (南京Vazyme)说明书。
3.2.3 PCR扩增
根据本实验室前期三浅裂野牵牛转录组拼接结果设计特异引物,5'端引物序列为:5'- TGCTCTAGAATGGCCGCAGAATCAAGC-3’(下划线部分为XbaⅠ酶切位点及保护碱基);3'端引物序列为:5'- CGAGCTCTTAAGATATCCCCATCAG-3' (下划线部分为SacⅠ酶切位点及保护碱基)。引物由上海生工合成。以cDNA第一链为模板进行PCR 扩增,反应体系为(20 μL):10×Buffer 2 μL,10 mmol/L dNTP 0.5 μL,10 pmol/L引物1 μL,cDNA 1 μL,5 U/μL Taq酶0.2 μL,ddH2O 14.3 μL。反应程序为:94℃预变性4 min;94℃变性45 s,55℃退火30 s,72℃延伸1 min,35次循环;72℃延伸10 min,4℃保存。
3.2.4 PCR产物的克隆
PCR产物经1.5%琼脂糖凝胶电泳检测后,用GenClean柱式琼脂糖凝胶DNA回收试剂盒(上海捷瑞)回收。回收产物与pTG dual-T (上海捷瑞)载体连接,连接产物转化DH5α感受态细胞,随机挑取6个抗性克隆,经菌落PCR检验后,将3个阳性克隆送至南京思普金生物科技公司测序。
3.2.5生物信息学分析
利用生物信息学分析软件对所得基因序列及其编码蛋白进行生物信息学分析和预测。具体分析内容和工具参见表1。
 表1 本文所用生物信息学工具及网页地址 Table 1 The bioinformatic tools and website addresses referred in this study |
3.2.6图片处理与显示
所有图片都经过Adobe PhotoShop CS6处理。蛋白质3D结构使用Chimera (http://plato.cgl.ucsf.edu/chimera/download.html)显示。
作者贡献
张安是本研究的实验设计和实验研究的执行人;曹清河完成数据分析,论文初稿的写作;赵冬兰和周志林参与实验设计,试验结果分析;唐君是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改,是本文的责任作者(通信作者)。全体作者都阅读并同意最终的文本。
致谢
本研究由国家甘薯产业技术体系(CARS-11-B-02-2012)、农业部948子课题(2011-G1-20)和国家863子课题(2012AA101204)共同资助。
参考文献
Bartels D., and Sunkar R., 2005, Drought and salt tolerance in plants, Critical Reviews in Plant Sciences, 24(1): 23-58
http://dx.doi.org/10.1080/07352680590910410
Chang C.C., Slesak I., Jordá L., Sotnikov A., Melzer M., Miszalski Z., Mullineaux P.M., Parker J.E., Karpinska B., and Karpinski S., 2009, Arabidopsis chloroplastic glutathione peroxidases play a role in cross talk between photooxidative stress and immune responses, Plant Physiol., 150(2): 670-683
http://dx.doi.org/10.1104/pp.109.135566
PMid:19363092 PMCid:2689974
Chappell T.M., and Rausher M.D., 2011, Genetics of resistance to the rust fungus Coleosporium ipomoeae in three species of morning glory (Ipomoea), PLoS One, 6(12): e28875
http://dx.doi.org/10.1371/journal.pone.0028875
PMid:22194936 PMCid:3237545
Chen S., Vaghchhipawala Z., Li W., Asard H., and Dickman M.B., 2004, Tomato phospholipid hydroperoxide glutathione peroxidase inhibits cell death induced by Bax and oxidative stresses in yeast and plants, Plant Physiol., 135(3): 1630-1641
http://dx.doi.org/10.1104/pp.103.038091
PMid:15235116 PMCid:519077
Depège N., Drevet J., and Boyer N., 1998, Molecular cloning and characterization of tomato cDNAs encoding glutathione peroxidase-like proteins, Eur. J. Biochem., 253(2): 445-451
http://dx.doi.org/10.1046/j.1432-1327.1998.2530445.x
PMid:9654095
Faltin Z., Holland D., Velcheva M., Tsapovetsky M., Roeckel-Drevet P., Handa A.K., Abu-Abied M., Friedman-Einat M., Eshdat Y., and Perl A., 2010, Glutathione peroxidase regulation of reactive oxygen species level is crucial for in vitro plant differentiation, Plant Cell Physiol., 51(7): 1151-1162
http://dx.doi.org/10.1093/pcp/pcq082
PMid:20530511
Gao M., Ashu G.M., Stewart L., Akwe W.A., Njiti V., and Barnes S., 2011, Wx intron variations support an allohexaploid origin of the sweetpotato [Ipomoea batatas (L.) Lam], Euphytica, 177(1): 111-133
http://dx.doi.org/10.1007/s10681-010-0275-z
Grabherr M.G., Haas B.J., Yassour M., Levin J.Z., Thompson D.A., Amit I., Adiconis X., Fan L., Raychowdhury R., Zeng Q.D., Chen Z.H., Mauceli E., Hacohen N., Gnirke A., Rhind N., di Palma F., Birren B.W., Nusbaum C., Lindblad-Toh K., Friedman N., and Regev A., 2011, Full-length transcriptome assembly from RNA-Seq data without a reference genome, Nature Biotechnology, 29: 644-652
http://dx.doi.org/10.1038/nbt.1883
PMid:21572440 PMCid:3571712
Holland D., Ben-Hayyim G., Faltin Z., Camoin L., Strosberg A.D., and Eshdat Y., 1993, Molecular characterization of salt-stress-associated protein in citrus: protein and cDNA sequence homology to mammalian glutathione peroxidases, Plant Mol. Biol., 21(5): 923-927
http://dx.doi.org/10.1007/BF00027124
PMid:8467085
Holmes-Davis R., Tanaka C.K., Vensel W.H., Hurkman W.J., and McCormick S., 2005, Proteome mapping of mature pollen of Arabidopsis thaliana, Proteomics, 5(18): 4864-4884
http://dx.doi.org/10.1002/pmic.200402011
PMid:16247729
Komiyama A., Sano Z., Murata T., Matsuda Y., Yoshida M., Saito A., and Okada Y., 2006, Resistance to two races of Meloidogyne incognita and resistance mechanism in diploid Ipomoea trifida, Breeding Science, 56(1): 81-83
http://dx.doi.org/10.1270/jsbbs.56.81
Liu G., Huang Q.Q., Lin Z.G., Huang F.F., Liao H.X., and Peng S.L., 2012, High tolerance to salinity and herbivory stresses may explain the expansion of Ipomoea cairica to salt marshes, PLoS One, 7(11): e48829
http://dx.doi.org/10.1371/journal.pone.0048829
PMid:23166596 PMCid:3499518
Liu Q.C., 2011, Sweet potato omics and biotechnology in China, Plant Omics Journal, 4(6): 295-301
Mahajan S., and Tuteja N., 2005, Cold, salinity and drought stresses: an overview, Arch. Biochem. Biophys., 444(2): 139-158
http://dx.doi.org/10.1016/j.abb.2005.10.018
PMid:16309626
Meira M., da Silva E.P., David J.M., and David J.P., 2012, Review of the genus Ipomoea: traditional uses, chemistry and biological activities, Rev. Bras. Farmacogn., 22(3): 682-713
http://dx.doi.org/10.1590/S0102-695X2012005000025
Miao Y.C., Lv D., Wang P.C., Wang X.C., Chen J., Miao C., and Song C.P., 2006, An Arabidopsis glutathione peroxidase functions as both a redox transducer and a scavenger in abscisic acid and drought stress responses, Plant Cell, 18(10): 2749-2766
http://dx.doi.org/10.1105/tpc.106.044230
PMid:16998070 PMCid:1626619
Mittler R., Vanderauwera S., Gollery M., and Van Breusegem F., 2004, Reactive oxygen gene network of plants, Trends Plant Sci., 9(10): 490-498
http://dx.doi.org/10.1016/j.tplants.2004.08.009
PMid:15465684
Noctor G., Gomez L., Vanacker H., and Foyer C.H., 2002, Interactions between biosynthesis, compartmentation and transport in the control of glutathione homeostasis and signalling, J. Exp. Bot., 53(372): 1283-1304
http://dx.doi.org/10.1093/jexbot/53.372.1283
PMid:11997376
Orjeda G., Freyre R., and Iwanaga M., 1991, Use of Ipomoea trifida germ plasm for sweet potato improvement. 3. development of 4x interspecific hybrids between Ipomoea batatas (L.) Lam. (2n=6x=90) and I. trifida (H.B.K) G. Don. (2n=2x=30) as storage-root initiators for wild species, Theor. Appl. Genet., 83(2): 159-163
http://dx.doi.org/10.1007/BF00226245
Srisuwan S., Sihachakr D., and Siljak-Yakovlev S., 2006, The origin and evolution of sweet potato (Ipomoea batatas Lam.) and its wild relatives through the cytogenetic approaches, Plant Sci., 171(3): 424-433
http://dx.doi.org/10.1016/j.plantsci.2006.05.007
PMid:22980213
Sugimoto M., and Sakamoto W., 1997, Putative phospholipid hydroperoxide glutathione peroxidase gene from Arabidopsis thaliana induced by oxidative stress, Genes Genet. Syst., 72(5): 311-316
http://dx.doi.org/10.1266/ggs.72.311
PMid:9511228
Tao X., Gu Y.H., Wang H.Y., Zheng W., Li X., Zhao C.W., and Zhang Y.Z., 2012, Digital gene expression analysis based on integrated de novo transcriptome assembly of sweet potato [Ipomoea batatas (L.) Lam], PLoS One, 7(4): e36234
http://dx.doi.org/10.1371/journal.pone.0036234
PMid:22558397 PMCid:3338685
Wang Z.Y., Fang B.P., Chen J.Y., Zhang X.J., Luo Z.X., Huang L.F., Chen X.L., and Li Y.J., 2010, De novo assembly and characterization of root transcriptome using Illumina paired-end sequencing and development of cSSR markers in sweetpotato (Ipomoea batatas), BMC Genomics, 11: 726
http://dx.doi.org/10.1186/1471-2164-11-726
PMid:21182800 PMCid:3016421
Xie F., Burklew C.E., Yang Y., Liu M., Xiao P., Zhang B., and Qiu D., 2012, De novo sequencing and a comprehensive analysis of purple sweet potato (Impomoea batatas L.) transcriptome, Planta, 236(1): 101-113
http://dx.doi.org/10.1007/s00425-012-1591-4
PMid:22270559
Zhou Y.J., Gao F., Liu R., Feng J.C., and Li H.J., 2012, De novo sequencing and analysis of root transcriptome using 454 pyrosequencing to discover putative genes associated with drought tolerance in Ammopiptanthus mongolicus, BMC Genomics, 13: 266
http://dx.doi.org/10.1186/1471-2164-13-266
PMid:22721448 PMCid:3407029