 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2010 年, 第 8 卷, 第 1 篇 doi: 10.5376/mpb.cn.2010.08.0001
收稿日期: 2010年07月12日 接受日期: 2010年09月10日 发表日期: 2010年09月26日
周长品等, 2010, PCR产物直接测序和混合克隆测序进行桉树EST-SSR标记开发, 分子植物育种 Vol.8 No.1 (doi: 10.5376/mpb.cn.2010.08.0001)
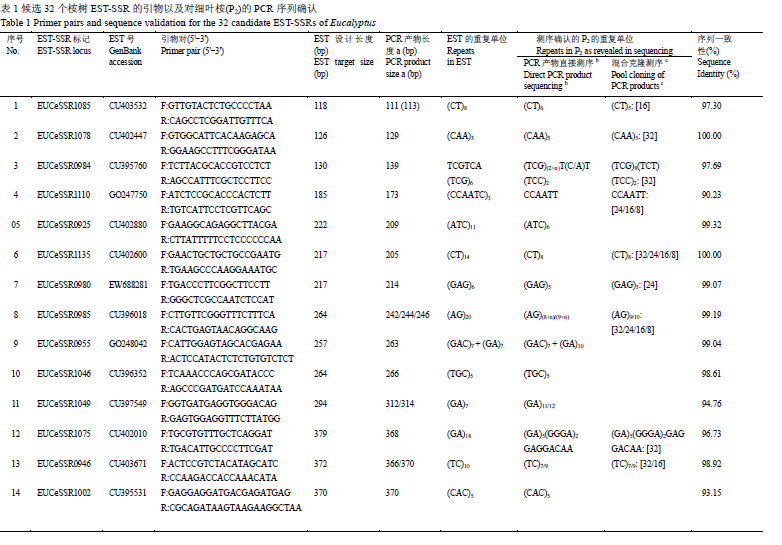
EST-SSR 标记开发中,合适的测序方案是保障序列质量、控制实验成本和提高实验效率的关键。本研究利用1株细叶桉(P2) 对 PCR 产物大小不同的32个桉树候选 EST-SSR 标记进行了 PCR 产物直接测序和混合克隆测序的比较。PCR 产物直接测序成功的标记有21个(65.6%),但有4个标记因简单重复序列邻近测序引物未能确认重复单位数、8个标记因在P2 中等位片段长度杂合而在重复单位后面的测序峰图中显示双峰。在8个、16个、24个和全部32个 EST-SSR 对 P2 的PCR 产物的混合克隆测序中,测序成功的标记分别为6个(75.0%)、11个(68.8%)、11个(45.8%)和14个(43.8%)。综合考虑克隆转化与测序的效率、成本和时间,推荐16个 PCR 产物为混合克隆测序的最佳数量。本研究中共有27个桉树EST-SSR 标记成功测序,与源 EST 比对的序列一致性介于90.23%和100%之间,包括在 P2 中片段长度杂合的标记10个、纯合的17个;在长度纯合的17个标记中,有8个在 P2 中存在1个以上的个体内 SNP。
桉树是桃金娘科(Myrtaceae)桉属(Eucalyptus)、杯果木属(Angophora)和伞房桉属(Corymbia)树种的简称,共有808种和137个变种(Hill and Johnson, 1995)。桉树的天然分布主要是澳大利亚、巴布亚新几内亚和印度尼西亚及其附近岛屿,因其具有速生、丰产、适应性广和用途多样等特点,在热带和亚热带地区广为引种,全球人工桉树林面积已达1 780万公顷以上(FAO, 2000)。
简单序列重复(simple sequence repeats, SSR),或称微卫星,在基因组中数量众多,作为分子标记具有重复性好、共显性和变异性高等特点(Tauz, 1989; Powell et al., 1996),在生物学有关研究中应用广泛(方宣钧等, 2001)。目前,桉树上已开发的SSR标记相对较少,包括367个基因组SSRs (Brondani et al., 2006)、35个叶绿体SSRs (Steane et al., 2005)和20个表达序列标签(expressed sequence tag, EST) SSRs,或称EST-SSR(Faria et al., 2010)。其中,EST-SSR因与基因相关,在近缘物种中的通用性较高(Varshney et al., 2005; Ellis and Burke, 2007),与功能基因的连锁不平衡也可能较强(Ayers et al., 1997)。公共数据库中日益增多的EST序列也为EST-SSR开发提供了便利。截止2010年7月12日GenBank中已有桉属EST序列37 751条(http://www.ncbi.nlm.nih.gov/nucest/?term=Eucalyptus)。分析表明,桉属EST序列约15%含有SSR (李淑娴等, 2010),非冗余EST序列中25%以上含有SSR (Rabello et al., 2005; Ceresini et al., 2005)。
虽然,在许多物种中开展了EST-SSR标记开发研究,但极少确认聚合酶链式反应(polymerase chain reaction, PCR)产物与源EST序列的一致性。据我们所知,只有3篇报道进行了部分EST-SSRs的序列确认,包括鹰嘴豆(Cicer arietinum L.) 60个EST-SSR标记中的5个(Choudhary et al., 2009)、生菜(Lactuca sativa L.) 61个标记中的30个(Simko, 2009)和茶树(Camellia sinensis L.) 61个标记中的部分标记(Sharma et al., 2009)。由于引物的低特异性可能导致PCR错误扩增,从而产生非目的片段或假阳性片段(Cha and Thilly, 1995; Kunkel and Bebenek, 2000; 张晓红等, 2009),因此,对于包括SSR在内的序列标签位点(sequence tagged site, STS)的序列确认是非常必要的。
PCR产物一般可以通过两种方法测序,即直接测序和克隆后的菌落测序。前者操作上相对快捷,但当PCR产物不纯时测序结果较难判读(张晓红等, 2009);后者针对单菌落测序,模板单一,可以获得理想的测序结果,尤其是可以获得PCR产物的完整序列,但需要进行克隆转化,增加了实验的成本以及工作量。因此,如何选择合适的测序方案以保障测序质量、控制实验成本和提高实验效率,是EST-SSR标记开发和其它PCR产物测序的一个关键问题。
本研究以1株细叶桉(E. tereticornis Smith)为材料,比较了桉树EST-SSR标记开发中PCR产物直接测序和混合克隆测序的效果,以期获得高效、经济的EST-PCR产物测序方案。同时,开发了一批桉树EST-SSR标记,有助于增加桉树的分子标记资源。
1结果与分析
1.1桉树32对EST-SSR引物的PCR扩增
为了在混合克隆后可以通过单菌落PCR产物查找长度对应的EST-PCR产物,并且避免对插入片段相同、菌落PCR产物长度亦同的菌落的重复测序,候选32对桉树EST-SSR引物对1株细叶桉(P2) DNA扩增的产物大小尽量不同,介于120 bp和1 500 bp之间(图1)。PCR产物基本都是单一、明亮的谱带,少量PCR产物具有较弱的杂带(图1中27, 29和31号)。另外,32对EST-SSR引物中,有的PCR产物大小与设计的EST长度相符,有的明显大于设计的EST长度(表1)。
|
图1 桉树32对EST-SSR引物对1株细叶桉(P2)的PCR扩增产物
Figure 1 PCR products amplified from the tested eucalyptus P2 using 32 EST-SSR primer pairs
|
1.2桉树32对EST-SSR引物的PCR产物的直接测序
32个EST-SSR对P2的PCR产物直接测序中,成功测序的有21个(表1),比例为65.6%。其中,4个EST-SSR (03, 08, 20和22号)的简单重复序列邻近测序引物,未能确认完整的重复单位数,因毛细管测序中引物序列后一般有20 50 bp不能测出。成功测序的21个EST-SSR中,P2的2个等位片段间在长度上纯合的标记有13个,杂合的有8个(08, 11, 13, 17, 18, 20, 22和28号),其中18号的杂合性是由于单个碱基重复数的变异所致(表1)。对于长度杂合的SSR标记,PCR产物直接测序峰图中重复单位变异后出现双峰(单峰也是双峰叠加的结果)。图2和图3A分别显示了P2中等位片段长度纯合标记EUCeSSR1135 (06号)和杂合标记EUCeSSR0946 (13号)的重复单位及其与源EST的对比。
|
Figure 2 Partial sequences of the length-homozygous EST-SSR marker EUCeSSR1135 (No. 06) within P2 and their comparison to the original EST
|
|
|
|
|
另外,片段长度纯合的13个标记中,有7个(02, 03, 04, 05, 10, 14和23号)在P2的2个等位片段间存在至少1个单核苷酸多态性(single nucleotide polymorphism, SNP),或称个体内SNP。
1.3 EST-PCR产物的混合克隆测序
在8个、16个、24个和全部32个EST-SSR对细叶桉P2扩增产物的混合克隆测序中,测序成功的标记数分别为6个(75.0%)、11个(68.8%)、11个(45.8%)和14个(43.8%) (表1)。可见,随着混合克隆的EST-PCR产物数量的增加,虽然测序成功的标记数量呈增加的趋势,但比例却逐渐下降。其中,16个PCR产物混合克隆的成功测序的标记数量较大、比例也较高。
不同数量PCR产物混合克隆共成功测序19个标记(表1)。与PCR产物直接测序相比,有6个标记(15、16、26、27、29和30号)只在混合克隆测序中被测到,包括2个片段长度杂合标记(15和29号)和1个长度纯合、含有个体内SNP的标记(26号);但有8个PCR产物直接测序成功的标记(5, 9, 10, 11, 14, 17, 18和19号)在混合克隆后没有被测到。
混合克隆的单菌落测序的峰图比较“干净”(图3B和C),有利于查找SSR的位置和数量;但对于长度杂合的EST-SSR标记,单菌落测序只能测到一个等位片段。本研究中,由于有的杂合标记参加了不同数量的PCR产物混合克隆,刚好不同的等位片段均被测序,如8、13、20、22和29号(表1)。
混合克隆测序中也发现了一些“误扩增”片段,即引物序列同某一个EST,但序列的长度与相应的PCR产物不同、且与源EST的序列一致性很低。该类片段可能为非特异性扩增所致,因长度刚好类似于另一个EST-PCR产物而被测序。
1.4桉树EST-SSR标记的开发
结合对细叶桉P2的PCR产物直接测序和混合克隆测序,共确认了27个桉树EST-SSR的序列,与源EST比对的一致性介于90.23%和100%之间(表1)。其中,在P2中片段长度杂合的标记10个、纯合的17个;在长度纯合的17个标记中,有8个(02, 03, 04, 05, 10, 14, 23和26号)存在1个以上的个体内SNP。
2讨论
目前,新一代的测序技术已有广泛的应用(Mardis, 2008),但传统的末端终止法仍是普遍使用的测序技术(周德贵等, 2008),尤其是针对小规模的目的序列的测序,如EST标记开发中的EST-PCR产物测序。因此,如何经济、快捷和高效地进行末端终止法测序仍然具有极为重要的应用价值。
PCR产物直接测序具有快速、简便和低成本的优点(徐祖元等, 2002; 张晓红等, 2009);并且,对于二倍体的DNA模板,可以同时测出2个等位片段(虽然存在插入/缺失时后面测序峰会出现双峰)。但是,因技术限制,PCR产物直接测序时前面3 040 bp左右往往遗失(苑克俊等, 2007),这不利于完整序列的确认,尤其是目的序列靠近测序引物位置时,如本研究中SSR重复单位未能完整判读的4个标记;并且,当DNA模板的引物结合序列发生变异时,引物特异性较差将导致非特异性PCR产物的产生,测序的峰图较乱,影响测序结果判读的准确性(张晓红等, 2009),这也是本研究中尚有11个(34.4%)扩增较好的EST-SSR标记未能成功测序的主要原因。因此,PCR产物直接测序主要应用于扩增特异性好、测序引物后面的几十个碱基不需完整确认的序列。
克隆测序可以克服PCR产物直接测序的不足,获得完整序列(毛伟华, 2005)。但是,以往的克隆测序多是针对单个PCR产物,这在待测样品较多时需要花费大量的经费和人力进行克隆转化,因此多个PCR产物混合克隆应是一个不错的选择。本研究中混合克隆测序的成功率为43.8~75.0%,并且有6个标记只在混合克隆中被成功测序,证明了混合克隆测序的有效性。
比较8个、16个、24个和32个EST-PCR产物混合克隆后的测序成功率,兼顾克隆转化与测序的效率、成本和时间,我们推荐16个EST-PCR产物混合克隆为最好的选择。混合克隆的PCR产物太少,克隆转化的成本较高、耗时也较多。PCR产物太多,非特异性扩增的谱带数增加(虽然切胶回收法可以解决这一问题,但增加成本和人力),菌落PCR后通过片段长度查找对应的PCR产物时易与长度近似的目的片段混淆,从而造成测序的浪费,如本研究32个EST-PCR产物的混合克隆中32次测序只确认了14个标记;并且,PCR产物增多,产物间长度差别相对较小,菌落PCR后通过琼脂糖凝胶电泳不易准确查找对应长度的EST-PCR产物(尤其对于长片段)。当然,在实际实验中,再挑取更多的菌落测序可以获得更多的标记,但增加了菌落PCR与测序的工作量和成本。
目前,已发表的桉树EST-SSR标记只有20个,且未进行测序确认(Faria et al., 2010)。本研究共开发了与源EST序列一致性好的27个桉树EST-SSR标记(表1),这将显著增加桉树的分子标记资源。并且,27个标记中,在P2中存在片段长度杂合的标记有10个、存在个体内SNP的标记有8个,这些标记将在以P2为父本的遗传图谱构建群体中分离,从而有效用于遗传图谱构建。这也表明在桉树中开发基于EST的分子标记具有极大潜力。
3材料与方法
3.1实验材料与DNA提取
1株细叶桉(P2)是Gan等(2003)遗传图谱构建群体的父本。DNA提取参考CTAB法(Doyle and Doyle, 1991),稍做改进,即在CTAB提取液中加入5%的聚乙烯吡咯烷酮(PVP)和2%的-巯基乙醇。pGEM®-T Easy载体及JM109大肠杆菌感受态细胞购自Promega公司。
3.2 EST序列、引物序列和PCR
桉树EST序列下载自GenBank (http://www.ncbi.nlm.nih.gov/projects/dbEST/),截止2009年1月2日共有35 643条。利用PHRAP软件(http://bozeman.mbt.washington.edu/phrap.docs/phrap.html)拼接后,通过MISA软件(http://pgrc.ipk-gatersleben.de/misa/)查找简单序列重复,标准为:2、3和4碱基重复的最短长度为12 bp,5和6碱基为15 bp。包含SSR的序列用于EST-SSR引物设计,软件为Primer Premier 5.0 (http://www.premierbiosoft.com/primerdesign/index.html),标准为:引物长度18~22 bp (最佳20 bp),Tm 55~65℃ (最佳60℃),GC含量40~60 %,目的片段长度100~500 bp。引物合成委托上海Invitrogen公司完成。
利用细叶桉(P2) DNA进行EST-SSR引物的PCR条件优化,主要针对PCR体系的Mg2+浓度和PCR程序的退火温度。PCR优化后,选择PCR产物长度不同的32个EST-SSR进行测序实验,引物序列和PCR产物大小见表1。PCR体系和PCR程序参考张晓红等(2009),退火温度均为56℃,但10×Buffer中Mg2+浓度改为15或20 mmol/L。
3.3 PCR产物纯化和直接测序
PCR产物经乙醇-醋酸钠法纯化后直接测序(张晓红等, 2009),测序一般利用前向引物,对较长片段也利用后向引物测序。测序采用20 μL体系,参考Bigdye Terminator V3.1操作手册(Applied Biosystems Co.),但测序试剂Bigdye用量为0.5 μL。测序仪为ABI 3130xl,数据收集利用软件Data Collection 2.0,序列查看和分析利用软件Sequencing Analysis 5.2。所测序列与目标EST序列的比对利用软件DNAMAN V5.2.2 (Lynnon Biosoft, Quebec, Canada)。
3.4 PCR产物的混合克隆和测序
对于候选32个桉树EST-SSR标记,按照对细叶桉P2扩增产物长度的相互差别尽量较大的原则,分别选取8个(04, 06, 08, 12, 15, 17, 22和26号)、16个(01, 04, 06, 08, 10, 11, 13, 16, 17, 20, 22, 23, 24, 26, 28和29号)、24个(去除02, 05, 09, 13, 14, 18, 22和32号)以及全部32个标记的PCR产物进行等摩尔混合,并利用载体pGEM®-T Easy (Promega)进行克隆、通过热激法转化JM109感受态细胞,转化细胞在含有X-gal/IPTG的LB固体培养基上进行蓝白斑筛选。操作过程按照试剂盒的说明。
从培养平板上挑取6倍于原PCR产物数量的阳性菌落进行PCR,引物为pUC/M13-F (5'-GTTTTCCCAGTCACGAC-3')和pUC/M13-R (5'-TCACACAGGAAACAGCTATGAC-3')。PCR体系参考张晓红等(2009),但10×Buffer中Mg2+浓度为20 mmol/L;PCR程序为95℃ 5 min,再30个循环:94℃ 30 s、55℃ 30 s、72℃ 3 min,最后72 ℃ 10 min。根据菌落PCR产物大小(减去载体序列253 bp)确定最可能对应的原EST-PCR产物,针对各个最可能插入了原EST-PCR产物的菌落进行测序,即各批测序的克隆数等于混合的标记数量。测序过程参考前3.3。所测序列与目标EST序列的比对利用软件DNAMAN V5.2.2 (Lynnon Biosoft)。
作者贡献
周长品和李发根是本研究的实验设计和实验研究的执行人;翁启杰和于晓丽及李梅参与实验执行以及结果分析;甘四明是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学基金(31070592)、广东省自然科学基金(10151052001000000)、国家高技术研究发展计划(863计划)重点项目(2006AA100109)和中央级公益性科研院所基本科研业务费专项(RITFKYYW2008-4和2010-04)共同资助。
参考文献
Ayers N.M., McClung A.M., Larkin P.D., Bligh H.F.J., Jones C.A., and Park W.D., 1997, Microsatellites and a single nucleotide polymorphism differentiate apparent amylose classes in an extended pedigree of US rice germplasm, Theor. Appl. Genet. 94(6-7): 773-781 doi:10.1007/s001220050477
Brondani R.P.V., Williams E.R., Brondani C., and Grattapaglia D., 2006, A microsatellite-based consensus linkage map for species of Eucalyptus and a novel set of 230 microsatellite markers for the genus, BMC Plant Biol., 6: 20 doi:10.1186/1471-2229-6-20
Ceresini P.C., Silva C.L.S.P., Missio R.F., Souza E.C., Fischer C.N., Guillherme I.R., Gregorio I., da Silva E.H.T., Cicarelli R.M.B., da Silva M.T.A., Garcia J.F., Avelar G.A., Neto L.R.P., Marcon A.R., Junior M.B., and Marini D.C., 2005, Satellypus: analysis and database of microsatellites from ESTs of Eucalyptus. Genet. Mol. Biol., 28(3s): 589-600
Cha R.S., and Thilly W.G., 1995, Specificity, efficiency, and fidelity of PCR. In: Dieffenbach C.W. and Dveksler G.S. (eds.), PCR primer: a laboratory manual, Cold Spring Harbor Press, New York, USA, pp.37-52
Choudhary S., Sethy N.K., Shokeen B., and Bhatia S., 2009, Development of chickpea EST-SSR markers and analysis of allelic variation across related species, Theor. Appl. Genet. 118(3): 591-608 doi:10.1007/s00122-008-0923-z
Doyle J.J., and Doyle J.L., 1991, Isolation of plant DNA from fresh tissue, Focus, 12(1): 13-15
Ellis J.R., and Burke J.M., 2007, EST-SSRs as a resource for population genetic analyses, Heredity, 99(2): 125-132 doi:10.1038/sj.hdy.6801001
Fang X.J., Wu W.R., and Tang J.L., eds., 2002, Molecular marker assistant breeding in crop, Science Press, Beijing, China, pp.10-21 (方宣钧, 吴为人, 唐纪良, 编著, 2002, 作物DNA标记辅助育种, 科学出版社, 中国, 北京, pp.10-21)
FAO, 2000, Global forest resources assessment 2000 – main report, FAO Forestry Paper 140, FAO, Rome, Italy
Faria D.A., Mamani E.M.C., Pappas M.R., Pappas G.J., and Grattapaglia D., 2010, A selected set of EST-derived microsatellites, polymorphic and transferable across 6 species of Eucalyptus. J. Hered., 101(4): 512-520 doi:10.1093/jhered/esq024
Gan S., Shi J., Li M., Wu K., Wu J., and Bai J., 2003, Moderate-density molecular maps of Eucalyptus urophylla S. T. Blake and E. tereticornis Smith genomes based on RAPD markers, Genetica, 118(1): 59-67 doi:10.1023/A:1022966018079
Hill K.D., and Johnson L.A.S., 1995, Systematic studies in the Eucalyptus. 7. A revision of the bloodwoods, genus Corymbia (Myrtarceae), Telopea, 6(2-3): 185-504
Kunkel T.A., and Bebenik K., 2000, DNA replication fidelity, Annu. Rev. Biochem., 69: 497-529 doi:10.1146/annurev.biochem.69.1.497
Li S.X., Zhang X.Y., Wang Y.Y., and Yin T.M., 2010, Content and characteristics of microsatellites detected in expressed sequence tag sequences in Eucalyptus, Zhiwu Xuebao (Chinese Bulletin of Botany), 45(3): 363-371 (李淑娴, 张新叶, 王英亚, 尹佟明, 2010, 桉树EST序列中微卫星含量及相关特征, 植物学报, 45(3): 363-371)
Mao W.H., 2005, Establishment of direct sequencing method with colony PCR products and its application in rice gene sequencing, Zhongguo Shuidao Kexue (Chinese Journal of Rice Science), 19(5): 463-466 (毛伟华, 2005, 菌落PCR产物直接测序方法的建立及在水稻基因测序中的应用, 中国水稻科学, 19(5): 463-466)
Mardis E.R., 2008, Next-generation DNA sequencing methods, Annu. Rev. Genomics Human Genet., 9: 387-402 doi:10.1146/annurev.genom.9.081307.164359
Powell W., Machray G.C., and Provan J., 1996, Polymorphisms revealed by simple sequence repeats, Trends Plant Sci. 1(7): 215-222
Rabello E, de Souza AN, Saito D, Tsai S-M (2005) In silico characterization of microsatellites in Eucalyptus spp.: abundance, length variation and transposon associations. Genet. Mol. Biol., 28(3s): 582-588
Sharma R.K., Bhardwaj P., Negi R., Mohapatra T., and Ahuja P.S., 2009, Identification, characterization and utilization of unigene derived microsatellite markers in tea (Camellia sinensis L.), BMC Plant Biol., 9: 53 doi:10.1186/1471-2229-9-53
Simko I., 2009, Development of EST-SSR markers for the study of population structure in lettuce (Lactuca sativa L.), J. Hered., 100(2): 256-262 doi:10.1093/jhered/esn072
Steane D.A., Jones R.C., and Vaillancourt R.E., 2005, A set of chloroplast microsatellite primers for Eucalyptus (Myrtaceae), Mol. Ecol. Notes, 5(3): 538-541 doi:10.1111/j.1471-8286.2005.00981.x
Tauz D., 1989, Hypervariability of simple sequences as a general source for polymorphic DNA markers, Nucleic Acids Res. 17(16): 6463-6471 doi:10.1093/nar/17.16.6463
Varshney R.K., Graner A., and Sorrells M.E., 2005, Genic microsatellite markers in plants: features and applications. Trends Biotechnol., 23(1): 48-55 doi:10.1016/j.tibtech.2004.11.005
Xu Z.Y., Bao Q.Y., and Niu Y.X., 2002, Factors that influence direct sequencing of PCR products, Yichuan (Hereditas), 24(5): 548-550 (徐祖元, 包其郁, 牛宇欣, 2002, PCR产物直接测序技术中影响因素的研究, 遗传, 24(5): 548-550)
Yuan K.J., Liu Q.Z., Ai C.X., and Wang J.H., 2007, A method for isolation of genomic DNA in fruit tree and sequencing its PCR product, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 5(6s): 173-176 (苑克俊, 刘庆忠, 艾呈祥, 王绛辉, 2007, 果树基因组DNA的提取和PCR扩增测序方法, 分子植物育种, 5(6s): 173-176)
Zhang X.H., Li F.G., Wang Y., Xu L.A., Li M., and Gan S.M., 2009, An optimized protocol for sequencing EST-PCR products in Eucalyptus, Jiyinzuxue Yu Yingyongshengwuxue (Genomics and Applied Biology), 28(3): 535-543 (张晓红, 李发根, 王宇, 徐立安, 李梅, 甘四明, 2009, 桉树EST-PCR产物测序方案的优化, 基因组学与应用生物学, 28(3): 535-543)
Zhou D.G., Zhao Q.Y., Fu C.Y., Li H., Cai X.F., Luo D., and Zhou S.C., 2008, The next generation sequencing and its effect on the rice molecular design breeding, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 6(4): 619-630 (周德贵, 赵琼一, 付崇允, 李宏, 蔡学飞, 罗达, 周少川, 2008, 新一代测序技术及其对水稻分子设计育种的影响, 分子植物育种, 6(4): 619-630)
.png)