2.上海中科新生命生物科技有限公司, 上海, 200233
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2010 年, 第 8 卷, 第 5 篇 doi: 10.5376/mpb.cn.2010.08.0005
收稿日期: 2010年06月22日 接受日期: 2010年09月12日 发表日期: 2010年10月08日
宋浩等, 2010, 烟草青枯菌分泌蛋白shotgun蛋白质组学研究, 分子植物育种 Vol.8 No.5 (doi: 10.5376/mpb.cn.2010.08.0005)
烟草青枯病是由茄科劳尔氏菌(Ralstonia solanacearum)引起的一种毁灭性的烟草种植灾害,可造成烟草的巨大减产。本实验首次采用shotgun蛋白质组学技术对致病性烟草青枯菌菌株的分泌蛋白进行系统分析,结果鉴定得到了384个蛋白质。按照青枯菌的致病特点和侵染过程将这384个蛋白分为糖蛋白、信号肽蛋白和跨膜蛋白等7个功能类群。其中,17个被前人研究报道为与青枯菌致病相关的蛋白质,表明使用shotgun蛋白质组学技术可有效的分析青枯菌分泌蛋白,具有可行性和有效性。327个迄今为止尚无文献报道其与青枯菌致病性相关,40个为未知蛋白。这些研究结果为进一步揭示青枯菌对烟草的致病机理及抗病基因的功能克隆提供了依据。
烟草青枯菌(Ralstonia solanacearum)是在世界范围内危害烟草生产,引起烟草青枯病的主要致病菌,致使每年烟草种植的大量减产。该病菌还会危害其它诸如土豆,番茄等世界主要经济作物(李林章等, 2005)。因此,明确青枯菌的致病机理,阐明烟草的抗病基因功能,对高产栽培调控和抗病分子育种都有十分重要的意义。到目前为止,国内外已经从基因定位和分子标记辅助育种(扬友才等, 2006; Jiang et al, 2007)、基因组序列比对(Remenant et al., 2010; Fujiwara et al., 2008; Guidot et al., 2007)、转录组学(Guidot et al., 2009)、蛋白质组学(宋浩等, 2010; Dahal et al., 2010)以及抗性基因转化(Zhang et al., 2009; Huang et al., 2007)等多个方向对青枯病的致病机理开展了大量深入的研究。这些研究表明青枯菌之所以能够引起多种植物的致死性枯萎,主要是通过根、茎和伤口或次生根的根冠进入植物体后,分泌大量胞外蛋白和多糖侵染寄主的木质部,然后在整个维管束中蔓延,引起严重的萎焉最终导致植物死亡(Denny et al., 1990; Poueymiro and Genin, 2009)。同时发现青枯菌分泌蛋白多为病原微生物与植物受体蛋白起作用的激发子和其它致病因子(Kang et al., 1994; 宋浩等, 2010),所以分析这些分泌蛋白的组成和功能对于人们认识烟草青枯菌的致病机理和建立有效地防治手段有着重要的意义。目前青枯菌(GM1000菌株)全基因组测序的完成(Salanoubat et al., 2002),使得从蛋白质组水平分析和研究该菌的致病机制成为可能。近年来,以不同青枯菌菌株以及宿主为研究对象的比较蛋白质组学研究已取得了一些新的进展(Dahal et al., 2009; Kang et al., 2008; Monchy et al., 2006; 宋浩等, 2010),但尚未见有关利用蛋白质组学方法研究青枯菌分泌蛋白的报道。随着蛋白质组学的日益发展,利用现代质谱技术可以快速,有效,高通量地鉴定生物样本的蛋白质。而蛋白质组学中常用的质谱shotgun (鸟枪法)技术可以通过对复杂的蛋白质样品进行酶解形成肽段,经过广泛的分级后,由串联质谱进行鉴定(夏其昌和曾嵘, 科学出版社, pp.278), shotgun蛋白质组学技术已经成为研究复杂蛋白质混合物的有力工具(Wu and MacCoss, 2002)。本实验首次采用shotgun蛋白质组学技术对致病性青枯菌分泌蛋白进行系统研究,为进一步阐述烟草青枯菌的致病机理提供了实验数据和理论基础。
1结果与分析
1.1菌体的培养
在进行青枯菌菌体扩培前,参考陈晓敏等的方法首先使用2,3,5-三苯基氯化四氮唑培养(TTC)琼脂平板培养基从表型上验证其致病性,平行接种的青枯菌菌体于TTC琼脂平板上30℃培养(陈晓敏等, 2000),48 h后培养结果如图1显示,致病性菌株呈现典型的周围光亮白边中心红点特征,结果与文献报道的一致(李文溶和段遒雄, 1988) ,说明本实验所用菌体样品符合要求。
1.2分泌蛋白质提取以及SDS-PAGE结果
致病性青枯菌接种于分泌培养基中,收集培养基并用Amicon Ultra-15 (截留分子量5 K)超滤装置浓缩超滤含有分泌蛋白的培养基后定量,结果在200 mL的分泌培养基中,致病青枯菌分泌了多达50 µg的蛋白质。致病性青枯菌培养基蛋白质SDS-PAGE电泳结果如图2所示:致病性青枯菌分泌蛋白大部分集中在27 KDa与97 KDa之间,在27 KDa左右有高丰度蛋白的存在。根据SDS-PAGE电泳蛋白条带的分布,我们将其分为5个条带组进行后续的酶解。
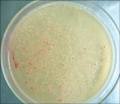 图1 TTC琼脂平板培养基上的致病青枯菌 Figure 1 The pathogenic Ralstonia solanacearum on the TTC agar medium |
 图2 致病性青枯菌分泌蛋白质SDS-PAGE结果 Figure 2 The SDS-PAGE result of the pathogenic Ralstonia solanacearum srcretory proteins |
1.3 LC-MS/MS 鉴定结果
致病青枯菌分泌蛋白质样品经SDS-PAGE分级后,酶解产物使用LC/MS/MS鉴定,鉴定控制假阳性率为P<0.05%, 并且控制至少含有两个以上唯一肽段的蛋白为可信蛋白,经过这些严格筛选,最后得到384个蛋白质(其中69537个MS/MS 色谱图, 得到2314个肽段, 鉴定到485个唯一肽段),鉴定结果见附表1。按照青枯菌分泌蛋白的输出和传递过程以及分泌蛋白的致病特点(Chun-Mei et al., 2002),我们将这384个蛋白质分为以下7大类:糖蛋白(3%)、信号蛋白(6%)、跨膜蛋白(6%)鞭毛蛋白(6%)、效应蛋白(2%)、毒性蛋白(4%)和其他蛋白(73%)。
1.3.1糖蛋白
青枯菌在侵染宿主过程中首先分泌大量胞外多糖,胞外多糖影响和阻碍植物体内的水分运输,特别是易于对叶柄结和小叶处较小孔径的导管穿孔板造成堵塞,因而引起植株萎蔫(Denny et al., 1990)。在我们的结果中,发现有3%的蛋白(11个)与糖代谢过程相关。其中胞外多糖分泌蛋白P58597已被证实为青枯菌致病性相关蛋白(Orgambide et al., 1991)。同时我们首次发现了另外5个胞外多糖分泌蛋白A3RX63/Q8XT40/Q8XTU8/Q8XX75/Q8XQI5 (附表1)。在青枯菌分泌的与糖代谢相关蛋白中,除了胞外多糖分泌蛋白外,还有一大部分为具有很强分解能力的糖蛋白,它们主要参与对宿主植株表层细胞壁的分解作用,这样使得病原菌可以成功侵入植株,到达质外体,通过向胞外空间甚至植物细胞内释放效应蛋白(effector proteins) (Dangl and Jones, 2001; Hammond-Kosack and Parker, 2003)。在我们的结果中也发现了5个类似的具有分解能力的糖蛋白:溶果胶酶P58601、多聚半乳糖醛酸酶Q8XRJ8、PheB多聚半乳糖醛酸酶Q53241和类枯草杆菌素蛋白酶A3RTC4和B2UBR3。其中类枯草杆菌素蛋白酶A3RTC4和 B2UBR3蛋白属于丝氨酸蛋白酶家族,在稻瘟病菌,烟曲霉等一些病原菌致病过程中的作用已有报道,具有很强的分解细胞壁功能(魏艺聪等, 2009)。
1.3.2信号蛋白和跨膜蛋白
在青枯菌成功侵入宿主质外体以后,通过精巧的蛋白传输系统, 产生各种与致病性相关的效应蛋白 (effector proteins)和毒性蛋白(Koster et al., 2000)。这些效应蛋白和毒性蛋白在传递之前必须被青枯菌膜蛋白上的信号肽(signal peptide)识别以后才能进入正常的分泌途径。信号肽(signal peptide)是跨膜蛋白起始密码子后,有一段编码疏水氨基酸序列的RNA片段,该片段所编码的肽段。新合成的蛋白质通过该信号肽的识别进入到正常的分泌途径。所以信号肽和跨膜蛋白在病原和寄主之间进行信息传递发挥,致病和诱导寄主的抗病反应中着重要的作用(Huang et al., 2007)。在我们得到的384个青枯菌分泌蛋白中,发现有12%的蛋白(47个)属于信号蛋白或者跨膜蛋白。其中有23个信号肽蛋白(A7CK23/A7CLD6/A7CM55/B2UDH0/Q8XPI6/Q8XQJ7/Q8XQM1/
Q8XR59/Q8XRC9/Q8XRS3/Q8XS79/Q8XUC7/Q8XV98/Q8XUU4/Q8XVH6/Q8XVV9/Q8XW22/Q8XX33/Q8XYK2/
Q8Y0Z1/Q8Y0Z3/Q8Y1X9/Q8Y382)和24个跨膜蛋白以及跨膜相关蛋白Q8XRS6/Q8XSE4/Q8XT54/Q8XTF1/Q8XTX0/
Q8XTY6/Q8XWU0/Q8Y119/Q8Y175/Q8Y1N4/Q8Y2A7/Q8Y2I4/A7C9Y2/A7CG74/A7CF52/B2U9G4/B2UFJ2/
B5RXC1/B5SER5/B5RXI8/B5S060/B5SKP6/Q8XR13/Q8XR28) (附表1)。
1.3.3鞭毛蛋白
细菌的分泌蛋白经过膜蛋白上的信号肽识别后进入分泌系统。细菌的分泌系统是转移所分泌蛋白和其它分子最重要的系统,主要有4种类型的分泌系统(Genin, 2002),其中Ⅱ型分泌系统(T2SS)和Ⅲ型分泌系统(T3SS)对青枯菌的致病性贡献最大(Genin and Boucher, 2004)。细菌的分泌蛋白在通过分泌系统进行分泌过程中,细菌的附属物鞭毛(flagellin)起到了不可或缺的媒介作用(Chun-Mei et al., 2002)。鞭毛蛋白(flagellin protein) 作为病原细菌毒力因子鞭毛的组成成分,参到分泌系统通路,同时它的运动性也是形成细菌侵染宿主的主要因素之一(Orans et al., 2010)。在我们的研究结果中,发现了6%的青枯菌分泌蛋白(23个)为鞭毛蛋白:鞭毛蛋白B5SA46/ Q8XST5、鞭毛蛋白片段Q0Q2Y4/Q0Q2Y5/Q0Q2Y8/Q0Q2Z3/Q0Q2Z4/ Q0Q2Z6/
Q0Q2Z8/Q0Q318/Q0Q320/Q0Q3/Q0Q326/Q83UT8/Q847Y78、L-环鞭毛蛋白B2UJW8/A7C9I6/ A3RUG8、菌毛转运蛋白B5RW01/B5SHR2/ Q8XVZ3和4型菌毛蛋白B5SGQ4。
1.3.4效应蛋白
通过传输系统的分泌和传递作用,细菌的效应蛋白(effector protein)便成功注入宿主细胞内。这些效应蛋白表现出多种毒性效应,包括促使宿主细胞骨架的改变,破坏信号转导途径和抑制凋亡,并在宿主细胞建立适于寄生的亚细胞环境等(Mota and Cornelis, 2005)。细菌正是通过这些效应蛋白的毒性作用来干扰植物正常的生理状态,抑制植物的防御反应,促进自身的大量增殖(曹鹏等, 2009)。在384个青枯菌分泌蛋白中,我们得到了8个效应蛋白,其中Lrr-gala家族3型分泌蛋白B5S4F6(Mukaihara and Tamura, 2009)和popf1分泌蛋白Q8XPT2 (Meyer et al., 2006)已经被证实是青枯菌致病性效应蛋白。同时我们也首次得到了另外6个效应蛋白:B7ZJG6/C0SPP3/Q8XRC8/Q8XZK9/C0SPN3/A3RZU5 (附表1)。
1.3.5毒性蛋白
除了以上提到的通过调节宿主细胞的代谢过程关键酶的活性来实现致病过程的效应蛋白外(Mota and Cornelis, 2005),我们在384个青枯菌分泌蛋白中发现了15个直接进行毒性作用的毒性蛋白。文献报道蛋白PopA(Q84IE8和Q9RBS0)可以明显增强青枯菌的致病性,随后的实验发现在PopA基因中插入特殊片段后可以明显抑制疾病形成,致使宿主的青枯现象明显改善(Kanda et al., 2008)。蛋白B5SAX2 (rela/spot domain protein)被发现在细菌侵入宿主和生长的过程中一直保持着生物活性(Sun et al., 2009)。外膜蛋白OmpW (A7CFB0和B2UFP6)和外膜蛋白OmpA/MotB (A7CM53和B2U879)被证实主要是与宿主的受体分子进行相互作用(Sun et al., 2009)。蛋白PilA (A3RQ41)和PilT (A3RTF1)已经被很明确的提到属于青枯菌的毒性蛋白(Kang et al., 2002)。蛋白FtsN (A3RWZ4)和异青霉素N差向异构酶(A3RQV4)有文献报道主要参与宿主的细胞分裂等过程,但是否与青枯菌的致病性相关还需进一步验证(Derouaux et al., 2008)。血溶素样蛋白Q8XT20/Q8Y2T5/Q8Y377/Q8Y378 (hemolysin-type protein)据报道主要参与大肠杆菌的致病过程(Blum et al., 1995)。
2讨论
烟草青枯病是由青枯菌(Ralstonia solanacearum)引起的一种毁灭性灾害,而青枯菌的胞外分泌蛋白多为致病过程中的关键因子(Kang et al., 1994; 宋浩等, 2010)。通过对分泌蛋白的检测与分析,可能对阐明分泌蛋白在青枯菌致病过程中的作用机制有较大帮助。目前与青枯菌分泌蛋白相关的蛋白质组学研究还没有报道,本研究是在这一领域进行的初步探索。
Shotgun技术是近年来发展迅速的蛋白质组学方法(Gavin et al., 2002),较传统的2D-MS (双向电泳-质谱)蛋白质组学策略具有高通量、易操作性、快速性以及很好的普适性等优点(Blonder et al., 2002; Janini et al., 2003)。通过shotgun蛋白质组学的应用,我们得到了384个具有高可信度的致病性烟草青枯菌菌株分泌蛋白,这为烟草青枯菌致病性研究积累了重要资料。但是,shotgun蛋白质组学技术本身也有一些不足之处,比如蛋白质直接酶切得到的肽段混合物过于复杂,较难实现对细胞全部蛋白质的识别和鉴定等(Matallana-Surget et al., 2010),所以,在青枯菌分泌蛋白的提取、分离纯化及分析鉴定等各个环节还有待进一步优化。
按照青枯菌侵染进程和致病特点(Chun-Mei et al., 2002),将这384个蛋白从糖蛋白、信号肽蛋白和跨膜蛋白等七个方面进行了分类,得到致病性青枯菌菌株分泌蛋白组分的详细信息。这其中17个蛋白质前人研究报道可能与青枯菌的致病性相关,其中包括草杆菌素蛋白酶A3RTC4和 B2UBR3两种(魏艺聪等, 2009)、Lrr-gala家族3型分泌蛋白B5S4F6 (Mukaihara and Tamura, 2009)、popf1分泌蛋白Q8XPT2 (Meyer et al., 2006)、蛋白PopA (Kanda et al., 2008)、蛋白B5SAX2 (Sun et al., 2009)、外膜蛋白OmpW和OmpA/MotB四种(Sun et al., 2009)、蛋白PilA和PilT (Kang et al., 2002)、蛋白FtsN、异青霉素N 差向异构酶(Derouaux et al., 2008)、血溶素样蛋白Q8XT20/Q8Y2T5/Q8Y377/Q8Y378三种(Blum et al., 1995)。这些结果说明使用shotgun蛋白质组学技术可有效的分析青枯菌分泌蛋白,具有可行性和有效性。同时在这384个蛋白中,有327个迄今为止尚无文献报道与青枯菌的致病性相关,有40个为未知蛋白。这个结果一方面的原因可能是因为目前关于青枯菌分泌蛋白的研究较少;另一方面是由于shotgun蛋白质组学技术较其他的传统蛋白检测手段例如免疫杂交等或者传统的基于凝胶系统的2D技术具有更高的检测水平,可以检测到更多的低丰度蛋白质(Listgarten and Emili , 2005)。这些首次鉴定到的蛋白和未知蛋白都为烟草青枯菌致病性研究及烟草青枯菌蛋白组数据库的建立积累了重要资料,为进一步阐述烟草青枯菌的致病机理提供了实验数据和理论基础,这也是我们下一步研究工作的重点。
3材料与方法
3.1菌种培养
供试菌株为茄青枯雷尔氏菌(Ralstonia solanacearum)。根据细菌的生理生化特性,属于为Race1 (Biovar 3)品种,来自中国农科院。本供试菌株为杆状,革兰氏染色阴性,参考陈晓敏的方法在TTC平板上30℃下划线培养(陈晓敏等, 2000)。48 h后,可出现两种不同的菌落:一种是中心呈粉红色或浅色的稀液状、较宽的白边、不规则形或近圆形、流动性强、致病力强的野生菌落;另一种是致病力弱或丧失致病力的变异菌落,它们呈圆形、光滑、干燥、较扁平、玫瑰红色、白边很窄(李文溶和段遒雄, 1988)。挑取致病菌菌株(virulent strain)继续培养。
3.2样品制备
致病性青枯菌分别接种于分泌培养基(10克酸水解酪素, 蒸馏水1 000 mL, PH 7.0~7.46) (陈晓敏等, 2000)中,30℃振荡培养(200转/min) 48小时,当培养液OD600nm=0.6时将培养液以6000转/min离心,收集上清。上清用Amicon Ultra-15(截留分子量5 K)超滤装置浓缩至1 mL后加入5 mL预冷的丙酮(含1%二硫苏糖醇)沉淀,沉淀产物冷冻干燥后加入含8 M尿素,150 mM甲基氨基甲烷,蛋白酶抑制剂的裂解液(pH 8.5),4℃放置30 min后以12 000转/min于4℃离心2 h,取上清,上清用GE-2D除盐试剂盒(GE)除盐后以Bradford法定量(Bio-Rad)。
3.3 SDS-聚丙烯酰胺凝胶电泳以及酶解
致病菌株提取的分泌蛋白样品加入二硫苏糖醇至终浓度10 mmol/L, 放置于37℃ 2.5 h后加入碘乙酰胺至终浓度50 mmol/L,室温避光反应40 min。还原和烷基化反应后的样品进行SDS-PAGE垂直电泳(12.5T%),每个泳道各上样20 µg。电泳条件为:15 mA电泳20 min后以30 mA电流直至溴酚蓝前沿迁移至底端。SDS-PAGE胶经考马斯亮蓝R-350 (GE)染色后每组样品分成5份,每份条带首先用50 mmol/L碳酸氢胺/30%乙腈脱色,再用100%乙腈处理5 min,干燥后加入50 mmol/L碳酸氢胺,10 min后弃去,再加入100%乙腈,5 min后弃去,干燥后加入以蛋白:酶=50:1的比例加入胰蛋白酶(Promega Biotech Co., Madison, WI, USA),37℃ 反应12 h。酶解产物用0.1% TFA/60%乙腈超声抽提后冷冻干燥,酶解样品于-70℃保存待用。
3.4 LC-MS/MS
酶解样品复溶于0.1%甲酸后用MDLC进行反相C18-HPLC (150×100 mm Column technology Inc., Fremont, CA)分离,泵流速65微升/分钟。经由分流器,柱出口流速为2 µL/min。流动相A为0.1%甲酸/水,流动相B为84%乙腈/0.1%甲酸/水。梯度设置如下:4%~50% B 105 min,100% B维持15 min。RP-HPLC柱出口经由电喷雾接口(ESI)与LTQ线形离子阱质谱相连,具体设置如下:加热毛细管温度为160℃,ESI needle 电压为3.0 kV,碰撞能量为35.0%。使用AGC模式采集信号,毛细管与四级杆等参数由调谐程序自动优化,采集方式为1个全扫描(profile模式)后对全扫描的5个相对最强离子进行MS/MS扫描,其中有动态排除设置(重复2次, 重复间隔0.5 min, 排除间隔1.5 min)。
3.5 LTQ结果分析
LTQ原始文件经由BioWorks 3.0软件组自带的Sequest Cluster进行查库分析,数据库来自swissprot,查库参数简要为母离子宽容度3 Da,碎片离子宽容度为0.8 Da, 固定修饰设置半胱氨酸(+57.02 Da),动态修饰设置甲硫氨酸氧化(+57.02 Da),胰蛋白酶最多允许两个内部酶切位点。利用BioWorks生成Ralstonia正反库,筛选查库结果假阳性率为0.05%,并且含有两个以上唯一肽段的蛋白,去除冗余数据后进行统计学分析。
作者贡献
宋浩、丁伟、沙伟、刘翀、周燕是本研究的实验设计和实验研究的执行人;丁伟和宋浩及沙伟完成数据分析,论文初稿的写作;宋浩和丁伟参与实验设计,试验结果分析;陈薇是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究受湖南中烟工业公司科技计划项目的资助。作者感谢中国农科院为本实验过程中提供实验菌株。感谢两位匿名的同行评审人的评审建议和修改建议。本文中提到了我们实验中涉及的有关试剂供应商和测序服务商,这并非我们为这些试剂供应商和测序服务商的产品和服务提供推荐或背书。
参考文献
Chen X.M., Hu F.P., and Wu Y.R., 2000, Pathotypes and biotypes of Ralstonia solanacearum isolated from bacterial wilt of peanut in Fujian Provinece, Fujian Nongye Daxue Xuebao (Journal of Fujian Agricultural University), 29(4): 470-473(陈晓敏, 胡方平, 吴燕榕, 2000, 福建省花生青枯病菌致病型及生物型的测定, 福建农业大学学报, 29 (4) : 470-473))
Chun-Mei L., Ian B., John M.,Conrad S., Tristan B., Martin R., and Suvi T., 2002, The Hrp pilus of Pseudomonas syringae elongatesfrom its tip and acts as a conduit for translocation of the effector protein HrpZ, EMBO, 21(8): 1909-1915)
Dahal D., Pich A., Braun H.P., and Wydra K., 2010, Analysis of cell wall proteins regulated in stem of susceptible and resistant tomato species after inoculation with Ralstonia solanacearum: a proteomic approach, Plant Mol Biol, 73(6): 643-658 doi:10.1007/s11103-010-9646-z
Dangl J.L., and Jones J.D., 2001, Plant pathogens and integrated defence responses to infection, nature, 411: 826-833
Denny T.P., Carney B.F., and Schell M.A., 1990, Inactivation of multiple virulence genes reduces the ability of Pseudomonas solanacearum to cause wilt symptoms, Mol Plant-Microbe Interact, 3: 293-300
Derouaux A., Wolf B., Fraipont C., Breukink E., Nguyen-Distèche M., and Terrak M., 2008, The monofunctional glycosyltransferase of Escherichia coli localizes to the cell division site and interacts with penicillin-binding protein 3, FtsW, and Fts N, J. Bacteriol, 190(5): 1831-1834 doi:10.1128/JB.01377-07
Fujiwara A., Kawasaki T., Usami S., Fujie M., and Yamada T., 2008, Genomic characterization of Ralstonia solanacearum phage phiRSA1 and its related prophage (phiRSX) in strain GMI1000, J. Bacteriol, 190(1): 143-156 doi:10.1128/JB.01158-07
Genin S., and Boucher C., 2002, Ralstonia solanacearum: secrets of a major pathogen unveiled by analysis of its Genome, Mol. Plant Pathol., 3: 111-318 doi:10.1046/j.1364-3703.2002.00102.x
Genin S., 2002, Ralstonia solanacearum: Secrets of a major pathogen unveiled by analysis of its genome, Molecular Plant Pathology, 3: 111-118 doi:10.1046/j.1364-3703.2002.00102.x
Guidot A., Coupat B., Fall S., Prior P., and Bertolla F., 2009, Horizontal gene transfer between Ralstonia solanacearum strains detected by comparative genomic hybridization on microarrays, ISME J., 3(5): 549-562 doi:10.1038/ismej.2009.14
Guidot A., Prior P., Schoenfeld J., Carrère S., Genin S., and Boucher C., 2007, Genomic structure and phylogeny of the plant pathogen Ralstonia solanacearum inferred from gene distribution analysis, J. Bacteriol, 189(2): 377-387 doi:10.1128/JB.00999-06
Hammond-Kosack K.E., and Parker J.E., 2003, Deciphering plant-pathogen communication: fresh perspectives for molecular resistance breeding, 14(2): 177-193
Huang H.E., Liu C.A., Lee M.J., Kuo C.G., Chen H.M., Ger M.J., Tsai Y.C., Chen Y.R., Lin M.K., and Feng T.Y., 2007, Resistance enhancement of transgenic tomato to bacterial pathogens by the heterologous expression of sweet pepper ferredoxin-I protein, Phytopathology, 97(8): 900-906 doi:10.1094/PHYTO-97-8-0900
Huang J.L., Wu J.Z., Xiao C.G., Li C.J., and Wang G.X., 2007, Analysis of signal peptides of the secreted proteins in Ralstonia solanacearum GMI1000, Yi Chuan, 29(11): 1409-1416 doi:10.1360/yc-007-1409
Jiang H., Liao B., Ren X., Lei Y., Mace E., Fu T., and Crouch J.H., 2007, Comparative assessment of genetic diversity of peanut (Arachis hypogaea L.) genotypes with various levels of resistance to bacterial wilt through SSR and AFLP analyses, J. Genet Genomics, 34(6): 544-554 doi:10.1016/S1673-8527(07)60060-5
Kanda A., Ohnishi K., Kiba A., and Hikichi Y., 2008, Implication of C-terminal mutation of PopA of Ralstonia solanacearum strain OE1-1 in suppression of bacterial wilt, Plant Pathology, 58(1): 159-169 doi:10.1111/j.1365-3059.2008.01938.x
Kang Y., Huang J., and Mao G., et al. Dramatically reduced virulence of mutants of R. solanacearum defective in export of extracellular proteins across the outer membrane, Mol. Plant Microbe Interact., 1994, 7: 370-377
Kang Y., Kim J., Kim S., Kim H., Lim J.Y., Kim M., Kwak J., Moon J.S., and Hwang I., 2008, Proteomic analysis of the proteins regulated by HrpB from the plant pathogenic bacterium Burkholderia glumae, Proteomics, 8(1): 106-21 doi:10.1002/pmic.200700244
Kang Y., Liu H., Genin S., Schell M.A., and Denny T.P., 2002, Ralstonia solanacearum requires type 4 pili to adhere to multiple surfaces and for natural transformation and virulence, Mol. Microbiol, 46(2): 427-37 doi:10.1046/j.1365-2958.2002.03187.x
Koster M., Bitter W., and Tommassen J., 2000, Protein secretion mechanisms in gram-negative bacteria, International Journal of Medical Microbiology, 290: 325-331
Li L.Z., Xie C.H., and Liu J., 2005, Molecular Biology and Pathogenicity of Ralstonia solanacearum, Zhongguo Malingshu(Chinese Potato Journal), 19(5): 290-294(李林章, 谢从华, 柳俊, 2005, 茄科雷尔氏菌(Ralstonia solanacearum)分子生物学基础及其致病机制, 中国马铃薯, 19(5): 290-294)
Listgarten J., and Emili A., 2005, Statistical and computational methods for comparative proteomic profiling using liquid chromatography-tandem mass spectrometry, Mol Cell Proteomics, 4(4): 419-34 doi:10.1074/mcp.R500005-MCP200
Li W.R., and Duan Q.X., 1988, Study of pathogenicity of Ralstonia solanacearum to peanut, Zhongguo Youliao (China Oil), 4:(1-4) (李文溶, 段遒雄, 1988, 花生青枯菌致病性的研究, 中国油料, (4) : 1-4)
Matallana-Surget S., Leroy B., and Wattiez R., 2010, Shotgun proteomics: concept, key points and data mining, Expert Rev. Proteomics, 7(1): 5-7 doi:10.1586/epr.09.101
Meyer D., Cunnac S., Guéneron M., Declercq C., Van Gijsegem F., Lauber E., Boucher C., and Arlat M., 2006, PopF1 and PopF2, two proteins secreted by the type III protein secretion system of Ralstonia solanacearum, are translocators belonging to the HrpF/NopX family, J. Bacteriol, 188(13): 4903-4917 doi:10.1128/JB.00180-06
Monchy S., Benotmane M.A., Wattiez R., van Aelst S., Auquier V., Borremans B., Mergeay M., Taghavi S., van der Lelie D., and Vallaeys T., 2006, Transcriptomic and proteomic analyses of the pMOL30-encoded copper resistance in Cupriavidus metallidurans strain CH34, Microbiology, 52(Pt 6): 1765-76 doi:10.1099/mic.0.28593-0
Mota L.J., and Cornelis., 2005, The bacterial injection kit:type III secretionsystems, Ann Med., 37(4): 234-249 doi:10.1080/07853890510037329
Mukaihara T., Tamura N., 2009, Identification of novel Ralstonia solanacearum type III effector proteins through translocation analysis of hrpB-regulated gene products, Microbiology, 155(Pt7): 2235-2244 doi:10.1099/mic.0.027763-0
Orans J., Johnson M.D., Coggan K.A., Sperlazza J.R., Heiniger R.W., Wolfgang M.C., and Redinbo M.R., 2010, Crystal structure analysis reveals Pseudomonas PilY1 as an essential calcium-dependent regulator of bacterial surface motility, Proc. Natl. Acad. Sci. U S A, 107(3): 1065-1070 doi:10.1073/pnas.0911616107
Orgambide G, Montrozier H, Servin P, Roussel J, Trigalet-Demery D, and Trigalet A, 1991, High heterogeneity of the exopolysaccharides of Pseudomonas solanacearum strain GMI 1000 and the complete structure of the major polysaccharide, J Biol Chem, 266(13): 8312-8321
Poueymiro M., Genin S.,2009,Secreted proteins from Ralstonia solanacearum: a hundred tricks to kill a plant, Curr. Opin. Microbiol, 12(1): 44-52 doi:10.1016/j.mib.2008.11.008
Remenant B., Coupat-Goutaland B., Guidot A., Cellier G., Wicker E., Allen C., Fegan M., Pruvost O., Elbaz M., Calteau A., Salvignol G., Mornico D., Mangenot S., Barbe V., and Médigue C., 2010, Prior P.,Genomes of three tomato pathogens within the Ralstonia solanacearum species complex reveal significant evolutionary divergence, BMC Genomics, 11: 379 doi:10.1186/1471-2164-11-379
Salanoubat M, Genin S, Artiguenave F, Gouzy J, Mangenot S, Arlat M, Billault A, Brottier P, Camus JC, Cattolico L, Chandler M, Choisne N, Claudel-Renard C, Cunnac S, Demange N, Gaspin C, Lavie M, Moisan A, Robert C, Saurin W, Schiex T, Siguier P, Thébault P, Whalen M, Wincker P, Levy M, Weissenbach J, and Boucher CA, 2002, Genome sequence of the plant pathogen Ralstonia solanacearum, Nature, 415: 497-502 doi:10.1038/415497a
Song H., Sha W., Ding W., Liu C., Zhou Y., and Chen W., 2010, The Application of Laber-free Quantification Proteomics in Tobacco Ralstonia solanacearum Pathogenicity Research, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 8(3): 1-10 (宋浩, 沙伟, 丁伟, 刘翀, 周燕, 陈薇, 2010, 非标记定量蛋白质组学在烟草青枯菌致病性研究中的应用, 分子植物育种, 8(3): 1-10)
Sun W., Roland K.L., Branger C.G., Kuang X., Curtiss R., 2009, The role of relA and spoT in Yersinia pestis KIM5 pathogenicity, PLoS One, 4(8): e6720 doi:10.1371/journal.pone.0006720
Wei Y.C., Wang Z.H., and Lu G.D., 2009, Bioinformatics Analysis of Sublitases in M. grisea and Secretion Detection of MGG_07965.6, Zhongguo Nongxue Tongbao (CHINESE AGRICULTURAL SCIENCE BULLETIN), 25(24): 50-55 (魏艺聪, 王宗华, 鲁国东, 2009, 稻瘟病菌Subtilases家族生物信息学分析及其中-蛋白的分泌性检验, 中国农学通报, 25(24): 50-55
Xia Q.C., and Zeng R., eds., 2004, Protein Chemistry and Proteomics, Science Press, Beijing, China, pp. 278 (夏其昌, 曾嵘, 编著, 2004, 蛋白质化学和蛋白质组学, 科学出版社, 中国, 北京, pp.278)
Yang Y.C., Zhou Q.M., and Zhu L.S., 2006, Heredity and RAPD markers analysis of resistance gene to tobacco bacterial wilt, Zhongguo Yancao Xuebao (Chinese Tobacco Science), 12(02): 38-42 (杨友才, 周清明, 朱列书, 2006, 烟草青枯病抗性基因的遗传分析及RAPD标记, 中国烟草学报, 12(02): 38-42)
Zhang G., Chen M., Li L., Xu Z., Chen X., Guo J., and Ma Y., 2009, Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco,J. Exp. Bot., 60(13): 3781-3796 doi:10.1093/jxb/erp214
Blum G., Falbo V., Caprioli A., and Hacker J., 1995, Gene clusters encoding the cytotoxic necrotizing factor type 1, Prs-fimbriae and alpha-hemolysin form the pathogenicity island II of the uropathogenic Escherichia coli strain J96, FEMS Microbiol Lett, 126(2): 189-195