 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 64 篇 doi: 10.5376/mpb.cn.2011.09.0064
收稿日期: 2011年03月12日 接受日期: 2011年04月28日 发表日期: 2011年05月27日
李春艳等, 2011, 小麦淀粉合成酶基因家族的生物信息学分析, 分子植物育种 Vol.9 No.64 (doi: 10.5376/mpb.cn.2011.09.0064)
为了进一步研究小麦淀粉生物合成机理,本研究利用生物信息学软件分析了六倍体小麦淀粉合成酶基因家族的组成、结构和进化关系。结果表明,小麦基因组中至少存在九个编码淀粉合成酶的基因,分别位于不同的染色体上。九种酶的多肽长度差异较大,而属于同种同工酶的多肽序列长度相近。根据氨基酸序列的催化和糖原转移两个结构域的起始位置,可以将小麦淀粉合成酶基因家族分为两个亚家族。
随着拟南芥和水稻等模式植物全基因组测序的完成(Yu et al., 2002),推动了人们在基因组水平上对植物重要基因家族进行挖掘、比较分析和功能预测。淀粉的生物合成是一个复杂的过程,涉及众多淀粉合成酶的共同作用(Tomlinson et al., 2003),从基因结构的角度可将这些由一个小的多基因家族编码的植物淀粉合成酶划分成GBSS、SSⅠ、SSⅡ、SSⅢ和SSⅣ五 个基因家族(Dian et al., 2005)。目前,对水稻和拟南芥淀粉合成相关酶基因结构、进化和功能已有众多深入研究(Jeng et al., 2009; Szydlowski et al., 2009)。已报道的小麦基因组中存在九个编码淀粉合成酶的基因,部分基因已被成功克隆(Peng et al., 2001; Dian et al., 2003; Leterrier et al., 2008),其中编码GBSSI蛋白的基因为gbssⅠ或waxy基因,有三个高度同源的waxy基因Wx-A1、Wx-B1和Wx-D1已被分离,三个基因序列的相似性达到95.6~96.3%,gbssⅡ基因分别位于2B、2D和2A染色体上,为单拷贝(Murai et al., 1999; Vrinten et al., 2000)。SSⅠ基因位于第7组染色体上,SSⅡ基因也位于第7组染色体上(Li et al., 1999),SSⅢ基因位于第1A染色体上(Li et al., 2000),SSⅣ基 因是一类全新的SS同工型,位于第1组染色体上(Leterrier et al., 2008),目前对其研究尚少。本研究以小麦的相关数据为模板分析了淀粉合成酶基因家族的组成、结构和进化关系,以期为今后进一步研究小麦淀粉生物合成和 淀粉品质遗传改良提供理论基础。
1结果与分析
1.1小麦淀粉合成酶基因家族的组成及结构
小麦基因组中至少存在九个编码淀粉合成酶的基因,包括2个GBSS (TaGBSSⅠ, TaGBSSⅡ),1个SSⅠ (TaSSⅠ),3个SSⅡ (TaSSⅡ-1, TaSSⅡ-2, TaSSⅡ-3),2个SSⅢ (TaSSⅢ-1, TaSSⅢ-2)和1个SSⅣ (TaSSⅣ)。其中SS主要分布在质体基质,部分也与淀粉粒结合,是一种葡萄糖转移酶,催化转移ADP-葡萄糖上的葡萄糖单元,通过α-1,4糖苷键不断增加寡聚糖的葡萄糖单位,最终合成以α-1,4 糖苷键连接的聚糖,聚糖又将作为淀粉分支酶的底物合成支链淀粉。GBSSI主要与淀粉颗粒结合,在直链淀粉合成中起关键作用,将ADP-葡萄糖的葡萄糖残 基转移到α-1,4葡聚糖链的非还原性末端(Vrinten et al., 2000)。为了深入分析小麦淀粉合成酶基因家族的结构与功能,本研究对已经报道的小麦淀粉合成酶家族的氨基酸序列进行了比对和分析。结果发现,九种酶的 多肽长度差异较大,如TaSSⅢ-1的长度几乎是GBSSⅡ的三倍,而属于同种同工酶的多肽序列长度相近。通过多序列比对结果,分别属于同种同工酶的氨基 酸序列之间相似性(60~95%)高于不同种同工酶(20~40%)之间(表1)。
.png) 表1 小麦淀粉合成酶氨基酸序列比较 Table 1 Comparison of wheat starch synthase protein sequences |
通过多序列比对发现(图1),这九种酶均存在两个结构域:催化结构域和糖原转移结构域。催化结构域的序列保守性要明显低于糖原转移结构域的保守性。按照两个结构域的起始位置,可以将九种酶分为两个亚家族:第一组:TaSSⅠ、TaSSⅡ-1、TaSSⅡ-2、TaSSⅡ-3、GBSSⅠ和GBSSⅡ,第二组:TaSSⅢ-1、TaSSSⅢ-2和TaSSⅣ。
 图1 小麦淀粉合成酶的氨基酸序列同源性比较 Figure 1 Comparison of the predicted amino acid sequences of wheat starch synthases |
通过分析发现,两个亚家族的不同结构域的氨基酸组分并无显著差别, 均由20种不同的氨基酸组成,但频率(%)不同(表2)。TaSSⅡ三个同工酶的催化结构域和糖原转移结构域中各氨基酸频率差异不大,GBSS两个同工酶的不同结构域中氨基酸频率差异最大。然而,通过GBSSⅠ和GBSSⅡ序列比对,发现其同源性很高,序列相似度达到60% (图2)。
表2 小麦淀粉合成酶不同结构域氨基酸组分分析 Table 2 Amino acid frequencies of starch synthases different domain. All frequencies are given in percent |
 图2 小麦淀粉粒结合淀粉合成酶的氨基酸序列同源性比较 Figure 2 Comparison of the predicted amino acid sequences of wheat granule-bounding starch synthases |
同时,本研究对部分淀粉合成酶基因全长做了结构分析,TaSSⅡ三个同工酶基因具有相似的内含子/外显子结构,有8个外显子和7个内含子。TaSSⅣ有16个外显子、15个内含子和2个非翻译区。GBSSⅠ基因长度相对较短,有11个外显子和10个内含子组成(图3)。
图3 部分小麦淀粉合成酶基因的结构分析 Figure 3 Schematic gene structure of partial wheat starch synthases gene |
1.2系统树分析
有研究将植物淀粉合成酶(SS)和原核生物糖原合成酶(GS)的氨基酸序列进行比对,识别出了SS/GS中保守的氨基酸,并认为这些保守的氨基酸可能形成了淀粉合成酶的催化结构域(Cao et al., 1999)。通过多序列比对和MEGA 4.0程序生成植物淀粉合成酶系统进化树(图4)。可将这31个氨基酸序列明显分为3个亚家族:SSⅠ和SSⅡ为同一亚家族,GBSS为一个亚家族,SS Ⅲ和SSⅣ属同一个亚家族,此结果和九个小麦淀粉合成酶的进化关系树相同(图5)。除个别节点外,其余节点支持率都较高,说明分枝的可信度高,进化过程中 亲缘关系较近,如水稻SSⅣb和小麦SSⅣ节点支持率为100,基因同源性达到86% (Leterrier et al., 2008)。每个亚家族中又可进一步细化为单子叶植物和双子叶植物。
.png) 图4 植物淀粉合成酶的进化关系 Figure 4 Phylogenetic tree showing the relationship of plant starch synthases based on their deduced amino acid sequences |
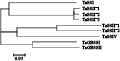 图5 小麦淀粉合成酶进化关系 Figure 5 Phylogenetic relationships of wheat starch synthases |
2讨论
水稻和拟南芥基因组测序的完成推动了对其基因和染色体的研究(Yu et al., 2002),使得人们在基因组水平上对植物重要基因家族进行挖掘、比较分析和功能预测变为现实。特别是生物信息学相关技术的开发,在植物大规模的基因分 离、基因功能注释及功能基因组学等各项研究中发挥越来越重要的作用(王岩等, 2009)。普通小麦是一个异源六倍体物种,有A、B、D三个不同的基因组。特别是B基因组含有丰富的抗病虫害等有益基因(于海霞和田纪春, 2008)。对小麦各个染色体组进行有关起源、进化、基因定位及基因与产量、生物性状的关联分析,可更好的发掘和利用各染色体上的有利基因,拓宽小麦的遗 传变异基础,为在小麦育种中如何引进新的遗传变异及杂优选育提供理论依据。
佃蔚敏等(2004)研究发现所有水稻SS基因家族蛋白质的C -端氨基酸序列保守性均较强,和细菌的糖原合成酶同源,与酵母和高等动物的糖原合成酶同源性极低,并且认为植物淀粉合成酶与酵母和高等动物的糖原合成酶可能源自于不同的祖先。小麦基因组中存在九个编码淀粉合成酶的基因,分散在不同染色体或染色体的不同位点,非成簇分布。和小麦相似,拟南芥和水稻基因组中淀粉合成酶也分别由不同位点基因编码,说明淀粉合成酶基因家族可能是由染色体大片断倍增形成的(佃蔚敏, 2004)。水稻基因组中淀粉合成酶基因的倍增可能发生在单子叶和双子叶植物分化之后,禾本科植物分化之前。小麦、玉米、高粱和大麦等禾本科植物基因组中 淀粉合成酶基因也发现有类似的的倍增(佃蔚敏, 2004)。
目前,关于植物淀粉合成酶基因的分子起源和进化研究较少。然而,研究基因的倍增方式、分化途径可以预测该基因在自然选择的压力下进化过程中的命运(Sankoff, 2001),这些研究将有利于对植物淀粉合成酶基因的表达特性、调控机理和生理功能的进一步探索,同时可为深入研究小麦淀粉合成的分子机理和品质的遗传改 良提供理论基础。
3试验材料与方法
3.1试验材料
从NCBI (http://www.ncbi.nlm.nih.gov)最新数据库下载六倍体小麦相关淀粉合成酶核酸和氨基酸序列,以及其它相关作物的淀粉合成酶氨基酸序列。
3.2淀粉合成酶基因系统发生学分析
应用多序列比对工具Clustal X构建系统进化树(Ramu et al., 2003),以相关淀粉合成酶氨基酸全序列联配的结果为基础,用MEGA 4.0程序生成(Tamura et al., 2007)。采用程序中的Neighbor-joining法,运行采用如下参数:选择Poisson Correction(泊松修正)这一模型,缺口设置为“Pairwise Deletion”,节点支持率使用1 000次Bootstrap检验。
3.3淀粉合成酶氨基酸序列分析
通过Pfam网站(The protein families database, http://www.sanger.ac.uk/Software/Pfam/),根据淀粉合成酶结构的不同,将其进行分类。通过Pfam数据库进行系 统分析得到的淀粉合成酶蛋白质结构域表现形式,进一步分析不同结构域的各氨基酸频率。
3.4淀粉合成酶基因结构分析
通过Gene Structure Display Server在线基因结构分析工具,分析部分淀粉合成酶基因全长的结构。
作者贡献
李春艳、李诚是本研究的实验设计和数据分析的执行人,并完成论文初稿的写作;李卫华、曹连莆指导实验设计与论文修改。全体作者都阅读并同意最终的文本。
致谢
本研究由石河子大学高层次人才科研启动资金专项(RCZX201004)和石河子大学科学技术研究发展计划动植物育种专项(GXJS2008-YZ04)共同资助。本文中提到了我们研究中涉及的有关软件,这并非我们为这些软件开发商提供推荐。
参考文献
Cao H.J., Imparl-Radosevich H.G., Keeling P.L., James M.G., and Myers A.M., 1999, Identification of the soluble starch synthase activities of maize endosperm, Plant Physiol., 120(1): 205-215 doi:10.1104/pp.120.1.205
Dian W.H., Chen J.Q., Liu F., and Wu P., 2003, Cloning and characterization of the granule-bound starch synthase Ⅱ gene in rice: gene expression is regulated by the nitrogen level, sugar and circadian rhythm, Planta, 218(2): 261-268 doi:10.1007/s00425-003-1101-9
Dian W.H., Jiang W.H., and Wu P., 2005, Evolution and expression analysis of starch synthase Ⅲ and Ⅳ in rice, J. Exp. Bot., 56(412): 623-632 doi:10.1093/jxb/eri065
Dian W.M., 2004, Analysis of the starch synthase gene family in rice: members, structure, evolution, function and expression, Dissertation for Ph.D., Supervisor: Wu Ping, Zhejiang University, pp.28-41 (佃蔚敏, 2004, 水稻淀粉合(成)酶基因家族基因组成、结构、进化、功能及其表达调控, 博士学位论文, 浙江大学, 导师: 吴平, pp.28-41)
Jeng T.L., Wang C.S., Tseng T.H., Wu M.T., and Sung J.M., 2009, Nucleotide polymorphisms in the waxy gene of NaN3-induced waxy rice mutants, J. Cereal Sci., 49(1): 112-116 doi:10.1016/j.jcs.2008.07.009
Leterrier M., Holappa L., Broglie K., and Beckles D., 2008, Cloning, characterisation and comparative analysis of a starch synthase Ⅳ gene in wheat: functional and evolutionary implications, BMC Plant Biol., 8: 98-118 doi:10.1186/1471-2229-8-98
Li Z., Chu X., Mouille G., Yan L., Kosar-Hashemi B., Hey S., Napier J., Shewry P., Clarke B., Appels R., Morell M., and Rahman S., 1999, The localization and expression of the class Ⅱ starch synthases of wheat, Plant Physiol., 120(4): 1147-1156 doi:10.1104/pp.120.4.1147
Li Z., Mouille G., Kosar-Hashemi B., Rahman S., Clarke B., Gale K., Appels R., and Morell M., 2000, The structure and expression of the wheat starch synthase Ⅲ gene. Motifs in the expressed gene define the lineage of the starch synthase Ⅲ gene family, Plant Physiol., 123(2): 613-624 doi:10.1104/pp.123.2.613
Murai J., Taira T., and Ohta D., 1999, Isolation and characterization of the three Waxy genes encoding the granule-bound starch synthase in hexaploid wheat, Gene, 234(1): 71-79 doi:10.1016/S0378-1119(99)00178-X
Peng M., Hucl P., and Chibbar R.N., 2001, Isolation, characterization and expression analysis of starch synthase Ⅰ from wheat (Triticum aestivum L.), Plant Sci., 161(6): 1055-1062 doi:10.1016/S0168-9452(01)00481-2
Ramu C., Sugawara H., Koike T., Lopez R., Gibson T.J., Higgins D.G., and Thomppson J.D., 2003, Multiple sequences alignment with the Clustal series of programs, Nucl. Acids Res., 31(13): 3497-3500 doi:10.1093/nar/gkg500
Sankoff D., 2001, Gene and genome duplication, Curr. Opin. in Gen&Develop., 11(6): 681-684
Szydlowski N., Ragel P., Raynaud S., Lucas M.M., Roldan I., Montero M., Munoz F.J., Ovecka M., Bahaji A., Planchot V., Pozueta-Romero J., D'Hulst C., and Merida A., 2009, Starch granule initiation in Arabidopsis requires the presence of either class Ⅳ or class Ⅲ starch synthases, Plant Cell, 21(8): 2443-2457 doi:10.1105/tpc.109.066522
Tamura K., Dudley J., Nei M., and Kumar S., 2007, MEGA4: Molecular Evolutionary genetic analysis (MEGA) software version 4.0, Mol. Bio. Evol., 24(8): 1596-1599 doi:10.1093/molbev/msm092
Tomlinson K., Denyer K., and Callow J.A., 2003, Starch synthesis in cereal grains, Advances in Botanical Research, Academic Press, 40: 1-61 doi:10.1016/S0065-2296(05)40001-4
Vrinten P., and Nakamura T., 2000, Wheat granule-bound starch synthase Ⅰ and Ⅱ are encoded by separate genes that are expressed in different tissues, Plant Physiol., 122(1): 255-263 doi:10.1104/pp.122.1.255
Wang Y., Li Z.Y., Tang X.L., Lu S., Xu P., Zhang J., Fang K., and Xi J.H., 2009, Bioinformatic analysis of the NBS-LRR gene family in Arabidopsis thaliana, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 25(15): 40-45 (王岩, 李兆阳, 唐心龙, 卢姗, 许鹏, 张静, 方奎, 席景会, 2009, 拟南芥基因组NBS-LRR类基因家族的生物信息学分析, 中国农学通报, 25(15): 40-45)
Yu H. X., and Tian J. C., 2008, Review of genome B in T. aestivum L., Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 6(4): 724-732 (于海霞, 田纪春, 2008, 普通小麦B 基因组的研究进展, 分子植物育种, 6(4): 724-732)
Yu J., Hu S., Wang J., Wong G., Li S., 2002, A draft sequence of the rice genome (Oryza Sativa L. ssp. Indica), Science, 296(5565): 79-92 doi:10.1126/science.1068037