2山东省农业科学院高新技术中心, 济南, 250100
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 107 篇 doi: 10.5376/mpb.cn.2011.09.0107
收稿日期: 2011年08月24日 接受日期: 2011年10月08日 发表日期: 2011年10月12日
引用格式(中文):
王俊峰等, 2011, 山东省12份水稻种质的DNA指纹数据库构建, 分子植物育种(online) Vol.9 No.107 pp.1776-1783 (doi: 10.5376/mpb.cn.2011.09.0107)
引用格式(英文):
Wang et al., 2011, Construction of DNA Fingerprinting Database for 12 Oryza Species in Shandong, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.9 No.107 pp.1776-1783 (doi: 10.5376/mpb.cn.2011.09.0107)
本文以12份山东省特有、优异的水稻种质资源为材料,根据国家水稻数据中心公布的水稻DNA指纹数据库构建标准,通过SSR技术对其进行了DNA指纹数据库的构建。结果显示,所采用的24对引物在本实验材料中共检测到73个等位基因,有较好的多态性。利用NTSYS进行UPGMA聚类分析后,发现这12种材料在遗传相似系数0.696为阀值处可以分为3个类群。实验证明该方法能够分辨各个种质间的亲缘关系,能够较好满足水稻DNA指纹数据库的构建要求,对保护山东省特有的水稻种质资源具有重要意义。
电泳指纹图谱技术,特别是DNA指纹图谱技术,具有不受环境、遗传稳定,多态性高,且不影响目标性状等优点,目前已被广泛用于农作物种质资源的鉴定(陈杰等, 2007, 农业考古, 3: 69-71),并在农作物种质资源的亲缘关系鉴定、品种审定和杂优类群划分等方面发挥着巨大的作用(滕海涛等, 2009, 1: 1-6)。通过计算机对可见的条码式的DNA指纹图谱进行系统管理而构建的数据库就被称为DNA指纹数据库。国际植物品种权保护组织(UPOV)于2005年拟定的BMT测试指南草案中将简单序列重复(Simple Sequence Repeats, SSR)和单核苷酸多态性(Single Nucleotide Polymorphisms, SNP)等分子标记技术确定为构建DNA指纹数据库的标记方法。
国外在DNA指纹数据库的构建领域的研究刚刚起步(Rekha et al., 2011; Fleury et al., 2010; Nelson et al., 2005)。我国在农作物种质资源DNA指纹数据库的构建上取得了重要进展(王凤格等, 2006, 2007; 刘冠明等, 2006; 王立新等, 2007; 陈英华等, 2009; 王俊芳等, 2009, 中国棉花, 36(3): 26-24),目前已有少数农作物在DNA指纹数据库的构建方面形成了一个统一的标准(王凤格等, 2006, 2007; 王俊芳等, 2009, 中国棉花, 36(3): 26-24),水稻是为数不多的几种农作物之一。尽管中国水稻研究所国家水稻数据中心(http://www.ricedata.cn)目前已公布了一系列的水稻分子标记的信息和技术标准,但是由于我国拥有比较丰富的水稻种质资源,目前我国仍处于进行种质资源的调查和整理阶段。
当前我国的水稻种质资源的来源地多集中于种质资源丰富的南方和东北等地区,所以目前关于水稻的研究大多集中在此范围内。2009年,陈英华等从500对 SSR引物中筛选出10对核心引物,用于构建东北地区近两年区域试验的水稻品种的DNA指纹图谱,并在100对多态性位点上检测到300个等位基因(陈英华等, 2009)。同年马红勃等以福建省三明市农科所选育,福建六三种业有限责任公司经营的24份杂交稻品种和18份杂交稻亲本为材料,依据中国水稻研究所推荐的12个SSR标记,建立了42份材料的12个SSR标记的DNA指纹图谱(马红勃等, 2009, 三明农业科技, 3(115): 1-4)。也是在2009年,中国水稻研究所的程本义等,利用前期研究建立的一套水稻品种DNA指纹检测技术体系,对2002-2006年浙江省主要的 98个水稻品种、以及2007-2008年155个浙江省区试水稻品种(181个, 含26个续试品种)进行了DNA指纹检测,构建了279个次水稻品种×12个微卫星标记的DNA指纹图谱数据库(程本义等, 2009)。2010年,贵州大学马琳等,从145对SSR引物中筛选出21对,对24份贵州地方水稻品种/禾0品种进行遗传多样性分析,初步建立了其 DNA指纹图谱,并通过聚类分析将所有材料分为5类,聚类结果表明品种间的亲缘关系与地理来源关系不大(马琳等, 2010)。而对于黄淮海地区,特别是山东等非水稻传统种植区域,由于水稻种质资源不多,对其所属地域中的水稻的研究相对于其他地区还比较少。本文根据中国水稻研究所国家水稻数据中心公布的信息和技术标准,对当前山东省特有的几种优异水稻种质资源进行了SSR分析,构建了其DNA指纹图谱数据库。由于山东省特有的水稻种质资源是国家水稻种质资源的有效补充,所以本研究对保护水稻种质资源的丰富性具有重要意义。现将主要结果公布如下。
1结果与分析
1.1 SSR引物标记多态性分析
在本实验采用的12份品种中(表1),所用24对引物共检测到73个等位基因,每个SSR位点检测到的等位基因为1~7个,平均每对引物的等位基因 3.04个(表2)。其中引物RM71、RM85、RM267、RM278、RM258和RM19检测到的等位基因数最少(各1个),引物RM274检测的等位基因数最多(7个)。实验表明,上述的24对引物在实验材料中有较好的多态性,基本上能够满足水稻DNA指纹数据库的构建要求。

表1 试验材料名称, 来源及编号
Table 1 List of cultivars used
![]()
表2 24个SSR标记座位上的带型数据
Table 2 Alleles detected at 24 SSR loci
在与国家水稻数据中心公布的等位基因的信息进行比对时,我们发现在本实验中个别SSR位点检测到的等位基因数目比国家水稻中心公布的要多。如RM274在本实验中分别检测到7个等位基因,比国家水稻中心公布的等位基因多出5个(图1)。

图1 12种山东省特有水稻品种中第五染色体长臂和短臂分子标记RM274, RM267, RM17和RM19的多态性分布; 1~12分别代表12种不同的水稻品种, 见表1
Figure 1 Polymorphism of markers RM274, RM267, RM17 and RM19 on the fifth chromosome for 12 kinds of unique rice cultivars in Shandong Province; 1~12: 12 kinds of unique rice cultivars, respectively, shown in table 1
1.2 12种水稻种质资源的DNA指纹数据库的初步构建
根据带型清晰、可重复的原则,由24个SSR标记中认定了73个等位基因,并依据AlphaView软件中条带与Marker的相对电泳位移计算出相应的分子量,将等位基因按分子量从大到小按引物名称加序号的方式进行编号,于是得到了12种水稻材料的DNA指纹数据库(表3),每种材料的指纹信息分别包含 24个SSR标记所检测到的不同等位基因的所处染色体位置等基本信息(表4)。

表3 24个SSR引物构建的12种山东水稻的DNA指纹图谱
Table 3 DNA finger print for 12 kinds of unique rice cultivars in Shandong Province at 24 SSR makers

表4 水稻SSR引物基本信息
Table 4 List of the 24 SSR markers selected for the identification of rice cultivars
1.3品种相似性及聚类分析
根据24个SSR引物所检测出的73个等位基因,对12份材料进行聚类分析。在图2中,遗传相似系数的变异范围为0.68~0.84。在遗传相似系数 0.696为阀值处,可以将12份材料均为粳稻,可分为3个类群,第Ⅰ类群包含彩之恢、黑壳香稻、香血稻、抗虫1和盐粳8号,它们都属于糯稻。第Ⅱ类群包含临稻10号、临稻11号、红旗16号、鲁粳1号和宫808,它们都属于粳型常规水稻。第Ⅲ类群包含临稻2号和临稻3号它们属于粳型常规水稻。实验结果分析表明,SSR的聚类结果能较好地反映出材料之间的亲缘关系。
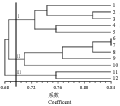
图2 12份材料的SSR聚类分析
Figure 2 Cluster dendrogram of 12 cultivars based on SSR makers
2讨论
2.1 SSR引物标记多态性分析
本实验中,由于所采用的材料为山东省特有的几种优异水稻种质资源,总材料数目较少,遗传多样性较窄(表2),绝大多数SSR引物检测到的等位基因数相对于国家公布数据较低,如国家水稻数据中心公布的RM71、RM336和RM278等位基因数分别为5、7和6,而本实验材料中所检测到的上述引物等位基因数分别为1、5和1。但是也有个别引物检测到的等位基因数高于国家水稻数据中心公布的数据,如本实验材料中RM274和RM190检测到的等位基因数分别为 7和5,高于国家水稻数据中心公布的2和4。这表明,山东省特有的、优异的水稻有着独特的遗传背景和基因资源,是水稻遗传育种工作不可多得的重要遗传种质材料,应该引起有关部门的重视并予以相应的保护。
2.2水稻DNA指纹数据库的兼容
经过国内专家多年的努力,国家初步制定了水稻的DNA指纹数据库标准。然而在构建山东省特有的、优异的水稻DNA指纹数据库时我们发现,各个省份所建立的数据库在兼容性上还存在一定的问题,导致了各个数据库在交换信息上有一定的障碍,不能整合为一个有效的全国性的综合数据库。兼容性问题主要表现在以下几个方面:
(一)建立水稻DNA指纹数据库的几个关键性因素仍没有统一标准。标记方法和标记来源、严格规范的检测平台和核心引物的选定。目前国内DNA指纹数据库的标记方法和来源一般是参照国际上的通用标准SSR标记方法,个别的采用非主流的方法和标准(陈英华等, 2009; 程本义等, 2009; 马琳等, 2010; 马红勃等, 2010; 马红勃等, 2009, 三明农业科技, 3: 1-4)。检测平台则因各个科研单位人员、技术、设备和资金等方面因素的差异而有所不同,一般为性聚丙烯酰胺凝胶电泳,少数单位采用毛细管电泳]。特别是核心引物的选用方面比较多样化,由于核心引物的选择差异,导致了各个数据库的兼容性方面出现了一定的难度,这可能成为今后工作的重点之一。
(二)国家标准上的一些漏洞。尽管国家水稻数据中心公布了水稻DNA指纹数据库构建要素的几个标准,如核心引物、凝胶选用和电泳方式。但是我们还是发现,在一些基本要素以外仍有必要进行进一步的规范。比如各个引物退火温度,这一问题直接影响了实验的可重复性和与国家数据库进行比对的可操作性。其次实验的精度问题也应引起注意,国家数据中心公布的个别等位基因(如RM208)在10个碱基内有7个等位基因,而其所用的6%的丙烯酰胺凝胶和分子量Marker 分辨率不够,明显的不能够对它们进行精确定位。最后国家数据中心公布的等位基因的信息不完整,如RM85的等位基因变异范围是90~100 bp,然而标准图板中给出的最小分子量Marker只有100 bp,明显不能分辨100 bp以下的等位基因,类似的问题在RM208和RM219的等位基因上也出现过。
(三) DNA数据库信息化标准不一。有些地方采用的是本实验用的数据处理方式,即根据国家数据中心公布的等位基因进行比对,对自身检测到的等位基因进行编号。然而个别地方没有与国家数据中心数据进行比对,直接按自身实验结果进行编号(马红勃等, 2009, 三明农业科技, 3: 1-4)。这就为以后各地数据库的兼容在信息处理层面造成了一定的困难。
总之,DNA指纹图谱技术已经在种质品种鉴定、种子质量和保护专利权上发挥着重要作用,它也被应用到品种的亲缘关系划分、农作物育种和农作物遗传作图等方面(王忠华, 2006; 陈杰等, 2007, 农业考古, 3: 69-71)。但是,由于我国在水稻DNA指纹图谱的研究上才刚刚起步,国内各个研究机构研究水平不一,DNA指纹数据库构建所采用的技术、标准和软件等各不相同,存在着若干个不同的DNA指纹数据库,且大多数数据库间存在不能兼容的现状(陈英华等, 2009; 程本义等, 2009; 马琳等, 2010; 马红勃等, 2010; 马红勃等, 2009, 三明农业科技, 3: 1-4)。这种情况的存在限制了各个数据库之间的数据交换并导致资源的浪费,更为重要的是它使水稻DNA指纹数据库间没有了统一标准,造成了单个数据库容量不足、覆盖率不足、分辨率不足和不稳定等缺点,阻碍了水稻DNA指纹数据库的构建和发展,影响了对水稻分子育种工作的深入研究,对该问题的研究及解决应该引起我们的重视。我国具有丰富且独特的水稻种质及其基因资源,加速构建水稻DNA指纹数据库的基本平台,统一技术标准,对实现我国水稻DNA指纹数据库的标准化、自动化和数字化有着重要意义。
3实验材料与方法
3.1实验材料
本研究的实验材料由山东省农业科学院高新技术中心的姚方印研究员提供。选取山东省特有、优异水稻资源12份品种名称及编号见表1。
3.2水稻的萌发
在15 cm×15 cm的方形培养皿中,放入4张大小合适的方形滤纸,加入蒸馏水润湿后,摆放水稻种子。将放有种子的培养皿置于27℃的培养箱中,暗培养7 d。
3.3 DNA的提取
取出于27℃暗培养7 d后的水稻幼苗,放入研钵中并加入液氮,研成粉末后,通过CTAB法提取其DNA (Doyle and Doyle, 1990)。加入RNaseA (北京天根),去除样品中的RNA。加入酚氯仿,变形样品中剩余的蛋白,并通过冷冻的异丙醇沉定DNA。
3.4 PCR
在10 μL 的Taq Plus PCR MasterMix (北京天根)中体系中加入0.1 μg DNA和1 μL 引物(中国水稻研究所国家水稻数据中心, 表4),通过以下条件进行PCR:94℃预热3 min,94℃ 30 s,51℃ 30 s,72℃ 30 s,35个循环结束后72℃延伸5 min。
3.5 SSR电泳
电泳采用6%的非变性丙烯酰胺凝胶电泳,设备为BIO-RAD的核酸序列分析电泳Sequi-Gen GT。每个泳道上样2 μL,恒定功率65 W,电泳80 min后,通过碳酸钠银染的方法显示条带(Röder et al.,1998)。
3.6数据记录与分析
利用AlphaView1.2.0.1软件(Alpha, 美国)进行SSR位点检测与分子量计算,所采用的分子量Marker为由单独制备的PCR产物混合而成的D2000 (北京天根)。根据国家水稻数据中心公布的24对SSR引物扩增所得的等位基因分子量及多态性等信息,对比本实验所检测到的位点多态性,将符合条件的条带视为1个等位基因并进行记录。根据相同迁移位置上(即分子量相同的DNA片段)条带分布情况对数据进行记录,有带时赋值为1,无带时赋值为0的标准,全部转化为1/0格式。数据采用NTSYS2.10e软件中similarity程序计算相似系数,以clustering程序中SHAN进行UPGMA (非加权组平均法)聚类分析(马红勃等, 2010)。
作者贡献
王俊峰为本实验的主要构思和实施者,为论文初稿的主要编写人员。李娜娜,张煜和马玉敏参与了部分实验的实施,余华参与了实验数据的分析。姚方印提供了本实验的材料并对实验实施进行了指导。丁汉凤为本文通讯作者,构思者及负责人,指导论文写作、修改及定稿。全体作者都阅读并同意最终的文本。
致谢
本文由山东省农业良种工程(主要农作物物种种质资源的收集, 鉴定与入库保存, 2010LZ01-01)和国家科技基础性工作专项资金(山东省沿海地区抗旱耐盐优异性状农作物种质资源调查, 2007FY110500)共同提供支持。
参考文献
Chen Y.H., Hou Y.M., Li H.Y., Li M.B., Yuan Y., and Xu Z.J., 2009, Establishment of DNA fingerprinting and analysis of genetic diversity among japonica rice cultivars attended regional trials in northeast region of China, Zhongzi (Seed), 28(3): 28-35 (陈英华, 侯昱铭, 李宏宇, 李茂柏, 袁媛, 徐正进, 2009, 东北地区水稻区试新品种的DNA指纹图谱构建及遗传多样性分析, 种子, 28(3): 28-35)
Cheng B.Y., Wu W., Xia J.H., Liu X., Zhuang J.Y., and Yang S.H., 2009, Construction and application of DNA fingerprint database of rice varieties in Zhejiang Province, Zhejiang Nongye Xuebao (Acta Agriculturae Zhejiangensis), 21(6): 555-560 (程本义, 吴伟, 夏俊辉, 刘鑫, 庄杰云, 杨仕华, 2009, 浙江省水稻品种DNA指纹数据库的初步构建及其应用, 浙江农业学报, 21(6): 555-560)
Doyle J.J., and Doyle J.I., 1990, Isolation of plant DNA from fresh tissue, Focus, 12(1): 13-15
Fleury D., Luo M.C., Dvorak J., Ramsay L., Gill B.S., Anderson O.D., You F.M., Shoaei Z., Deal K.R., and Langridge P., 2010, Physical mapping of a large plant genome using global high-information-content-fingerprinting: the distal region of the wheat ancestor Aegilops tauschii chromosome 3DS, BMC Genomics, 11: 382 http://dx.doi.org/10.1186/1471-2164-11-382
Liu G.M., Zheng Y.X., and Li G.L., 2006, SSR Fingerprinting map of 20 peanut cultivars, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 22(6): 49-51 (刘冠明, 郑奕雄, 黎国良, 2006, 20个花生品种的SSR标记指纹图谱构建, 中国农学通报, 22(6): 49-51)
Ma H.B., Xu X.M., Wei X.Y., Yang W.X., and Zou W.G., 2010, DNA fingerprints and genetic diversity analysis based on SSR markers for rice cultivars in Fujian, Fujian Journal of Agricultural Sciences, 25(1): 33-38 (马红勃, 许旭明, 韦新宇,杨旺兴, 邹文广, 2010, 基于SSR标记的福建省若干水稻品种DNA指纹图谱构建及遗传多样性分析, 福建农业学报, 25(1): 33-38)
Ma L., Yu X.Q., and Zhao F.S., 2010, Establishment of SSR Fingerprint Map of Local Rice Varieties “He” in Guizhou, Xinana Nongye Xuebao (Southwest China Journal of Agricultural Sciences), 23(1): 5-10 (马琳, 余显权, 赵福胜, 2010, 贵州地方水稻品种“禾”的SSR指纹图谱构建, 西南农业学报, 23(1): 5-10)
Nelson W.M., Bharti A.K., Butler E., Wei F., Fuks G., Kim H., Wing R.A., Messing J., and Soderlund C., 2005, Whole-genome validation of high-information-content fingerprinting, Plant Physiol., 139(1): 27-38 http://dx.doi.org/10.1104/pp.105.061978
Rekha T., Martin K.P., Sreekumar V.B., and Madassery J., 2011, Genetic diversity assessment of rarely cultivated traditional indica rice (Oryza sativa L.) varieties, Biotechnol Res. Int., doi: 10.4061/2011/784719 http://dx.doi.org/10.4061/2011/784719
Röder M.S., Korzun V., Wendehake K., Plaschke J., Tixier M.H., Leroy P., and Ganal M.W., 1998, A microsatellite map of wheat, Genetics, 149(4): 2007-2023
Teng H.T., Lv B., Zhao J.R., Xu Y., Wang F.G., Du Y.Y., Yang K., Tang H., and Li X.Y., 2009, DNA fingerprint profile involved in plant variety protection practice, Biotechnology Bulletin, 1: 1-6 (滕海涛, 吕波, 赵久然, 徐岩, 王凤格, 堵苑苑, 杨坤, 唐浩, 李祥羽, 2009, 利用DNA指纹图谱辅助植物新品种保护的可能性, 生物技术通报, 1: 1-6)
Wang F.G., Zhao J.R., Dai J.R., Guo J.L., Yuan Y.P., Wang L., Yi H.M., Sun S.X., and Lv B., 2006, Criteria for the construction of maize DNA fingerprint database, Yumi Kexue (Journal of Maize Sciences), 14(6): 66-68 (王凤格, 赵久然, 戴景瑞, 郭景伦, 原亚萍, 王璐, 易红梅, 孙世贤, 吕波, 2006, 玉米DNA指纹数据库建库标准规范的建立, 玉米科学, 14(6): 66-68)
Wang F.G., Zhao J.R., Dai J.R., Guo J.L., Yuan Y.P., Wang L., Yi H.M., Sun S.X., and Lv B., 2007, Criteria for the construction of maize DNA fingerprint database, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 5(1): 128-132 (王凤格, 赵久然, 戴景瑞, 郭景伦, 原亚萍, 王璐, 易红梅, 孙世贤, 吕波, 2007, 玉米品种DNA指纹数据库构建的标准化规范, 分子植物育种, 5(1): 128-132)
Wang L.X., Li Y.F., Chang L.F., Huang L., Li H.B., Ge L.L., Liu L.H., Yao J., and Zhao C.P., 2007, Method of ID Constitution for Wheat Cultivars, Zuowu Xuebao (Acta Agronomica Sinica), 33(10): 1738-1740 (王立新, 李云伏, 常利芳, 黄岚, 李宏博, 葛玲玲, 刘丽华, 姚骥, 赵昌平, 2007, 建立小麦品种DNA指纹的方法研究, 作物学报, 33(10): 1738-1740)
Wang Z.H., 2006, DNA fingerprinting technology and its application in crop germplasm resources, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 4(3): 425-430 (王忠华, 2006, DNA指纹图谱技术及其在作物品种资源中的应用, 分子植物育种, 4(3): 425-430)