2. 海南省热带农业资源开发利用研究所, 三亚, 572025
3. 广西大学生命科学与技术学院, 南宁, 530005
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2015 年, 第 13 卷, 第 1 篇 doi: 10.5376/mpb.cn.2015.13.0001
收稿日期: 2015年01月07日 接受日期: 2015年02月08日 发表日期: 2015年03月25日
引用格式(中文):
李宗飞等, 2015, 植物生物钟的基因调控网络, 分子植物育种(online), 13(1): 1001-1008 (doi: 10.5376/mpb.cn.2015.13.0001)
引用格式(英文):
Li et al., 2015, Gene Regulation Network of Biological Clock in Plant, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding), 13(1): 1001-1008 (doi: 10.5376/mpb.cn.2015.13.0001)
生物钟是生物生理活动的周期性波动的内生节奏,这种内生节奏的周期实际上并不是准确的24 h,而是在22~28 h之间,因此称为近似昼夜节律,也称为昼夜节律时间钟或生物钟。受植物生物钟的调节,植物的许多生理和生化反应都表现出一种内在的近似于24 h的昼夜节律现象。在大多数高等植物中,其内在的生物钟机制几乎参与调控植物体所有的新陈代谢、生长发育过程,明显的控制着自身的许多生理生化反应,如花期转化,叶片运动,气孔闭合,种子萌发,光合作用等。植物的生物钟系统主要由生物钟输入部分,中央振荡器,生物钟输出部分和阀门效应器组成,而生物输入部分主要是光照和温度的输入。目前已有不少专家学者对生物钟调控模型及其作用机制的进行了研究,其中以双子叶植物之模式植物拟南芥的研究最为详实,但是依然还有许多关键基因等待挖掘。
生物钟是生物生理活动的周期性波动的内生节奏,这种内生节奏的周期实际上并不是准确的24 h,而是在22~28 h之间,因此称为近似昼夜节律(circadian rhythms),也称为昼夜节律时间钟(circadian clock)或生物钟(biological clock)。受植物生物钟的调节,植物的许多生理和生化反应都表现出一种内在的近似于24 h的昼夜节律现象。
植物生物钟以近24 h的周期控制着植物体内大部分的生理活动(Dunlap, 1999; Young and Kay, 2001; 李宗飞等, 2015)。这种近24 h周期的生理节奏的分子基础就是植物生物钟的大量相关基因不断地周期性的表达。在模式植物拟南芥中的基因组功能研究和遗传学研究已经发现数百个基因的表达受到生物钟的调控(Harmer et al.,2000; Schaffer et al., 2001)。植物叶片的运动,植物应对外界环境中的非生物胁迫,胚轴的延伸,尤其是受光周期调控的花期都是由植物内在的生物钟控制的(Barak et al., 2000; Mcclung, 2001; Nakamichi et al., 2009)。植物生物钟调控涉及到一系列与外界环境因子同步的机制(Devlin and Kay, 2001)。植物体内在的定时系统可以根据到环境中温度和光照以近似24 h为周期的变化,调节自身的生理活动,使得能够在特殊的阶段进行特定的生理活动(Johnson, 2001),如植物会通过体内的生物钟感受到昼夜节律,因此调控植物的开花时间、光合作用、代谢、生长、结实等生理过程。植物的生物钟是一种进化的结果,是地球上的主要环境因子如光照和温度以24 h为变化周期的长期的自然选择的结果。
1植物生物钟调控模型及基因网络的特点
MAS(2005)构建了高等植物中的生物钟一个经典的模型(Más, 2005),植物的生物钟系统被分为4个主要的部分:生物钟输入部分,中央振荡器,生物钟输出部分和阀门效应器(图1)。生物钟输入部分主要包括光照和温度的输入,其实还包括各种非生物胁迫等信息(Fowler et al., 1999; Kreps et al., 1999; Salter et al., 2003)。植物体内存在着2类主要的光受体,一类是接受红光或者远红光的光敏色素PHY(Phytochrome)基因,在拟南芥中鉴定有5个同源基因,分别是PHYA,PHYB,PHYC,PHYD,PHYE,一类是接受蓝光的隐花色素CRY(Cryptochrome)基因。此外,ZTL(Zeitlupe),FKF1(Flavin-Binding, Kelch Repeat, F-Box 1),LKP2(Lov, Kelchprotein 2)也能接受蓝光信号。他们都参于了植物生物钟的调控网络。ZTL家族不但可以接受蓝光信号,还可能在介导了前面两类光受体和生物钟其他成分之间的作用。但是,至今为止还没有相关的报道明确鉴定出温度信号究竟由何种基因输入植物的生物钟调控网络,只是有相关的研究表明,拟南芥中的伪反应调控因子(pseudo-response regulator) PRR7和PRR9基因以及GI(gigantea)基因可能在温度介导的生物钟系统中发挥作用(Salome and McClung, 2005)。
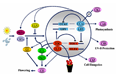 图 1 拟南芥中生物钟调控网络的分子模型 Figure 1 Molecular model of circadian clock regulation network in the Arabidopsis |
在拟南芥中,中央振荡器是一个正向调控和反向调控的成员构成的负调控环,这些成员可以相互调控其节律性的表达量和活性。这种生物钟成员的节律性的表达和调控就产生了生物钟的节律性,并转变成多重生物钟信号的输出。CCA1 (circadian clock associated 1)和LHY(late elongated hypocotyl)蛋白通过直接与(toc1 timing of cab 1)启动子中的特殊序列(被称为黑夜元件)的结合来抑制TOC1的表达(Harmer et al.,2000; Alabadíet al., 2001)。在白天,CCA1/LHY表达水平的降低可以逐渐解除其对TOC1的抑制作用,使得TOC1的mRNA表达量逐渐增加。当TOC1的表达量达到一定量的时候,又会正向调控CCA1和LHY的表达,使得CCA1和LHY的表达量增加,从而形成了一个振荡的生理循环。TOC1对CCA1和LHY表达的正向调控可能不是直接的,因为至少存在着3个其他的成分也参与了正向调控CCA1/LHY的表达,他们分别是ELF3 (early flowering 3),GI,和ELF4 (early flowering 4) (Fowler et al., 1999; Schaffer et al., 2001; Doyle et al., 2002)。
同时,植物生物钟控制着植物的许多生理活动,包括植物的开花(Mariko et al., 2007),胚轴的延伸以及植物的光合作用,蒸腾作用,以及其它各种植物的生长发育活动(Kreps and Kay, 1999; Green et al., 2002; Blasing et al., 2005; Dodd et al., 2005; Niwa et al., 2009)。
2光信号的输入和生物钟的控制
植物生物钟节律性这种内在的生理周期与地球上24小时的日周期相似(Devlin and Kay, 2000)。光是外界环境中时间同步机制的一种极其重要的信号。于是研究者们就把鉴定在光信号途径中与生物钟相关的成员作为生物钟研究的一个重要的线索。在拟南芥中,两类主要的光受体,光敏色素(PHY)和隐花色素(CRY)都参与了由光周期调控的生物钟(Somers et al., 1998)。5个光敏色素中,有4个(PHYA, PHYB, PHYD和PHYE)在红光控制的生物钟途径中起作用,并且它们的作用是叠加的。
同时,CRY作为生物钟的光受体,能够接受蓝光,CRY1和CRY2的功能是冗余的,作为蓝光介导生物钟的受体(Devlin and Kay, 2000)。CRY1和CRY2接受不同强度的蓝光信号,CRY1在蓝光条件下稳定,主要接受高强度的蓝光,而CRY2在蓝光条件下迅速降解,主要接受低强度的蓝光(Lin et al., 1998),蓝光诱导下CRY2的降解可能与ZTL有关。
植物光反应往往是是由隐花色素和光敏色素共同调节的。光敏色素和隐花色素在不同的光照条件下共同作用于一系列相关的生理途径,能够在转录组水平上大量地调控许多下游基因的表达水平。CRY2突变株系在红光条件下影响光信号的输入(Más et al., 2000),表明CRY2基因与光敏色素之间存在着相互作用。隐花色素的突变体植株也会影响红光的输入,表明无论是在红光条件下还是在蓝光条件下,CRY1在PHYA介导的生物钟途径中具有相关的功能(Salome and McClung, 2005)。
另外,ZTL、FKF1、LKP2也能接受蓝光,并且还可以控制光敏色素接受红光和隐花色素接受蓝光的生理过程。ZTL,FKF1,LKP2基因编码的蛋白质含有1个F-box功能结构域。F-box结构域出现在作为受体的蛋白质中,能够将特定的底物如TOC1与蛋白质泛素连接酶连接与降解(Craing and Tyers, 1999)。ZTL蛋白质家族的这些结构域的独特的结合特性表明,他们可能介导生物钟某些成分的降解需要依赖于光的生理过程。对ZTL突变株系的研究表明,生物钟的周期长短依赖于光的密度(Somer et al., 2000),但是,LKP2的过量表达却能导致一些受生物钟控制的基因的无节律性表达(Schultz et al., 2001)。而FKF1在光周期中起着控制CO基因的表达量的作用(Imaizumi et al., 2003)。
这些基因在光输入途径的作用可能是由它们与光受体的直接相互作用决定的。ZTL在体内被发现与PHYB和CRY1基因有相互作用(Jarillo et al., 2001)。光受体作为植物感光的开关,其表达受到外界环境中光信号的紧密控制(Toth et al., 2001)。
无论是光敏色素还是隐花色素,一方面它们通过感知外界环境中的光信号来调控体内的生物钟的振荡节律,另一方面,它们自身的表达也受到内在的生物钟机制的精密调控。即使在外界光照条件恒定的情况下,除了PHYC外,其它的光受体基因的表达也具有生物钟控制的节律性,它们的mRNA的表达水平受到生物钟的控制。这些结果表明在植物体内,存在着一个调控光信号输入和设置生物钟节律的调控环。
同时,植物体内还存在着一系列的阀门机制来控制外界光信号输入生物钟系统,在拟南芥中,ELF3、LUX (lux arrhythmo)、TIC(timeforcoffee)和XCT (xap5 circadian timekeeper)监控着光敏色素介导的光信号的输入,即当外界红光或者远红光的输入达到一定的临界值时,就不再进入生物钟系统(Schaffer et al., 2001; Onai and Ishiura, 2005; Ding et al., 2007; Martin-Tryon and Stacey, 2008)。与之相反,FHY3 (far-red elongated hypocoty l3)和SRR1 (sensitivity to red light reduced 1)却对光敏色数介导的红光或者远红光信号有着正向调控的作用(Staiger et al., 2003; Allen et al., 2006)。
3生物钟调控途径核心途径
生物钟的生理机制在不同的生物体中都是比较保守的,基于它中心振荡器形成的负反馈调控环(Dunlap, 1999)。这个负反馈调控环包括正向调控和反向调控的成员,这些成员之间可以相互调控,来控制每个成员节律性的表达和活性。这种生物钟成员的振荡性的表达和调控就产生了生物钟的节律性,并转变成多重生物钟信号的输出,包括季节性的开花和光形态建设等。
在拟南芥中,CCA1/LHY和TOC1之间的相互构成了中央振荡器的反馈调控环(Alabadí et al., 2001)。LHY和CCA1是一类R1类Myb类型转录因子,在白天达到表达的最高峰,是生物钟中央振荡器的负调控因子,其表达受到TOC1的正向调控。在夜晚,LHY/CCA1的启动子区域含有1个G-box结构,可以与PIF3结合(McWatters et al., 2000),从而抑制LHY/CCA1在夜晚的表达;而在白天经红光照射后,PHYB被激发为活性状态,并与PIF3结合,解除其对CCA1和LHY等基因的抑制;并进一步诱导下游日间基因的基因表达并抑制夜间基因的表达。
LHY与TOC1等夜间基因的启动子与黑夜元件(AAATATCT)相结合从而抑制其的转录表达(Harmer et al., 2000)。在白天,CCA1/LHY表达水平的降低可以逐渐解除其对TOC1的抑制作用,使得TOC1的mRNA表达量的增加。当TOC1的表达量达到一定量的时候,又会正向调控CCA1和LHY的表达,使得CCA1和LHY的表达量增加,从而形成了一个振荡的生理循环。所以,在拟南芥中由CCA1/LHY和TOC1构成的调控途径中,TOC1首先正向调控LHY/CCA1的表达,当LHY/CCA1达到一定的表达量,又会负向抑制TOC1的表达。按照这个模型,MYB转录因子CCA1和LHY是作为转录调控环的负调控元件可以直接结合到TOC1的启动子区域,而TOC1对CCA1和LHY表达的正向调控不是直接的。
CCA1、LHY和TOC1这3者中任何一个的过量表达都会造成拟南芥生物钟相关基因表达,叶片运动,胚轴延伸的无节律性,并且还会造成LHY或者CCA1表达的剧烈降低。而与之不同的是,CCA1,LHY和TOC1这3者中任何一个的缺失突变却会造成生物钟周期的缩短(Wang and Tobin, 1998; Green and Tobin, 1999),并且LHY和CCA1双缺失突变体的生物钟周期比它们2个任何1个的单突变的生物钟周期更短,表明LHY和CCA1的功能存在着部分冗余,相应的研究表明,CCA1和LHY可以在体内或者体外形成二聚体从而相互作用(Sheen et al., 2009)。在拟南芥体内的研究表明,存在着一系列的阀门机制来控制TOC1的表达量,PRR3,ELF4ZIK4以及ZTL都可以监控TOC1在拟南芥体内的表达量,而LHY/CCA1相关研究还未见报道(Doyle et al., 2002; Fujiwara et al., 2008)。
在以往的研究中虽然发现了当CCA1和LHY积累到一定得程度时,就会通过直接结合到TOC1的启动子区域来抑制TOC1的表达,但是与之相反,在TOC1蛋白中,却没有发现相关的功能结构域来调控LHY和CCA1的表达。为了解决这个问题,用酵母单杂交技术来筛选与LHY和CCA1基因调控区域结合的转录因子(Pruneda-Paz et al., 2009),发现TCP转录因子I类家族专一的结合到CCA1基因起始密码子前面的一段长约171 bp的区域,命名为CHE (cca1 hiking expedition),但是CHE蛋白却不结合到LHY基因的相关区域。CHE蛋白结合CCA1基因区域的序列为GGNCCCAC,命名为TBS,进一步的研究表明,在夜晚,CCA1和LHY的高表达一致了CHE的表达,而在白天,CHE的表达量不断增加,从而将CCA1和LHY的表达量抑制到最低。在白天结束的时候,TOC1会通过与CHE的结合,来重新开始下一个循环(图2)。
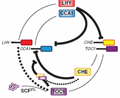 Figure 2 The model of Feedback loops of core path of circadian clock regulation in plant |
在拟南芥中,还存在着4个与TOC1编码相似蛋白的基因,它们分别是PRR3,PRR5,PRR7,PRR9。这5个基因编码的蛋白质都含有1个与细菌中的反应调控结构域类似的结构域,但是缺乏信号传导途径中双成分保守结构域中所普遍具有的能够接受磷酸化的冬氨酸残基(Mizuno and Nakamichi, 2005)。PRR因子还含有1个CTT(首先在CO和TOC1蛋白中被鉴定)保守结构域,这个结构域的主要功能是能够定位于细胞核中(Makino et al., 2000)。PRR基因对生物钟的周期,某个阶段以及幅度,开花时间以及对红光控制的下胚轴的延伸的敏感性都有影响(Mizuno, 2004)。PRR转录物有节律性的积累,顺序为PRR9-PRR7-PRR5-PRR3-TOC1。其表达最高峰出现在黄昏后2个小时到10个小时从PRR9到TOC1(Matsushika et al., 2000; Strayer et al., 2000)。
在PRR的这5个基因中,PRR7和PRR9是日间表达基因,受到LHY和CCA1的正向调控,但是PRR7和PRR9却可以抑制LHY和CCA1的表达。这样,LHY、CCA1、PRR7、PRR9之间也有可能形成了负反馈转录调控环,并且可能与LHY、CCA1和TOC1构成的中央振荡器有着紧密的关系(Zeilinger et al., 2006)。
4植物生物钟的转录后调控
在植物中,CK激酶的磷酸化和ZTL蛋白介导的E3泛素连接酶SCF复合物降解作用是目前已知生物钟相关蛋白转录后调控的主要方式。果蝇,脉孢菌和人类中的相关研究表明,CK1 (casein kinase 1)和CK2是生物钟核心成员磷酸化过程中的主要蛋白激酶(Toh et al., 2001; Dunlap, 2004; Edery, 2005) 。在拟南芥中,CK2对CCA1的磷酸化对维持拟南芥生物钟的正常功能是非常重要的(Daniel et al., 2004)。而PRR3、PRR、PRR7、PRR9以及XCT甚至CKB4自身都受到磷酸化来调控它们在体内的稳定甚至降解途径(Perales et al., 2006; Ding et al., 2007; Farre and Kay, 2007; Ito et al., 2007; Fujiwara et al., 2008)。
ZTL介导的蛋白质降解作用在生物钟的转录后调控中起着重要的作用。ZTL可以与TOC1和PRR5结合,以介导它们在体内的降解作用。由于在ztl-1突变株系中不存在TOC1与ZTL的相互作用,TOC1蛋白可以持续地进行表达(Kim et al., 2003)。TOC1蛋白的降解主要发生在夜晚,并且需要ZTL的参与。GI在是这个过程中重要的一员,在蓝光条件下,ZTL与GI的结合比较稳定,而在无光条件下,ZTL,GI,TOC1,PRR5的讲解速度都快于有光条件下的速度。PRR3可以与TOC1结合,以保护TOC1免受ZTL介导的蛋白降解作用(Para et al., 2007)。TOC1和ZTL的相互作用对维持TOC1蛋白的稳定性有着十分重要的作用,在黎明时,当ZTL蛋白的表达量达到一定的值时,就会很快地被降解掉(Kim et al., 2003)。
5生物钟控制的输出途径中的基因表达
对受到生物钟控制的基因的表达进行全局的分析,可以有效地鉴定生物钟系统中的新成员,并且可以发现在相同的生理代谢途径中一起表达的基因。核苷酸微阵列的应用为拟南芥生物钟调控的转录网络提供了一个全局的视角(Harmer et al.,2000; Schaffer et al., 2001)。
Covington(2008)通过整合多个微阵列的实验,来从整体上研究拟南芥体内受生物钟调控的相关基因。通过对生物钟相关基因的启动子区域的分析,Covington发现了与生物钟相关基因在在4个不同时间阶段表达相关的启动子元件,分别是夜间元件EE (evening element, AAAATATCT)、CBS (CCA1- binding site, AAAAAATCT)、ME (morning element,AACCACGAAAAT)、PBX (protein box element,ATGGGCC)。并发现了一些受生物钟调控比较明显的生理途径。Covington的研究表明,拟南芥体内受生物钟调控而表达的基因占所有表达基因的三分之一左右,同时在激素和各种胁迫相关的生理途径中大量基因的表达都受到生物钟的调控(Covington et al., 2008)。
6展望
生物钟几乎参与植物体所有的新陈代谢、生长发育的调控过程,在绝大多数的信号转导途径中起着重要的作用,使植物体自身的内源节律与与外界环境达到时间和空间的同步,极大地增强了植物环境适应性和竞争能力(徐小冬和谢启光, 2013)。生物钟的研究必定可以在农业的应用上提供很好的理论依据,有利于提高作物的适应性、抗逆性和提高作物的产量等。不过就目前我们所知的植物的生物钟基因调控网络还不完善,还有很多未知的环节、关键基因等待我们去挖掘、鉴定。
作者贡献
李宗飞是本研究的执行人,负责文献收集及文章初稿写作;张洁、刘振鹏参与文献调研、初稿的形成和修改;方宣钧指导论文的写作及修改。全体作者阅读并同意最终的文本。
致谢
本研究由海南省热带农业资源开发利用研究所开放研究基金项目资助。
Alabadí D., Oyama T., Yanovsky M.J., Harmon F.G., Más P., Kay S.A., 2001, Reciprocal regulation between TOC1 and LHY/CCA1 within the Arabidopsis circadian clock, Science, 293(5531): 880-883
http://dx.doi.org/10.1126/science.1061320
Allen T., Koustenis A., Theodorou G., Somers D.E., Kay S.A., Whitelam G.C., and Devlin P.F., 2006, Arabidopsis FHY3 specifically gates phytochrome signaling to the circadian clock, Plant Cell, 18(10): 2506-2516
http://dx.doi.org/10.1105/tpc.105.037358
Barak S., Tobin E.M., Andronis C., Sugano S., and Green R.M., 2000, All in good time: the Arabidopsis circadian clock, Trends Plant Sci, 5: 517-522
http://dx.doi.org/10.1016/S1360-1385(00)01785-4
Blasing O.E., Gibon Y., Gunther M., Hohne M., Morcuende R., Osuna D., Thimm O., Usadel B., Scheible W.R., and Stitt M., 2005, Sugars and circadian regulation make major contributions to the global regulation of diurnal gene expression in Arabidopsis, Plant Cell, 17(12): 3257–3281
http://dx.doi.org/10.1105/tpc.105.035261
Covington M.F., Maloof J.N., Straume M., and Kay S.A., 2008, Harmer SL.Global transcriptome analysis reveals circadian regulation of key pathways in plant growth and development, Genome Biol, 29(8): R130
http://dx.doi.org/10.1186/gb-2008-9-8-r130
Craing K.L., and Tyers M., 1999, The F-box: a new motif for ubiquitin dependent proteolysis in cell cycle regulation and signal transduction, Prog Biophys Mol Biol, 72(3): 299-328
http://dx.doi.org/10.1016/S0079-6107(99)00010-3
Daniel X., Sugano S., and Tobin E.M., 2004, CK2 phosphorylation of CCA1 is necessary for its circadian oscillator function in Arabidopsis, Proc Natl Acad Sci U S A, 101(9): 3292-3297
http://dx.doi.org/10.1073/pnas.0400163101
Devlin P.F., and Kay S.A., 2000, Cryptochromes are required for phytochrome signaling to the circadian clock but not for rhythmicity, Plant Cell, 12: 2499-2510
http://dx.doi.org/10.1105/tpc.12.12.2499
Devlin P.F., and Kay S.A., 2001, Circadian photoperception, Annu Rev Physiol, 63: 677-694
http://dx.doi.org/10.1146/annurev.physiol.63.1.677
Ding Z., Millar A.J., Davis A.M., Davis S.J., 2007, TIME FOR COFFEE encodes a nuclear regulator in the Arabidopsis thaliana circadian clock, Plant Cell, 19(5): 1522-1536
http://dx.doi.org/10.1105/tpc.106.047241
Dodd A.N., Salathia N., Hall A., Kevei E., Toth R., Nagy F., Hibberd J.M., Millar A.J., and Webb A.A., 2005, Plant circadian clocks increase photosynthesis, growth, survival, and competitive advantage, Science, 309(5734): 630–633
http://dx.doi.org/10.1126/science.1115581
Doyle M.R., Davis S.J., Bastow R.M., Mcwatter H.G., Kozma-Bognar L., Nagy F., Millar A.J., and Amasino R.M., 2002, The ELF4 gene controls circadian rhythms and flowering time in Arabidopsis thaliana, Nature, 419(6902): 74-77
http://dx.doi.org/10.1038/nature00954
Dunlap J.C., 1999, Molecular bases for circadian clock, Cell, 96: 271-290
http://dx.doi.org/10.1016/S0092-8674(00)80566-8
Dunlap J.C., 2004, Kinases and circadian clocks: Per goes it alone, 6(2): 160-161
Edery I., 2005, Role of posttranscriptional regulation in circadian clocks: Lessons from Drosophila, Chronobiol Int, 16(4): 377-414
http://dx.doi.org/10.3109/07420529908998716
Farre E.M., and Kay S.A., 2007, PRR7 protein levels are regulated by light and the circadian clock in Arabidopsis, Plant J, 52(3): 548–560
http://dx.doi.org/10.1111/j.1365-313X.2007.03258.x
Fowler S., Lee K., Onouchi H., Samach A, Richardson K., Morris B., Coupland G., and Putterill J., 1999, Gigantea : a circadian clock-controlled gene that regulates photoperiodic flowering in Arabidopsis and encodes a protein with several possible membrane-spanning domains, EMBO J, 18(17): 4679-4688
http://dx.doi.org/10.1093/emboj/18.17.4679
Fujiwara S., Wang L., Han L., Suh S., Salome P.A., McClung C.R., and Somers D.E., 2008, Post-translational regulation of the Arabidopsis circadian clock through selective proteolysis and phosphorylation of pseudo-response regulator proteins, J Biol Chem, 283(34): 23073-23083
http://dx.doi.org/10.1074/jbc.M803471200
Green R.M., and Tobin E.M., 1999, Loss of the circadian clock-associated protein 1 in Arabidopsis results in altered clock-regulated gene expression, Proc Natl Acad Sci U S A, 96(7), 4176–4179
http://dx.doi.org/10.1073/pnas.96.7.4176
Green R.M., Tingay S., Wang Z.Y., and Tobin E.M., 2002, Circadian rhythms confer a higher level of fitness to Arabidopsis plants, Plant Physiol, 129(2): 576-584
http://dx.doi.org/10.1104/pp.004374
Harmer S.L., Hogenesch J.B., Straume M., Chang H., Han B., Zhu T., Wang X., Kreps J.A., and Kay S.A., 2000, Orchestrated transcription of key pathways in Arabidopsis by the circadian clock, Science, 290: 2110-2113
http://dx.doi.org/10.1126/science.290.5499.2110
Imaizumi T., Tran H.G., Swartz T.E., Briggs W.R., and Kay S.A., 2003, FKF1 is essential for photoperiodic-specific light signaling in Arabidopsis, Nature, 426(6964): 302-306
http://dx.doi.org/10.1038/nature02090
Ito S., Nakamichi N., Kiba T., Yamashino T., and Mizuno T., 2007, Rhythmic and light-inducible appearance of clock-associated pseudoresponse regulator protein PRR9 through programmed degradation in the dark in Arabidopsis thaliana, Plant Cell Physiol, 48(11): 1644-1651
http://dx.doi.org/10.1093/pcp/pcm122
Jarillo J.A., Capel J., Tang R.H., Yang H.Q., Alonso J.M., Ecker J.R., and Cashmore A.R., 2001, An Arabidopsis circadian clock component interacts with both CRY1 and PHYB, Nature, 410(6827): 487-490
http://dx.doi.org/10.1038/35068589
Johnson C.H., 2001, Endogenous time keepers in photosynthetic organisms, Annu Rev Physiol, 63: 695-728
http://dx.doi.org/10.1146/annurev.physiol.63.1.695
Kim W.Y, Geng R., and Somers D.E., 2003, Circadian phase-specific degradation of the F-box protein ZTL is mediated by the proteasome, Proc Natl Acadm Sci U S A, 100(8): 4933-4938
http://dx.doi.org/10.1073/pnas.0736949100
Kreps J.A., and Kay S.A., 1999, Coordination of plant metabolism and development by the circadian clock, Plant Cell, 9(7): 1235–1244
http://dx.doi.org/10.1105/tpc.9.7.1235
Kreps J.A., Wu Y., Chang H.S., Zhu T., Wang X., and Harper J.F., 2002, Transcriptome changes for Arabidopsis in response to salt, osmotic, and cold stress, Plant Physiol, 130: 2129–2141
http://dx.doi.org/10.1104/pp.008532
Lin C., Yang H., Guo H., Mockler T., Chen J., and Cashmore A.R., 1998, Enhancement of blue-light sensitivity of Arabidopsis seedlings by a blue light receptor cryptochrome 2, Proc Natl Acad Sci USA, 95(5): 2686–2690
http://dx.doi.org/10.1073/pnas.95.5.2686
Li Z.F., Zhuo W., Liu Z.P., and Fang X.J., 2015, Effects of Gene Regulation of Circadian Clock on Plant Growth and Development, Douke Jiyinzuxue Yu Yichuanxue (online) (Legume Genomics and Genetics), 6(1): 1-4 (李宗飞, 周味, 刘振鹏, 方宣钧, 2015, 生物钟的基因调控对植物生长发育的影响, 豆科基因组学与遗传学(online), 6(1): 1-4)
Makino S., Kiba T., Imamura A., Hanaki N., Nakamura A., Suzuki T., Taniguchi M., Ueguchi C., Sugiyama T., and Mizuno T., 2000, Genes encoding pseudoresponse regulators: insight into His-to-Asp phosphorelay and circadian rhythm in Arabidopsis thaliana, Plant Cell Physiol, 41(6): 791–803
http://dx.doi.org/10.1093/pcp/41.6.791
Mariko S., Nusinow D.A., Kay S.A., and Imaizumi T., 2007, FKF1 and GIGANTEA complex formation is required for day-length measurement in Arabidopsis, Science, 318(5848): 261-265
http://dx.doi.org/10.1126/science.1146994
Martin-Tryon E.L., and Stacey L.H., 2008, XAP5 CIRCADIAN TIMEKEEPER coordinates light signals for roper timing of photomorphogenesis and the circadian clock in Arabidopsis, Plant Cell, 20(5): 1244–1259
http://dx.doi.org/10.1105/tpc.107.056655
Más P., 2005, Circadian clock signaling in Arabidopsis thaliana: from gene expression to physiology and development, Int J Dev Biol, 49(5-6): 491-500
http://dx.doi.org/10.1387/ijdb.041968pm
Más P., Devlin P.F., Panda S., and Kay S.A., 2000, Functional interaction of phytochrome B and cryptochrome 2, Nature, 408(6809): 207-211
http://dx.doi.org/10.1038/35041583
Matsushika A., Makin0 S., Kojima M., and Mizuno T., 2000, Circadian waves of expression of the APRR1/TOC1 family of pseudo-response regulators in Arabidopsis thaliana : insight into the plant circadian clock, Plant Cell Physiol, 41(9): 1002-1012
http://dx.doi.org/10.1093/pcp/pcd043
Mcclung C.R., 2001, Circadian rhythms in plants, Annual Review of Plant Biology, 52(52), 139-162
http://dx.doi.org/10.1146/annurev.arplant.52.1.139
McWatters H.G., Bastow R.M., Hall A., and Millar A.J., 2000,The ELF3 zeitnehmer regulates light signalling to the circadian clock, Nature, 408(6813): 716–720
http://dx.doi.org/10.1038/35047079
Mizuno T., 2004, Plant response regulators implicated in signal transduction and circadian rhythm, Curr Opin Plant Biol, 7(5): 499-505
http://dx.doi.org/10.1016/j.pbi.2004.07.015
Mizuno T., and Nakamichi N., 2005, Pseudo-response regulators (PRRs) or true oscillator components (TOCs), Plant Cell Physiol, 46(5): 677–685
http://dx.doi.org/10.1093/pcp/pci087
Nakamichi N., Kusano M., Fukushima A., Kita M., Ito S., Yamashino T., Saito K., Sakakibara H., and Mizuno T., 2009, Transcript profiling of an arabidopsis pseudo response regulator arrhythmic triple mutant reveals a role for the circadian clock in cold stress response. Plant Cell Physiology, 56(1), 71-76
http://dx.doi.org/10.1093/pcp/pcp004
Niwa Y., Yamashino T., and Mizuno T., 2009, The circadian clock regulates the photoperiodic response of hypocotyl elongation through a coincidence mechanism in Arabidopsis thaliana, Plant and Cell Physiology, 50(4): 838-854
http://dx.doi.org/10.1093/pcp/pcp028
Onai K., and Ishiura M., 2005, PHYTOCLOCK 1 encoding a novel GARP protein essential for the Arabidopsis circadian clock, Genes Cells, 10(10): 963-972
http://dx.doi.org/10.1111/j.1365-2443.2005.00892.x
Para A., Farre E.M., Imaizumi T., Pruneda-Paz J.L., Harmon F.G., and Kay S.A., 2007, PRR3 Is a vascular regulator of TOC1 stability in the Arabidopsis circadian clock. Plant Cell 19(11): 3462–3473
http://dx.doi.org/10.1105/tpc.107.054775
Perales M., Portoles S., and Mas P., 2006, The proteasome-dependent degradation of CKB4 is regulated by the Arabidopsis biological clock, Plant J, 46(5): 849–860
http://dx.doi.org/10.1111/j.1365-313X.2006.02744.x
Pruneda-Paz J.L., Breton G., Para A., and Kay S.A., 2009, A functional genomics approach reveals CHE as a component of the Arabidopsis circadian clock ,Science, 323(5920): 1481-14855
http://dx.doi.org/10.1126/science.1167206
Salome´ P.A., and McClung C.R., 2005, Pseudo-response regulator 7 and 9 are partially redundant genes essential for the temperature responsiveness of the Arabidopsis circadian clock, Plant Cell, 17(3): 791–803
http://dx.doi.org/10.1105/tpc.104.029504
Salter M.G., Franklin K.A., and Whitelam G.C., 2003, Gating of the rapid shade-avoidance response by the circadian clock in plants, Nature, 426: 680–683
http://dx.doi.org/10.1038/nature02174
Schaffer R., Landgraf J., Accerbi M., Simon V., Larson M., and Wisman E., 2001, Microarray analysis of diurnal and circadian-regulated genes in Arabidopsis, Plant Cell, 13(1): 113-123
http://dx.doi.org/10.1105/tpc.13.1.113
Schultz T.F., Kiyosue T., Yanovsky M., Wada M., and Kay S.A., 2001, A role for LKP2 in the circadian clock of Arabidopsis, Plant Cell, 13: 2659-2670
http://dx.doi.org/10.1105/tpc.13.12.2659
Sheen X.L., Stephen M.K., Christos A., May S.O., and Elaine M., 2009, Circadian clock associated 1 and late elongated hypocotyl function synergistically in the circadian clock of Arabidopsis, Plant Physiology, 150(2): 834-843
http://dx.doi.org/10.1104/pp.108.133272
Somer D.E., Schultz T.F., Milnamow M., and Kay S.A., 2000, ZEITLUPE encodes a novel clock-associated PAS protein from Arabidopsis, Cell, 101(3): 319-329
http://dx.doi.org/10.1016/S0092-8674(00)80841-7
Somers D.E., Devlin P.F., and Kay S.A., 1998, Phytochromes and cryptochromes in the entrainment of the Arabidopsis circadian clock, Science, 282: 1488-1490
http://dx.doi.org/10.1126/science.282.5393.1488
Staiger D., Allenbach L., Salathia N., Fiechter V., Davis S.J., Millar A.J., Chory J., Fankhauser C., 2003, The Arabidopsis SRR1 gene mediates phyB signalling and is required for normal circadian clock function, Genes Dev, 17(2): 256-268
http://dx.doi.org/10.1101/gad.244103
Strayer C.A., Oyama T., Schultz T.F., Raman R., Somers D.E., Más P., Panda S., Kreps J.A., and Kay S.A., 2000, Cloning of the Arabidopsis clock gene TOC1, an autoregulatory response regulator homolog, Science, 289(5480): 768-771
http://dx.doi.org/10.1126/science.289.5480.768
Toh K.L., Jones C.R., He Y., Eide E.J., Hinz W.A., Virshup D.M., Ptacek L.J., and Fu Y.H., 2001, An hPER2 phosphorylation site mutation in familial advanced sleep phase syndrome, Science, 291(5506): 1040-1043
http://dx.doi.org/10.1126/science.1057499
Toth R., Kevei E., Hall A., Millar A.J., Nagy F., and Kozma-Bognar L., 2001, Circadian clock-regulated expression of phytochrome and cryptochrome genes in Arabidopsis, Plant Physiology, December, 127(4): 1607-1616
Wang Z.Y., and Tobin E.M., 1998, Constitutive expression of the circadian clock associated 1 (CCA1) gene disrupts circadian rhythms and suppresses its own expression, Cell, 93(7): 1207–1217
http://dx.doi.org/10.1016/S0092-8674(00)81464-6
Xu X.D., and Xie Q.G., 2013, The circadian clock in plants, Ziran Zazhi (Chinese Journal of Nature), 35(2): 118-126 (徐小冬, 谢启光, 2013, 植物生物钟研究的历史回顾与最新进展, 自然杂志, 35(2): 118-126)
Young M.W., and Kay S.A., 2001, Time zones: a comparative genetics of circadian clocks, Nat. Rev. Gen., 2: 702-715
http://dx.doi.org/10.1038/35088576
Zeilinger M.N., Farre E.M., Taylor S.R., Kay S.A., and Doyle F.J., 2006, A novel computational model of the circadian clock in Arabidopsis that incorporates PRR7 and PRR9, Mol Syst Biol, 2(1): 58