 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 37 篇 doi: 10.5376/mpb.cn.2012.10.0037
收稿日期: 2012年05月24日 接受日期: 2012年06月06日 发表日期: 2012年07月31日
引用格式(中文):
张春宵等, 2012, 78份高粱种质的遗传关系和遗传结构解析, 分子植物育种(online) Vol.10 No.37 pp.1265-1277 (doi: 10.5376/mpb.cn.2012.10.0037)
引用格式(英文):
Zhang et al., 2012, Analysis of Genetic Relationship and Genetic Structure Among 78 Sorghum Accessions, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.37 pp.1265-1277 (doi: 10.5376/mpb.cn.2012.10.0037)
本研究利用筛选确立的41对SSR引物对78份高粱材料(常规种和地方品种)进行遗传关系和遗传结构分析,并对遗传距离聚类和模型聚类方法进行比较。结果表明:模型聚类法更适用于高粱的遗传关系研究。根据模型聚类的结果,78份高粱材料可归属为Ⅰ、Ⅱ、Ⅲ和Ⅳ共4个类群。第Ⅱ类群与第Ⅰ类群、第Ⅳ类群都有明显的重叠区域,说明该群中有部分材料含有其它类群的遗传成分。第Ⅰ类群和第Ⅱ类群间的遗传关系最为接近,与第Ⅳ类群的遗传关系最远。根据各群间遗传关系的远近,4个群可以简化为A (Ⅰ群, Ⅱ群)、B (Ⅲ群)、C (Ⅳ群)3个类群,也可以进一步简化为A (Ⅰ, Ⅱ, Ⅲ群)、B (Ⅳ群)2个类群。本研究结果对融合多种高粱优良基因的基础群体的构建,对现存的高粱杂种优势模式的简化,有目标的高效定向高粱育种提供了理论依据。
高粱在中国分布极广,几乎中国各地均有种植,但主产区却主要集中在秦岭、黄河以北,特别是长城以北。由于高粱栽培区的气候、土壤、栽培制度的不同,栽培品种的多样性特点也不一样,故高粱的分布与生产带有明显的区域性,中国分为4个栽培区:春播早熟区、春播晚熟区、春、夏兼播区和南方区。
目前,生产上所应用的亲本系大多属于中国高粱和印度高粱,高粱种质的遗传基础较为狭窄,高粱可利用的育种材料贫乏,杂种优势的利用模式陈旧,最终导致杂交育种难度大。因此,应该利用现代的分子生物学技术研究不同生态型,对现有的种质加以改良和利用(高士杰, 2006, 作物杂志, 3: 11-13)。基于高粱种质类群的遗传多样性及其类群间遗传关系的研究,不仅可以对高粱种质进行改良,确定杂种优势的模式,对其育种效率的提高也具有积极意义(吴承来等, 2010)。
分子标记已被证明在遗传关系分析上是一种强有力的工具。与其它的遗传标记相比,SSR标记具有均匀的基因组覆盖、高水平的多态性和共显性遗传模式(Tautz, 1989),因此,被广泛应用于玉米、高粱等各类农作物品种间遗传差异和多样性研究(吴承来等, 2010; 余传涨等, 2010)。依据SSR分子标记进行种质聚类时,主要有距离聚类法和模型聚类法。距离聚类法主要是基于遗传距离,通过NJ、ME或是UPGMA法进行聚类,这种方法已经广泛用于高粱、玉米等农作物的遗传关系研究(余传涨等, 2010; 赵香娜等, 2010; 孙友位, 2007)。而模型聚类法则是通过设置参数模型运用标准算法(如最大似然法, 贝叶斯法等),对个体的归属群进行推断(吴承来等, 2010)。Pritchard等(2000)通过模型聚类法(STRUCTURE软件)分析群体遗传结构。此法可以由供试材料的遗传成分进行聚类。
前人已对高粱种质间的遗传关系进行了很多研究,但是同时使用距离聚类和模型聚类两种方法进行遗传关系分析及化简杂种优势群和杂种优势模式的报道目前尚无。本研究采用SSR标记并利用上述两种聚类方法,对78份(常规种, 地方品种)高粱分别进行聚类分析,探究高粱试材的遗传结构及其类群间的遗传关系,试验结果将为高粱杂种优势群的划分和简化,有目的性的改良高粱种质,进一步提高杂交种选育效率提供理论依据。
1结果分析
1.1 78份高粱材料的遗传关系和遗传结构
1.1.1 UPGMA聚类
利用41对SSR引物共标记出193个等位基因,利用这193个等位基因对78份材料(42份常规品种和36份地方品种)进行GS中心化聚类分析,供试材料可以划分为2个类群(图1),A类群有2个亚群,B类群有2个亚群(表1)。同时表明,A类群的第Ⅱ亚群与B类群的第Ⅱ亚群种质间的遗传进化关系接近。
 图1 根据遗传相似矩阵(中心化)对42份常规种和36份地方品种的UPGMA聚类 Figure 1 Association among 78 sorghum materials including 42 conventional cultivars and landraces revealed by UPGMA clustering analysis ansed on the centered genetic similarity coefficientsgenerated by 41 pairs of SSR |
|
表1 78份高粱材料的UPGMA聚类结果
|
1.1.2基于N-J、ME和UPGMA法进化树分析
根据Nei遗传距离(GD)对78份高粱材料进行系统发育树的重建(图2)。NJ、ME和UPGMA法进化树构建的结果表明,可以将78份高粱材料可以划分为2个种质类群,种质类群划分结果与UPGMA聚类基本一致。
|
图2 进化树分析
|
1.1.3模型聚类和遗传结构分析
78份高粱材料的混合模型遗传结构分析结果表明,类群数K,由似然值可以推测出K>3,由于当K=3时,似然值明显升高,而且缺少了个别亚群,说明亚群数可能大于3。而K>4时的似然值,较K=4的似然值有极小的增长。当K取不同值(2~10)时,Ln(P)随着K值的增大而连续升高,当K=4时,变化幅度趋于平缓(图3-A),并取得较稳定的α值(图3-B)。结合以上划分结果,可以得出K取值为4是合理的,即78份高粱材料划分为4个类群。与UPGMA、系统发育树重建方法划分结果也基本吻合。
|
图3取不同K值时的Ln(P), △K、α值的变化情况
|
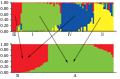
当K=4时,将78份高粱材料分为地方品种(Ⅰ)、地方品种衍生的自交系(Ⅱ)、新疆品种(Ⅲ)、国外品种(Ⅳ)共4个亚类(图4)。
|
图4 基于SSR标记的78份高粱材料的遗传结构比较(K=4变为K=2)
|
若78份高粱材料适合划分为较少的潜在类群,也可以应用STRUCTURE软件对较少的类群数(K=2, 3)进行聚类,最适合似然值出现在K=3时,同时也说明K=4与K=3时的似然值相比,K=4时的似然值更为适合,因此说明78份高粱材料的确包含4个亚群。当K=2被允许时,78份高粱材料主要是依据地理起源进行聚类的。将Ⅰ、Ⅱ合为A类群,Ⅲ、Ⅳ合为B类群(图4)。A类群为地方品种及其衍生的自交系,B类群为国外品种及亲本具有国外品种的新疆品种。如第Ⅱ亚群的0-30是(分枝大红穗×晋粱五)×4003后选育的自交系,很显然与第Ⅰ亚群的晋梁5号具有极相近的血缘关系。第Ⅱ亚群的铁恢208为6060×(铁紧穗×晋57-1)后选育的自交系,显然铁紧穗为地方品种,因此铁恢208与地方品种具有极相近的血缘关系。又如第Ⅱ亚群的三尺三为山西地方品种选育的自交系,辽恢115为辽宁自选系,铁恢157为铁岭自选系等,根据以上示例表明,第Ⅱ亚群与第Ⅰ亚群具有极相近的血缘关系,因此将这两个亚群归为A类群。
第Ⅲ亚群的新疆品种虽是属于中国的地方品种,但是中国新疆的高粱最初是由国外引入的,与第Ⅳ亚群的国外品种具有很近的亲缘关系。因此将其分为一类。其结果与STRUCTURE软件得到各群的无根进化树(图5)的结果一致。黄色、绿色部分为一类,红色、蓝色为一类。
|
图5 模型聚类的各群的无根进化树
|
由STRUCTURE软件得到的Q值(表2)可以看出各高粱材料不同亚群遗传成分多少。根据最大Q值分布(表3),78份高粱材料中有6份材料小于0.6,占总材料数的7.7%,;Q值大于0.8和0.9的材料占74.4和60.3,说明各群中有部分材料含有其他类群的遗传成分,其中晋福1号含有48.8%的Ⅰ群种质和50.3%的Ⅲ群种质,三尺三含有48.6%的Ⅰ群种质和49.7%的Ⅳ群种质,传种高粱含有38.6%的Ⅱ群种质和33.8%的Ⅳ群种质,铁恢6号含有55.1%的Ⅰ群种质和43.6%的Ⅲ群种质。
|
表2 42份常规种和36份地方品种及其Q值
|
|
表3 模型聚类的各群材料最大Q值分布
|
1.2高粱各类群的遗传多样性和类群间遗传关系分析
1.2.1各类群的遗传多样性分析
分析表明(表4),Ⅳ群在等位变异数、有效等位变异数、Shannon-Weaver多样性指数和期望杂合度四个方面在四个亚群中均属最高,而遗传固定指数相对其他亚群较高,说明该群的遗传多样性高,遗传分化程度较明显。Ⅰ类群的遗传多样性较高,但其遗传分化程度最明显(0.763 1)。Ⅱ类的遗传多样性及遗传分化程度均较高。Ⅲ类群的等位变异数、有效等位变异数、Shannon-Weaver指数在四个亚群中最低,遗传固定指数也最低,这说明Ⅲ类群具有遗传多样性低、遗传分化程度低、遗传特点不明显等特点。
|
表4 模型聚类的各类群遗传多样性
|
1.2.2各类群的遗传关系
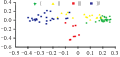
由表5可以看出,各类群间的遗传相似系数均较低。相比之下,第Ⅰ类群和第Ⅱ类群间的遗传相似系数最大(0.300 7)。表明四个类群间,第Ⅰ类群和第Ⅱ类群的遗传关系最为接近,与第Ⅳ类群的遗传相似系数最小,表明第Ⅰ类群与第Ⅳ类群的遗传关系最远。第Ⅱ类群与第Ⅳ类群的遗传相似系数较大(0.294 9)。从图6可以看出,第Ⅱ类群与第Ⅰ类群、第Ⅳ类群都有明显的重叠区域,说明该群中有部分材料含有其他类群的遗传成分。
表5 类群见的遗传相似系数(对角线上方)和遗传距离(对角线下方) Table 5 Nei’s genetic identity (above diagonal) and genetic distance (below diagonal) between groups |
|
图6 基于模型聚类的78份高粱材料遗传成分(Q值)的PCO分析
|
由表6可以看出,第Ⅱ类群与第Ⅰ类群、第Ⅳ类群间的基因流均较大,其中第Ⅱ类群与第Ⅰ类群间的基因流最大(1.488 3),第Ⅱ类群与第Ⅲ类群间的基因流最小(0.790 7)。该结果与各类群间的遗传相似系数大小关系相吻合,这一点说明造成高粱各类群间遗传关系远近的主要原因可能是基因流。
|
表6 模型聚类的各类群间基因流
|
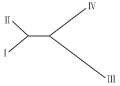
为进一步明确各类群间的遗传关系,根据类群间的Nei's的遗传距离对模型聚类的4个类群进行无根树状遗传关系聚类分析(图7)。当该群体划分为3类时,第Ⅰ类群和第Ⅱ类群首先聚为一类;若划分为2类,第Ⅰ、Ⅱ、Ⅲ群适宜合并为一类,而第Ⅳ群单独成为一个群。
|
图7 模型聚类的4个类群的聚类关系
|
2讨论
2.1不同聚类结果的比较和类群化简
种质聚类方法主要有遗传距离聚类法和模型聚类法两种。(陈红菊等, 2004)。
基于遗传距离的聚类方法已广泛应用于高粱遗传关系分析,农艺性状近似的聚在同一个类群内(赵香娜等, 2010)。余传涨等(2010)等利用52对SSR引物对41个高粱材料进行遗传多样性分析,通过距离聚类结果将41个高粱材料分为2个类群(籽粒高粱, 甜高粱)。
用于群体遗传结构研究时,模型聚类STRUCTURE软件需要设定群体的分群数(K值) (吴承来等, 2010)。一般K值是通过由STRUCTURE软件得到的最高Ln(P)值来确定,估算真正的群体数目(K值)是很困难的(Pritchard et al., 2000)。而在现实情况下,仅有很少的数据可以准确的证实STRUCTURE模型,并且当K值已达到真正的类群数时,Ln(P)值仍然不断增大。因此,Evanno等(2005)等提出△K,当K值达到准确类群数目时,△K的变化是很理想的。孙友位(2007)在利用STRUCTURE软件对375个自交系进行混合模型遗传结构分析时,当K=6时,Ln(P)值增幅趋于平缓,并取得较稳定的α值,而且似然值获得较高的值,因此得出K取值为6。本研究通过距离聚类和模型聚类两种方法,对78份高粱材料进行类群划分,两种方法的类群划分结果大体一致。
确立种质杂种优势模式,并在此基础上化简杂种优势群,可以有目的的进行种质遗传改良及提高杂交种选育效率。关于玉米杂优划分的相关研究较多(吴承来等, 2010),而关于高粱的类群化简尚无相关报道。本研究模型聚类的4个群中,第Ⅰ群和第Ⅱ群的遗传关系最近,第Ⅱ群与第Ⅲ群的遗传关系较近,与第Ⅳ群的遗传关系较远,第Ⅰ群与第Ⅳ群的遗传关系最远。根据各群间遗传关系的远近,4个群可以简化为A (Ⅰ群, Ⅱ群)、B (Ⅲ群)、C (Ⅳ群)3个类群,也可以进一步简化为A (Ⅰ, Ⅱ, Ⅲ群)、B (Ⅳ群)2个类群。
2.2基于SSR标记的高粱种质资源遗传多样性分析
Agrama等(2003)等利用32对RAPD引物和28对高粱SSR引物,对22份高粱材料进行多态性检测,RAPD引物具有较低的多态性并且40%的片段是单态的;而SSR标记具有高度的多态性,每对引物平均检测到4.5个等位变异,基于SSR数据测算的遗传距离与高粱自交系的地理起源、种族分类高度一致,因此说明SSR标记对于评估高粱不同基因型间的遗传相似性是很适用的。Abu Assar等(2005)等利用16个SSR引物对96份高粱材料进行UPGMA聚类分析,将其分为非改良种质和改良种质两大类,与其原始地域、系谱和形态特征等相符,说明高粱材料间存在着强烈的分化。
根据聚类结果,地方品种(Ⅰ)与地方品种衍生的自交系(Ⅱ)遗传距离最接近,与国外品种(Ⅳ)的遗传关系最远。根据基因流的数值,Ⅱ类群与Ⅰ类群、Ⅳ类群(新疆品种)间的基因流均较大,其中Ⅱ类群与Ⅰ类群间的基因流最大,Ⅱ、Ⅲ类群间和Ⅰ、Ⅳ类群间的基因流最小,由此推测,新疆高粱可能由来源于国外,而中国的地方品种可能为中国本土高粱,进而选育出一系列的高粱自交系。因此怀疑中国也是高粱起源地之一。
3材料与方法
3.1供试材料
本实验收集由吉林省农业科学院作物育种研究所、农业部植物新品种测试(公主岭)分中心等单位提供的高粱材料共计78份(表7)。品种类型包括地方品种、常规种(含有国外种质, 下简称F; 包括保持系和恢复系, 下分别简称B和R)和杂交种。
|
表7 78份高梁材料清单
|
本实验所使用的41对核心引物(表8)是经过筛选确立的,其具有均匀分布于高粱染色体,扩增带型清晰、稳定且多态性水平高等特点。引物由北京三博远志公司合成。
|
表8 41对SSR引物清单
|
3.2试验方法
3.2.1 DNA提取
本实验采用CTAB法(余传涨等, 2010)并进行了适当的优化:1、称适量样品,液氮下迅速磨成粉末;2、将粉末转入2.0 ml Eppendorf管中,加入65℃预热的CTAB缓冲液700 µl,并使其混匀;3、65℃水浴加热45 min,不断地轻轻倒转摇动。水浴后,取出离心管,冷却至室温;4、通风橱下加入700 µl的氯仿:异戊醇(24:1),轻轻倒转摇动5~10 min;5、室温下12 000 rpm离心10 min,用去头枪尖将上清转至一新2.0 ml Eppendorf管中;6、加入10 µl RNA酶溶液(10 mg/ml),37℃下温浴30 min;7、重复4~5步骤;8、加入-20℃预冷的异丙醇或无水乙醇于2.0 ml Eppendorf管中,轻轻混匀。-20℃冰箱静置一段时间后,至DNA凝集,室温下钩出DNA;9、76%乙醇洗涤两次,洗涤完毕后,将DNA晾干;10、加入适量的1×TE (PH 8.0)溶解于试管中,4℃下保存备用。取2 μl DNA溶液,用0.8%的琼脂糖凝胶电泳检测。
3.2.2 PCR扩增体系及程序
扩增体系:采用10 µL反应体系,其中含1×PCR Buffer2+ (含2 mmol/L Mg2+),100 µmol/L dNTP,0.24 µmol/L SSR引物,0.4 U Taq DNA聚合酶,20 ng DNA模板,其余用双蒸水补足。
扩增程序:94℃ 10 min,1个循环;94℃ 1 min,55℃ 1 min,72℃ 1 min,共36个循环(根据引物特点可作适当调整);72℃ 10 min,4℃保存。(赵泽双等, 2012)。扩增反应在Bio-Rad公司MyCyclerTM PCR仪上进行。
3.2.3电泳检测
SSR扩增产物在6%变性聚丙烯酰胺凝胶电泳上分离。70W预电泳45 min,70W电泳45 min。银染:将凝胶板置于10%醋酸溶液固定20 min;双蒸水漂洗3 min;在2L新配的染色液(3g AgNO3)中染色20~30 min;双蒸水快速漂洗1次,时间不超过10 s;2L显影液(25 g NaOH+7 ml甲醛溶液)显影;10%醋酸溶液中定影5 min;双蒸水漂洗5 min后置于室温下自然晾干。
3.2.4数据统计分析
SSR扩增产物以0、1和9统计建立数据库。在相同迁移率位置上,有带标记为1,无带标记为0,缺失数据标记为9。采用Popgene1.32软件(Yeh and Yang, 1999)分别计算:(1)总等位变异数(number of alleles, na);(2)有效等位变异数(effective number of alleles, ne);(3)Shannon-Weaver指数(Shannon's information index, I);(4)基因流(gene flow, Nm);(5)Nei期望杂合度(expected heterozygosity, He);(6)多态性信息量(polymorphism information content, PIC)。
计算材料间的遗传相似系数(Genetic similarity, GS)和遗传距离(Genetic distance, GD)采用软件NTSYpce-2.11 (Exeter Software, Setauket, NY)。根据材料间的遗传相似系数矩阵以及中心化(Double-center)后的相似系数矩阵,按UPGMA (Unweighted pair Group Method Arithmetic Average)方法进行聚类分析,构建树状图。中心化数据是指将矩阵X的每一个元素减去相应变量(即列)的平均值所得的矩阵。本研究中所采用NTSYpce-2.11软件中Dcenter模块对GS矩阵进行中心化处理(赵泽双等, 2012)。
用软件Structure2.3.1对常规种群体结构进行分析(Pritchard et al., 2000)。设置“Burnin Period”和“after Burnin”两Structure参数为10 000次,K值1~10,每个K重复10次。画出基于模型的群体遗传结构图,并计算各常规材料的Q值(第i常规材料其基因组变异源于第k群体的概率) (赵泽双等, 2012)。
作者贡献
李晓辉和王凤华负责实验设计和论文修改;杨德光参与实验设计和数据分析;郝彩环和周海涛整理和收集实验材料;张春宵和王晶是本研究的具体执行人,共同开展试验、数据分析和论文写作。全体作者都阅读并同意最终的文本。
致谢
本研究由农业部“DUS测试品种、信息DNA测试技术研究”项目(200903008),“DNA指纹图谱鉴定技术方法研究和数据采集”课题(200903008-07),“高梁DNA指纹图谱鉴定技术标准研制及数据库构建”子课题(200903008-07-08)共同资助。感谢吉林省农业科学院作物育种研究所、农业部植物新品种测试(公主岭)分中心等单位为本研究提供实验材料。感谢匿名的同行评审人的评审建议和修改建议。
参考文献
Abu Assar A.H., Uptmoor R., Abdelmula A.A., Salih M., Ordon F., and Friedt W., 2005, Genetic variation in sorghum germplasm from sudan, ICRISAT and USA assessed by simple sequence repeats (SSRs), Crop Sci., 45: 1636-1644
http://dx.doi.org/10.2135/cropsci2003.0383
Agrama H.A., and Tuinstra M.R., 2003, Phylogenetic diversity and relationships among sorghum accessions using SSRs and RAPDs, Afr. J. Biotechnol., 2(10): 334-340
Chen H.J., Yue Y.S., Fan X.Z., Zhang C.S., and Du L.X., 2004, A Comparative study of genetic distance and clustering analysis among Shandong indigenous chicken breeds, Xumu Shouyi Xuebao (Acta Veterinaria et Zootechnica Sinica), 35(1): 33-36 (陈红菊, 岳永生, 樊新忠, 张传生, 杜立新, 2004, 山东地方鸡种遗传距离与聚类分析方法比较研究, 畜牧兽医学报, 35(1): 33-36)
Evanno G.., Regnaut S., and Goudet J., 2005, Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study, Mol. Ecol., 14: 2611-2620
http://dx.doi.org/10.1111/j.1365-294X.2005.02553.x PMid:15969739
Pritchard J.K., Stephens M., and Donnelly P., 2000, Inference of population structure using multilocus genotype data, Genetics, 155: 945-959
PMid:10835412 PMCid:1461096
Sun Y.W., 2007, Analysis of genetic diversity among maize inbred lines using SSR markers, Thesis for M.S., Chinese academy of agricultural sciences, Supervisor: Zhang S.H., pp.42-44 (孙友位, 2007, 利用SSR标记分析玉米自交系的遗传多样性, 硕士学位论文, 中国农业科学院, 导师: 张世煌, 42-44)
Tautz D., 1989, Hypervariability of simple sequences as a general source for polymorphic DNA markers, Nucl. Acid. Res., 17(16): 6463-6471
http://dx.doi.org/10.1093/nar/17.16.6463 PMid:2780284 PMCid:318341
Wu C.L., Zhang Q.Q., Dong B.X., Li S.F., and Zhang C.Q., 2010, Analysis of genetic structure and genetic relationships of partial maize inbred lines in China, Zuowu Xuebao (Acta Agronomica Sinica), 36(11): 1820-1831 (吴承来, 张倩倩, 董炳雪, 李圣福, 张春庆, 2010, 我国部分玉米自交系遗传关系和遗传结构解析, 作物学报, 36(11): 1820-1831)
Yeh F.C., and Yang R.C., 1999, http://www.ualberta.ca/~fyeh/popgene.pdf
Yu C.Z., Zhai G.W., Zou G.H., Tao Y.Z., and Wang H., 2010, Assessment of genetic diversity among 41 sorghum varieties using SSR marker, Jiangsu Nongye Xuebao (Jiangsu Journal of Agricultural Sciences), 26(2): 248-253 (余传涨, 翟国伟, 邹桂花, 陶跃之, 王华, 2010, 41个高粱品种遗传多样性的SSR标记检测, 江苏农业学报, 26(2): 248-253)
Zhao Z.S., Yang S.H., zhang C.X., Yang D.G., Li X.H., 2012, Analysis of genetic relationships of 69 maize inbred lines in China and screening of effective markers used in three main diseases identification, Yumi Kexue (Journal of Maize Sciences), 20(2): 16-26 (赵泽双, 杨书华, 张春宵, 杨德光, 李晓辉, 2012, 69份玉米自交系的遗传关系分析及三大病害鉴定有效标记的筛选, 玉米科学, 20(2): 16-26)
Zhao X.N., Yue M.Q., Liu Y., Dun B.Q., Zhao W.H., Tan L., and Li G.Y., 2010, Genetic diversity of domestic and foreign sweet sorghum germplasm revealed by SSR markers, Zhiwu Yichuan Ziyuan Xuebao (Journal of Plant Genetic Resources), 11(4): 407-412 (赵香娜, 岳美琪, 刘洋, 顿宝庆, 赵伟华, 谭亮, 李桂英, 2010, 国内外甜高梁种质遗传多样性的SSR分析, 植物遗传资源学报, 11(4): 407-412)

.png)


.png)
.png)

.png)