2. 东北林业大学盐碱地生物资源环境研究中心, 哈尔滨, 150040
3. 海南省农作物分子育种重点实验室, 海南省热带农业资源开发利用研究所, 三亚, 572025
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 23 篇 doi: 10.5376/gab.cn.2011.30.0023
收稿日期: 2011年05月04日 接受日期: 2011年05月11日 发表日期: 2011年05月12日
引用格式(中文):
汪社亮等, 2011,水稻环氧化物酶基因(OsEH1)的克隆及在原核真核中的表达与分析,基因组学与应用生物学(online), Vol.30 No.23 pp.1146-1154 (doi: 10.5376/gab.cn.2011.30.0023)
引用格式(英文):
Wang et al., 2011, Studies on rice epoxide hydrolase: Gene cloning and expressing in E. coli, yeast and arabidopsis, Rice Genomics and Genetics(Online), Vol.1 No.2 (DOI:10.5376/rgg.2010.01.0002)
环氧化物酶(epoxide hydrolase, EH, EC 3.3.2.10),亦称环氧化物水合酶(epoxide hydratase, EC 3.3.2.3),在植物、昆虫、真菌、细菌、酵母和哺乳动物等各种生物中普遍存在。环氧化物酶通过在环醚上加入一个水分子将环氧化物转化成反式二氢二醇化合物,起着多样化的生物学功能。基于水稻基因组序列和环氧化物酶氨基酸序列的保守性,借助全基因组扫描技术已知在水稻基因组中存在不少于10个的环氧化物酶类似蛋白。然而,关于水稻环氧化物酶的基因结构、表达特性以及生物学功能研究甚少。本研究利用反转录PCR从水稻日本晴中克隆该基因,获得一个933 bp的ORF,编码311个氨基酸序列,获得的ORF定位于水稻第十染色体上,BAC注释为一个含有4个内含子分开的5个外显子的mRNA序列转录链。我们将这个基因命名为OsEH1 (Oryza sativa epoxide hydrolase1, 水稻环氧化物酶基因1)。在本研究中,我们构建了原核表达重组质粒,命名为pQE-30-OsEH1,应用热激法将其转入蛋白表达菌株E. coli M15中,诱导表达,蛋白大小约为36 kD。构建了酵母表达pYES2-OsEH1-GFP质粒,确定OsEH1编码蛋白在真核细胞中主要分布于细胞质中。构建了植物表达质粒pBI121-OsEH1质粒,电转化法将其转入农杆菌EHA105中,PCR鉴定获得转OsEH1基因的农杆菌,转化拟南芥获得kana抗性及PCR检测阳性的转OsEH1基因拟南芥。水稻中的Western bloting分析表明,在日本晴幼苗期,OsEH1蛋白水平在叶子中表达比较显著,而在鞘和根中几乎难以检测出表达信号。
环氧化物酶(epoxide hydrolase),亦称环氧化物水合酶(epoxide hydratase),在植物、昆虫、真菌、细菌、酵母和哺乳动物等各种生物中普遍存在(Archelas and Furstoss, 1998; Rehm, 2001)。环氧化物酶通过在环醚上加入一个水分子将环氧化物转化成反式二氢二醇化合物(Bellevik et al., 2002; Blée and Schuber, 1992; Morisseau et al., 2000)。但是,在自然界中环氧化物酶的生物学功能则是非常多样化的,根据其功能可将环氧化物酶分成解毒酶、特定碳源的代谢酶、胁迫响应酶和信号分子调控酶等(Arand et al., 2003; Leung et al., 2003)。在细菌中常常是代谢酶,哺乳动物中则是信号调控及解毒酶,而在植物中则是逆境响应酶(Blée, 2002; Howe and Schilmiller, 2002)。
植物中环氧化物酶的生物学作用尚未完全清晰,环氧化物酶在不同植物中体现的角色及功能不同,本质上应归结于环氧化物酶基因编码的蛋白质的高级结构的特征。马铃薯环氧化物酶是研究得比较详细的植物环氧化物酶之一(Elfström and Widersten, 2006; Mowbray et al., 2006),马铃薯环氧化物酶的表达受发育信号和环境信号的调控,在叶片中表达的环氧化物酶主要与角质层形成有关(Beuerle and Schwab, 1999; Kolattukudy, 2001; Lequeu et al., 2003)。进一步的研究证实环氧化物酶含有高度相似的或保守的氨基酸序列,属于α/β折叠型水解酶类(Summerer et al., 2002)。
环氧衍生物(epoxides),亦称环氧化物,是一类含有环氧化物单元的环醚分子(图1)。环氧化物的基本单元是由两个碳原子和一个氧原子组成的三元杂环,这种等边三角形的杂环使得环醚类环氧化物与其它醚类相比具有更高的活性或应变性。
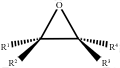 图1 环氧化物的典型结构 |
水稻是人类最重要的粮食作物,也是全基因组已经测序的禾谷类作物的模式植物。在最近的十年来,水稻基因组注释与日俱增。基于水稻基因组序列和环氧化物酶氨基酸序列的保守性,借助全基因组扫描技术已知在水稻基因组中存在不少于10个的环氧化物酶类似蛋白(郑柳成和朱宏波, 2009),这些类似蛋白分布在水稻的不同染色体上,而同一条染色体上的基因都聚集在一起,形成若干基因簇。在染色体分布位置上接近的基因在系统发育树上同样是成簇相聚的。然而,关于水稻环氧化物酶的基因结构、表达特性以及生物学功能研究甚少。
本研究通过同源基因的克隆,在大肠杆菌、酵母和拟南芥中表达,其目的是试图进一步弄清环氧化物酶在水稻中的生物学功能。
1结果与分析
1.1水稻环氧化物酶基因(OsEH1)的克隆及in silico分析
根据已知环氧化物酶的氨基酸序列的保守性,我们设计了一对特异的PCR引物,以水稻日本晴总RNA反转录的cDNA第一链为模板进行RT-PCR扩增。经琼脂糖凝胶电泳鉴定获得一个大小约为950 bp的目标条带(图2A)。构建pMD18-T-OsEH1载体后,转入JM109菌株获得相应pMD18-T-OsEH1质粒,利用限制性内切酶Kpn I和Sal I双酶切该质粒,琼脂糖凝胶电泳鉴定。结果表明除去载体pMD18-T的2.69 kb大小条带外,还出现了大约950 bp大小的一条带,此条带正好符合设计目标基因大小(图2B),送华大生物公司测序获得一个933 bp的ORF,推定编码311个氨基酸序列。
 图2 OsEH1的PCR产物(A)与pMD18-T-OsEH1质粒双酶切(B)琼脂糖凝胶电泳 |
我们对获得的OsEH1的cDNA进行水稻(Oryza sativa)基因组的BlastN分析,结果显示在第10染色体上存在一个NC008403的核酸序列与OsEH1的cDNA有5处高度同源重叠区,其有中4处100%同源,1处有一个碱基差异。因此,我们可以判断获得的基因全序列位于第10染色体上与NC008403序列重叠区域内,精确位置为从NC008403序列的起始端19 446 513 bp处对应包含OsEH1编码基因的1 166 bp末位核酸,在NC008403序列的终端19 448 317 bp处对应包含编码基因的起始第二个碱基,两个核酸序列方向恰好相反。通过BLASTx分析,我们获得的ORF定位于水稻第10染色体上,BAC注释为一个含有4个内含子分开的5个外显子的mRNA序列转录链。我们将这个基因命名为OsEH1 (Oryza sativa Epoxide Hydrolase 1, 水稻环氧化物酶基因1)。ProtParam在线预测分析OsEH1编码蛋白的理论分子量和等电点分别是35 059.4 Da和5.51。在标准的蛋白分型中,SOPMA在线分析结果表明OsEH1编码蛋白含有112个α螺旋,占36%,β转角21个占6.75%,扩展链50占16.08%,随机卷曲128个占41.16%,OsEH1蛋白属于α/β型。
1.2原核表达载体的构建与表达
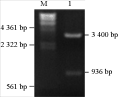
为了在体外表达水稻OsEH1基因,并且尽量不影响基因本来编码的蛋白的各种性质,我们采用了目前常用于蛋白质表达的原核表达载体pQE-30。依据方法中设计的顺序进行载体构建,以前述克隆OsEH1的方法,在引物两端加上特异的限制性内切酶位点进行PCR,在经过pMD18-T载体克隆后,利用T4 DNA连接酶构建重组质粒pQE-30-OsEH1,对获得的阳性重组质粒用Sac I和Kpn I限内切酶酶切鉴定。电泳检测结果表明(图3),获得了预计分子量大小936 bp的目的片段,同时剩余载体片段为3.4 kb,证明获得了正确的重组质粒,命名为pQE-30-OsEH1。应用热激法转入蛋白表达菌株E. coli M15中,诱导表达。
|
|
为了检测融合蛋白在大肠杆菌中的表达情况,首先实验对含有pQE-30-OsEH1的E. coli M15菌株进行了小量的IPTG诱导表达,同时以未加诱导剂的为对照。1 mmol/L IPTG,37℃随诱导时间的不同,表达情况经浓度为12.5%的SDS-PAGE检测(图4)。结果表明,重组质粒pQE-30-OSEH1在IPTG的诱导下产生了重组蛋白6×His-OsEH1,蛋白大小约为36 kD,随着诱导时间的延长,重组蛋白的表达量也分别逐渐增大,并且在2.5 h时达到最大值(图4, 泳道5)。
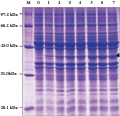 图4 6×His-OsEH1重组蛋白时间梯度SDS-PAGE分析 |
1.3酵母表达载体的构建及融合蛋白在酵母细胞中的定位
将构建好的pYES2-GFP质粒用BamH I和Not I双酶切电泳鉴定,可以看到切下来的片段大约800 bp,符合分析结果(图5A)。同理,我们用Sac I和EcoR I限制性内切酶双酶切pEGFP-OsEH1质粒,切得片段1.7 kb左右,符合正确的目的片段936 bp与800 bp左右的GFP片段融合长度的分析结果(图5B)。以引物2进行PCR扩增获得特定设计位点的1.7 kb的片段(图6A),通过载体的克隆及与pYES2载体的重组,限制性内切酶鉴定获得正确连接的pYES2-OsEH1-GFP质粒(图6B)。根据所设计的引物2,以转化后获得重组质粒pYES2-OsEH1-GFP的酵母菌为模板进行PCR鉴定,实验结果证明待表达质粒已经转入酵母菌中(图7)。
 图5 质粒pYES2-GFP (A)和pEGFP-OsEH1的双酶切琼脂糖凝胶电泳(B) |
 图6 质粒pEGFP-OsEH1的PCR (A)和质粒pYES2-OsEH1-GFP的双酶切琼脂糖凝胶电泳(B) |
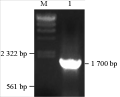 图7 pYES2-OsEH1-GFP酵母菌液PCR琼脂糖凝胶电泳 |
经半乳糖诱导后并制成玻片,在光学显微镜里观察结果(图8)。从图中观察可知:GPF编码蛋白是整个细胞分布,即非特定定位。就单个细胞来看,OsEH1-GFP编码的融合蛋白分布与对照GFP编码的蛋白的分布差别不是很明显(图9),从而可以初步确定OsEH1编码蛋白在真核细胞中主要分布于细胞质中,这个结果与我们利用生物学软件预测OsEH1编码蛋白分布于细胞质中的结果相吻合。
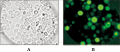 图8 pYES2-OsEH1-GFP重组酵母细胞的显微观察 |
 图9 OsEH1-GFP及GPF的激光共聚焦显微观察 |
1.4植物表达载体的构建与拟南芥转化鉴定
以特异的引物构建好pMD-18-OsEH1质粒,利用限制性内切酶Sma I和Sac I处理pMD-18-OsEH1质粒与植物表达载体pBI121质粒,T4连接酶作用连接,转入克隆菌株JM109中,同样以上述限制性内切酶双酶切鉴定所得pBI121-OsEH1质粒正确(图10A)。此后,将所得质粒电转化法转入农杆菌EHA105中,PCR鉴定转化成功(图10B),获得转OsEH1基因的农杆菌。
在小型塑料皿中放入灭菌适当湿润的营养土,撒入拟南芥种子短日照发芽两周,其后移入塑料钵中独立培养至开花期,移入长日照中培养,期间注意剔除顶端优势。转基因前一天适当进行湿润土壤,进行转基因操作,恢复正常生长管理至种子收获,休眠种子一周即可进行卡那霉素抗性筛选阳性苗。相比于野生型拟南芥,获得转基因阳性苗在kana抗性的培养皿中生长状况良好,植株比野生型茁壮,野生型小苗则发黄发暗(图11)。
对获得的T1代拟南芥阳性苗种子播种,在人工气候箱中正常生长至茁壮期,取各阳性苗叶子进行总基因组提取,并以此为模板,以前述引物和程序进行PCR鉴定。结果显示,图12中有很亮的核酸带,且大小与阳性对照相当,说明部分阳性苗已经成功的转入了OsEH1,同时也有一些阳性苗的测试结果则不明显,基本可以确定为假阳性。至此,我们已经通过PCR鉴定确定获得的阳性苗中有转OsEH1基因成功的拟南芥。
 图10 质粒pBI121-OsEH1的双酶切和E. coli EHA105的PCR琼脂糖凝胶电泳 |
 图11 卡那抗性筛选阳性拟南芥苗 |
图12 转基因拟南芥的PCR琼脂糖凝胶电泳 |
1.5水稻中Western bloting分析
免疫前家兔耳缘静脉采血作为阴性对照。按免疫方案进行免疫,免疫后数天采血测定抗6×His-OsEH1血清的效价。由图13可以看出来两次免疫后血清稀释62 500倍时都能表现明显的效价,说明血清中抗体的生成效果良好,符合实验要求。
 图13 兔抗6×His-OsEH1血清的测定结果 |
首先进行兔二抗预实验,以5 μg、10 μg、15 μg和20 μg的重组蛋白样进行蛋白分析。从western blotting分析的结果(图14)可知,每条泳道均有两条信号带出现,随着蛋白浓度的升高,信号检测出来的背景带愈加明显(泳道上端条带为背景带),但是蛋白与抗体之间的信号强度不因浓度的改变而发生显著差异(各泳道底端条带)。由此说明抗原抗体之间识别响应明显,确定重组蛋白浓度5 μg,二抗浓度1:2000的时候为最佳条件。所获可溶性融合蛋白可进行水稻种组织性OsEH1蛋白表达水平分析。
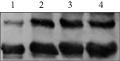 图14 6×His-OsEH1重组蛋白浓度梯度western分析 |
我们以预杂交实验确定的重组蛋白条件为参照(图15, 泳道1),以水稻品种日本晴幼嫩小苗中的叶、鞘、根进行植物活体内特定目标蛋白表达水平定性分析。从图中信号可看出在日本晴幼苗期,OSEH1蛋白水平在叶子中表达比较显著(图15, 泳道2),而在鞘和根中几乎难以检测出可见信号(图15, 泳道3和泳道4)。
图15 水稻中OsEH1蛋白的表达分析 |
2结论
本研究获得的水稻环氧化物酶OsEH1是一个933 bp的ORF编码311个氨基酸序列,定位于水稻第10染色体上,BAC注释为一个含有4个内含子分开的5个外显子的mRNA序列转录链。应用热激法将其转入蛋白表达菌株E. coli M15中,诱导表达,蛋白大小约为36 kD。酵母中表达确定OsEH1编码蛋白在真核细胞中主要分布于细胞质中。通过构建植物表达质粒pBI121-OsEH1,电转化法将其转入农杆菌EHA105中,转化拟南芥获得kana抗性及PCR检测阳性的转OsEH1基因拟南芥。水稻中的Western bloting分析表明,在日本晴幼苗期,OsEH1蛋白水平在叶子中表达比较显著,而在鞘和根中几乎难以检测出可见的表达信号。
3材料与方法
3.1 OsEH1的克隆与in silico定位
依据大豆、马铃薯、烟草等植物环氧化物酶基因的氨基酸保守结构和相似性,我们通过Primer premier5.0软件设计一对引物(正向引物: 5’ATGGATCAGAGGATTGAGCAC-3’; 反向引物: 5’-TCATGCATGTTTACTGACGAAG-3’),利用Biozal RNA提取试剂盒提取水稻粳稻日本晴总RNA,然后进行反转录PCR。将PCR产物连接至pMD-18T载体,转JM109菌株复制克隆(氨苄抗性50 ug/mL),获得pMD18-T-OsEH1质粒,最后利用限制性内切酶KpnI和SalI双酶切鉴定该质粒初步确定正确,送华大生物公司测序。
在NCBI网站的BLASTx (http://blast.ncbi.nlm.nih.gov/Blast.cgi)上进行目的序列及其推导的氨基酸序列与GenBank中其它序列的比对,分析其全长完整性、蛋白质序列同源性性和保守功能结构域。
3.2原核表达载体的构建与表达
水稻环氧化物酶编码的外显子全序列通过多聚酶链式反应(PCR)扩增,程序为:(94°C 2 min, 94°C 40 s, 54.5°C 40 s, 72°C 90 s; 30 cycles)所用引物为:F`:5`-CGGGTACCATGGATCAGAGGATTG-3` (Kpn I) R`:5`-CGGTCGACTCATGCATGTTTACTG-3` (Sal I )。通过pMD18-T载体获得正确的质粒,用相应的限制性内切酶处理质粒和表达载体pQE-30,T4链接酶连接过夜并转入E. coli JM109菌株中获得正确的表达质粒pQE-30-OsEH1。以大肠杆菌E. coli M15为宿主细胞转入含有pQE-30-OsEH1的质粒,用于表达含有6个His标签的融合蛋白。
用IPTG (isopropylβ-D-thiogalactoside)为诱导剂诱导表达转入pQE-30-OsEH1质粒的大肠埃希杆菌E. coli M15。以不加诱导剂为对照,分别于诱导剂加入后0.5 h、1.0 h、1.5 h、2.0 h、2.5 h,、3.0 h和4.0 h取出菌体离心冰浴保存,待所有菌样收集齐全各加等量细胞裂解磷酸盐缓冲液PBS和上样缓冲液,100°C沸水中5 min,快速离心,12.5% SDS-PAGE胶分析蛋白样。
3.3酵母表达载体的构建及酵母细胞中的定位
利用一对特异引物:(F1: 5`-CGGTCGACATGGATCAGAGGATT-3`; R1: 5`-CGGGTACCCATGCATGTTTACT G-3`),经多聚酶链式反应(94°C 2 min, 94°C 40 s, 54.5°C 40 s, 72°C 90 s; 30 cycles)构建含特异限制性内切酶位点(Sal I位点与Kpn I位点)的pMD18-T-OsEH1质粒,采取此两种内切酶及T4连接酶处理将目的基因融合进入质粒pEGFP (CLONTECH)中,获得重组质粒pEGFP-OsEH1。另设计一对引物(F2: 5`-CGGAGCTCAACATGGATCAGAGG-3`;R2: 5`-GTTGGAATTCTAGAGTCGCGGC-3`)进行PCR扩增,程序同上,构建pMD18-T-OsEH1-GFP融合重组质粒,用T4连接酶处理经过限制性内切酶Sac I与EcoR I处理的pMD18-T-OsEH1-GFP质粒回收片段与酵母表达载体pYES2回收片段,转入JM109复制菌株,构建成pYES2-OsEH1-GFP重组质粒。此外,对照pYES2-GFP质粒则直接用限制性内切酶BamH I与Not I处理pEGFP质粒与pYES2质粒,T4连接酶连接处理回收片段,转入JM109复制菌株中获得大量pYES2-GFP酵母表达质粒。
取1 μg重组质粒pYES2-GFP与pYES2-OsEH1-GFP分别加入到酵母菌株INVSc1 (Invitrogen)中,加入100 μg热处理的Carrier DNA,600 μL 1xLiAc/40%PEG/1xTE混匀摇床30 min,70 μL DMSO混匀,42℃热激15 min,瞬时离心,弃上清;1 mL 1XTE轻微重悬细胞,瞬时离心,弃上清,再用100 μL的1xTE轻微重悬细胞,铺在YPD板上,在30℃恒温箱培养1~2 d。挑取单克隆菌于3 mL SC-ural液体培养基中30℃摇床培养,收集200 μL菌液,瞬时离心弃上清,无菌双蒸水轻微重悬沉底,重复3次,SC-ural液体培养基培养,保持半乳糖总浓度2%;30℃摇床培养,制作酵母载破片显微镜下观察结果。
3.4植物表达载体的构建与拟南芥转化鉴定
设计一对特异引物:(F: 5’-TACCCGGGAATGGATCAGAGGATT-3’; R: 5’-CGGAGCTCTCATGCATGTTTACTG-3’) 进行PCR扩增目的基因,程序同上,构建pMD-18-OsEH1质粒,Sma I和Sac I限制性内切酶双酶切处理pMD-18-OsEH1质粒与植物表达载体质粒pBI121,T4连接酶连接回收片段,转入JM109复制菌株中获得大量重组的植物表达质粒pBI121-OsEH1。采用电击法转入农杆菌感受态EHA105菌株中。
采用真空浸入法将含目的表达质粒的农杆菌转入拟南芥花序中,照常管理至收种。初次筛选采用卡那霉素抗性1/2 MS平板培养基筛选,待具有明显生长优势的拟南芥小苗长至两叶以上移栽至无菌营养土中生长。收集T1代种子,利用设计好的特异引物进行PCR鉴定T1代种子生长植株的叶子中目的基因的转录情况。
3.5水稻中Western bloting分析
将经过不连续SDS-PAGE电泳后的蛋白凝胶修剪至适度大小,按三明治夹层方法滤纸、胶、膜和滤纸放在半干转膜仪装置中(amersham pharrnacia),电流适度1 h,将膜用blocking封闭1 h,加入一抗1 h,洗液清洗10 min,加入二抗1 h,洗净膜,加入发光剂CDP-Star,5 min,保鲜膜包裹,ImageMasterVDS-CL (Amersham Pharmacia)成像系统检测。
作者贡献
汪社亮和柳参奎是本研究的实验设计和实验研究的执行人;汪社亮、柳参奎、玉光惠及巩鹏涛完成数据分析,论文初稿的写作;赵德刚参与实验设计和试验结果分析;方宣钧是项目的构思者及负责人,指导实验设计,数据分析及论文写作与修改。全体作者都阅读并同意最终的文本。
基金项目
本研究由国家科技支撑计划(2007BAD59B05)资助
致谢
作者感谢东北林业大学盐碱地资源环境研究中心张欣欣博士在本实验过程中的技术支持和有益的建议。感谢两位匿名的同行评审人的评审建议和修改建议。本文中提到了我们实验中涉及的有关试剂供应商和测序服务商,这并非我们为这些试剂供应商和测序服务商的产品和服务提供推荐或背书。
参考文献
Arand M., Cronin A., Oesch F., Mowbray S.L., and Jones TA., 2003, Thetelltale structures of epoxide hydrolases, Drug Metab. Rev., 35(4): 365-383doi:10.1081/DMR-120026498 PMid:14705866
Archelas A., and Furstoss R., 1998, Epoxide hydrolases: New tools for the synthesis of fine organic chemicals, Trends Biotechnol. 16(3): 108-116 doi:10.1016/S0167-7799(97)01161-X
Bellevik S., Zhang J.M., and Meijer J., 2002, Brassica napus soluble epoxide hydrolase (BNSEH1), Eur. J. Biochem., 269(21): 5295-5302 doi:10.1046/j.1432-1033.2002.03247.x PMid:12392563
Beuerle T., and Schwab W., 1999, Metabolic profile of linoleic acid in stored apples: Formation of 13(R)-hydroxy-9(Z), 11(E)-octadecadienoic acid, Lipids, 34(4): 375-380doi:10.1007/s11745-999-0375-7 PMid:10443970
Blée E., 2002, Impact of phyto-oxylipins in plant defense, Trends Plant Sci., 7(7): 315-322 doi:10.1016/S1360-1385(02)02290-2
Blée E., and Schuber F., 1992, Occurrence of fatty acid epoxide hydrolases in soybean (Glycine max). purification and characterization of the soluble form, Biochem. J., 282(Pt 3): 711-714 PMid:1554352 PMCid:1130845
Elfström L.T., and Widersten M., 2006, Implications for an ionized alkyl-enzyme intermediate during StEH1-catalyzed trans-stilbene oxide hydrolysis, Biochemistry, 45(1): 205-212 doi:10.1021/bi051893g PMid:16388596
Howe G.A., and Schilmiller A.L., 2002, Oxylipin metabolism in response to stress, Curr. Opin. Plant Biol., 5(3): 230-236 doi:10.1016/S1369-5266(02)00250-9
Kolattukudy P.E., 2001, Polyesters in higher plants, Advances in Biochemical Engineering, Biotechnology, 71: 1-49 doi:10.1007/3-540-40021-4_1
Lequeu J., Fauconnier M.L., Chammai A., Bronner R., and Blée E., 2003, Formation of plant cuticle: Evidence for the occurrence of the peroxygenase pathway, Plant J., 36(2): 155-164 doi:10.1046/j.1365-313X.2003.01865.x PMid:14535881
Leung A.K., Andersen J.S., Mann M., and Lamond A.I., 2003, Bioinformatic analysis of the nucleolus, Biochem. J., 376(Pt 3): 553-569 doi:10.1042/BJ20031169 PMid:14531731 PMCid:1223824
Morisseau C., Beetham J.K., Pinot F., Debernard S., Newman J.W., and Hammock B.D., 2000, Cress and potato soluble epoxide hydrolases: Purification, biochemical characterization, and comparison to mammalian enzymes, Arch. Biochem. Biophys., 378(2): 321-332 doi:10.1006/abbi.2000.1810 PMid:10860549
Mowbray S.L., Elfström L.T., Ahlgren K.M., Andersson C.E., and Widersten M., 2006, X-ray structure of potato epoxide hydrolase sheds light on substrate specificity in plant enzymes, Protein Sci., 15(7): 1628-1637 doi:10.1110/ps.051792106 PMid:16751602 PMCid:2265100
Rehm B.H., 2001, Bioinformatic tools for DNA/protein sequence analysis, functional assignment of genes and protein classification, Appl. Microbiol. Bielechnol., 57(5-6): 579-592doi:10.1007/s00253-001-0844-0 PMid:11778865
Summerer S., Hanano A., Utsumi S., Arand M., Schuber F., and Blée E., 2002, Stereochemical features of the hydrolysis of 9,10-epoxystearic acid catalysed by plant and mammalian epoxide hydrolases, Biochem. J., 366(Pt 2): 471-480 doi:10.1042/BJ20011778 PMid:12020347 PMCid:1222791
Zheng L.C., and Zhu H.B., 2009, Bioinformatics analysis on epoxide hydrolase of rice (Oryza saliva), Shengwu Xinxixue (China Journal of Bioinformaties), 7(2): 108-112, 139 (郑柳成, 朱宏波, 2009, 水稻水溶性环氧化合物水解酶的生物信息学分析, 生物信息学, 7(2): 108-112, 139)