1. 东北林业大学, 东北油田盐碱植被恢复与重建教育部重点实验室,盐碱地生物资源环境研究中心, 哈尔滨, 150040
2. 东京大学亚洲生物资源环境研究中心, 东京, 188-0002
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 40 篇 doi: 10.5376/gab.cn.2011.30.0040
收稿日期: 2011年06月29日 接受日期: 2011年07月04日 发表日期: 2011年07月06日
2. 东京大学亚洲生物资源环境研究中心, 东京, 188-0002
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 40 篇 doi: 10.5376/gab.cn.2011.30.0040
收稿日期: 2011年06月29日 接受日期: 2011年07月04日 发表日期: 2011年07月06日
© 2011 BioPublisher 生命科学中文期刊出版平台
这是一篇采用Creative Commons Attribution License进行授权的开放取阅论文。只要对本原作有恰当的引用,版权所有人允许和同意第三方无条件的使用与传播。
推荐引用:
引用格式(中文):
刘畅等, 2011,高等植物中NADP-ME的研究进展,基因组学与应用生物学(online), Vol.30 No.40 pp.1255-1260 (doi: 10.5376/gab.cn.2011.30.0040)
引用格式(英文):
Liu et al., 2011, Research progress of NADP-ME in higher plants, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.40 pp.1255-1260 (doi: 10.5376/gab.cn.2011.30.0040)
摘要
NADP-苹果酸酶(NADP-ME)是广泛存在于植物、动物以及原核生物的一类氧化脱羧酶,可以催化苹果酸氧化脱羧的可逆反应,产生丙酮酸和CO2,以及伴随NAD(P)+的还原反应。参与了多个代谢过程,包括C3植物中生成的还原力NADPH用于木质素、类黄酮等物质的合成、C4植物中的固碳作用等。是生物体生命活动中重要的酶之一。并且在植物的抗逆境生理过程中扮演了重要角色。文章就高等植物NADP-ME的克隆、功能和基因表达调控方面作一介绍。
关键词
植物;NADP-ME;功能;影响因子
苹果酸酶(malic enzyme, ME)是调控苹果酸代谢的关键酶,可以催化苹果酸氧化脱羧的可逆反应,产生丙酮酸和CO2,以及伴随NAD(P)+的还原反应(Chang and Tong, 2003; Detarsio et al., 2004; Bologna et al., 2007)。ME广泛存在于自然界中,包括真菌、细菌、动物和植物(Wynn et al., 1999;Song et al., 2001; Wheeler et al., 2005)。根据辅酶因子的不同,苹果酸酶分为NAD-苹果酸酶(NAD-Malic enzyme, NAD-ME)和NADP-苹果酸酶(NADP-Malic enzyme, NADP-ME),在植物体内已经发现了两种类型的NADP-ME:质体型NADP-ME和胞质型NADP- ME。它们分别存在于植物细胞的质体和细胞质中,发挥着不同的生理功能。除此之外,根据其在植物体内发挥的功能不同,主要可分为光合型和非光合型,其中在C4植物中参与光合途径的光合型NADP-ME研究得最为详尽。它位于C4植物的维管束鞘细胞中,通过其脱羧反应生成的CO2被Rubisco酶重新固定而进入Calvin循环途径。由于苹果酸是代谢过程的中间产物之一,所以NADP-苹果酸酶在植物的生长代谢及发育过程中扮演着重要角色,对保持植物细胞的渗透势,稳定细胞质的pH和保持植物根系的离子吸收平衡起重要作用,是生物体生命活动中重要的酶之一(Martinoia and Rentsch, 1994; Drincovich et al., 2001)。
1高等植物NADP-ME基因克隆
在C4植物中,Rothemel和Nelson (1989)首次报道了在高等植物中NADP-ME基因全长cDNA序列,全长2 184 bp,预测有636个氨基酸残基。根据对预测氮基酸序列分析,在其N-末端含有叶绿体转酶,并且参与了光合作用,被认为是C4植物叶绿体型。此外,另一类质体型NADP-ME基因,研究组分别从玉米、Flaveria.bidentis中分离报道了此类基因,然而对于它们的生理作用却不了解(Maurino et al., 1996)。另外,一个胞质型的NADP-ME从C4植物玉米分离出来,但是对于这个蛋白的生理功能却不是很清楚。最近,Detarsio等(2008)研究小组首次从玉米中克隆出了胞质型的NADP-ME命名为(ZmCytNADP-ME),研究发现这个蛋白的转录水平仅仅表达在胚胎和萌发的幼根中,并且这个重组的ZmCytNADP-ME在动力学参数和代谢调控上明显不同于质体型的NADP-ME。
在C3植物中,多种NADP-ME基因被克隆报道。如豆类(Walter et al., 1990),葡萄(Franke and Adams, 1995)和白杨(van Doorsseleare et al., 1991)等在植物中被分离,其中豆类NADP-ME基因的表达情况研究的最为广泛。有直接证据表明在UV-B情况下可以诱导豆类的NADP-ME的表达(Barkan et al., 2006)。并有许多研究表明NADP-ME参与了植物的防御功能,可能提供NADPH用于木质素和类黄酮的生物合成。另外,质体型NADP-ME也在多种C3植物中被分离,如Flaveria pringlei (Lipka et al., 1994),番茄(GenBank AF001269),葡萄(GenBank U67426),拟南芥(GenBank AC010793)和蓖麻(GenBank AF262997),并且经过序列测定发现都具有公认的质体转酶肽序列。
在CAM (景天科酸代谢途径)植物中,Cushman等(1992)在逆境条件下从冰草(Mesembryanthemun crystallinum)克隆了一个胞质型NADP-ME基因,这个蛋白在高盐和干旱的条件可以从C3光合途径改为CAM代谢途径,并且基因组Southern分析表明冰草还存在其他的NADP-ME基因。后来研究表明冰草中存在两种功能的NADP-ME (Saitou et al., 1994)。另外,Honda等(2000)也从(Aloe arborescens)叶片中克隆得到了一个NADP-ME基因,结果表明这个NADP-ME参与了光和作用,并且这个基因的转录表达水平在根中很低,而在叶中比较高。同时Honda等(1997)通过Southern分析表明AME1是一个多基因家族,并且克隆了第二个全长的cDNA (AME2),两个NADP-ME同源性超过90%,它编码一个胞质型的NADP-ME,并且较低水平组成型的表达在所有器官中。研究表明它也许在非光合成过程中发挥着一些重要的功能。但是这两个NADP-ME同工酶生理功能还不是很清楚。
因此,在高等植物中编码NADP-ME的基因并不是唯一的,而是由多个基因组成的基因家族所编码。目前,在水稻、拟南芥中发现了4个编码该酶的基因,研究表明它们的表达具有组织的特异性,由于NADP-ME广泛的存在于植物体组织中,所以被认为与不同的环境胁迫密切相关(Chi et al., 2004; Wheeler et al., 2005)。
2高等植物NADP-ME的功能
C4植物维管束鞘细胞叶绿体型NADP-ME主要功能是对苹果酸氧化脱羧产生CO2用于Rubisco酶的碳固定,除此之外,报道植物体NADP-ME可能的功能有几个方面。
NADP-ME参与了果实的成熟。有研究表明果实的发育先是有机酸的积累,成熟时将进行快速脱酸。一些气候型水果成熟期时,呼吸作用突然剧烈,糖含量增加、酸含量下降,苹果酸是有机酸之一,并且NADP-ME酶活性显著上升(Krishnamurthy and Patwardhan, 1971; Drouet and Hartmann, 1977; Goodenough et al., 1985)。
另一个早期报道的NADP-ME功能——调节平衡细胞内的pH值。NADP-ME通过苹果酸代谢来平衡细胞内的pH (Martinoia and Rentsch, 1994)。Davies等(1986)认为NADP-ME酶和PEPCA酶(磷酸烯醇式丙酮酸羧化酶)相关联,NADP-ME酶在pH值低的情况下通过对苹果酸脱梭,从而达到脱酸升高细胞内pH值;PEPC酶则在pH值较高情况下通过羧化作用,从而达到酸化降低细胞内pH值。另有报道表明,NADP-ME通过代谢提供碳源和NADPH调节一些底物和辅助因子来参与脂肪酸的合成。
此外,大量的研究事实表明,在纤维素、真菌激发子及机械损伤、UV-B等诱导植物防卫应答的条件处理下,植物胞质型NADP-ME和质体型NADP-ME的表达显著提高(Casati et al., 1999; Fushimi et al., 1994; Cushman et al., 1992)。研究表明NADP-ME通过苹果酸代谢参与植物的防御反应,特别是C3植物NADP-ME,它们参与了植物的防御功能可能在于NADP-ME反应生成的还原力NADPH用于木质素、类黄酮等物质的合成(Drincovich et al., 2001)。此外,NADP-ME在CAM植物的活性调节中起重要作用,研究表明在NaCl、干旱逆境诱导下,NADP-ME酶活性明显增加。然而,NADP-ME与植物的防御关系还没有深入地解释。
3高等植物NADP-ME基因的转录调控及其影响因子
植物生长常常受到环境的影响,了解环境变化对于作物的生长有着重要的意义,由于NADP-ME 广泛的存在于植物体组织中,并且有着不同的代谢途径,目前有很多报道表明NADP-ME与不同的环境胁迫密切相关。对植物生长来说外部影响的非生物胁迫影响因子主要有盐分、温度和干旱。
盐胁迫能够使植物的质膜受到选择性破坏,胞内的大量离子和有机质外渗,从而导致细胞内的生理生化过程受到干扰。有研究表明为了降低盐分对植物的造成伤害,植物本身形成一些生理生化途径来抵御盐分带来的伤害盐信号,从而降低对植物的伤害(Shao et al., 2008; Zhang et al., 2009)。许多研究表明,盐信号能够诱导NADP-ME的表达。Cushman等(1992)研究表明在盐处理下,能够诱导冰草NADP-ME的mRNA转录水平增加8~10。同时研究表明,在盐的诱导下,CAM植物中的两种芦荟(Aloe vera L.和Aloe saponarea Haw) mRNA表达量逐步上升,并且酶的积累受到强烈的诱导。另外兼性CAM植物,如冰叶日中花(M. crystailinum),可以在高盐条件下由C3光合途径改为CAM代谢途径,并且在盐的处理下NADP-ME的活性可以提高4~10倍(Holtum and Winter, 1982;Winter et al., 1982)。最近,Fu等(2009)用NaCl处理了小麦的幼苗叶片,结果表明在用盐胁迫诱导后小麦幼苗叶片NADP-ME基因表达及酶活性检测都有所提高。同时,Liu等(2007)以及Cheng和Long (2007)也有研究表明,在NaCl处理下,水稻NADP-ME基因表达可以受到强烈的诱导,酶活性有所增加。由此可以看出,NADP-ME是代谢途径中的关键酶,与盐胁迫的关系密切。
温度对植物NADP-ME影响也有所报道。研究表明温度过高和过低都会对植物造成危害。温度过高会破坏植物的酶系统,从而使植物体的酶失活。温度过低会使原生质流动性下降,代谢失调等(许文花和杨清辉, 2003;徐燕等, 2007)。Fu等(2009)通过半定量RT-PCR研究分析表明,小麦的两个NADP-ME基因在经过低温处理后,它们的表达情况也都出现上升的趋势。并且处理小麦叶片24 h后,它的NADP-ME酶活性也有所升高。这些证据表明冷信号可能够诱导NADP-ME基因表达。
干旱也是影响植物生长的一个主要影响因子,它将引起渗透胁迫。对植物生长方面影响主要表现在引起气孔的关闭、降低叶绿素含量、抑制植物的光和作用等。目前报道有多种耐旱机制,在干旱条件下一些蛋白可以启动自身的防御的系统来抵御干旱胁迫带来的危害。其中NADP-ME就是其中一个。现在许多研究表明可能是植物体内某个胁迫信号刺激了基因表达,从而抵御干旱所带来的影响(Shao et al., 2008; Ni et al., 2009)。Cushman等(1992)研究表明在干旱条件能够诱导冰草NADP-ME 的mRNA的转录水平有所增加。另外,Chi等(2004)研究编码水稻的4个NADP-ME时,发现其中3个NADP-ME可以在10%PEG诱导下基因表达是上升的,仅仅有一个是下降的。这表明了并不是所有的NADP-ME都和干旱胁迫相关。
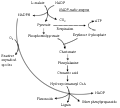
高等植物NADP-ME基因的转录水平受到多种环境因子的诱导,这可能是通过苹果酸的代谢参与了植物的防御代谢(Casati et al., 1999) (图1)。Rhodes等(1968)首次报道了NADP-ME与机械损伤有关,研究表明在切片的苹果中苹果酸的代谢水平起初在6 h比较缓慢,随后这种代谢速率快速上升2~3倍,24 h后又达到了一个稳定的水平,虽然这个实验没有直接表明NADP-ME与机械损伤有关,但是却暗示了NADP-ME与机械损伤相关。另外,Walter等(1988)研究组从诱导处理的豆分离出了一个全长的cDNA,通过实验分析表明在真菌诱导子得诱导下,它的转录水平是比较快的在处理1.5 h时达到最大,但是随后这个NADP-ME基因的mRNA转录水平却检测不到任何信号。同样这个研究小组在1990年通过同源性比对确定为豆的苹果酸酶基因(Walter et al., 1900)。与此同时,Casati等(1997)在研究用纤维素处理小麦叶片48 h后,发现NADP-ME活性增加了2.2倍。表明这个反应是由于这个诱导子参与了这个蛋白合成。
 |
影响NADP-ME的另一个因子UV-B也经过了广泛的研究,许多研究表明,UV-B能够诱导NADP-ME基因的表达并且NADP-ME活性和UV-B光照响应。Barkan等(2006)研究组发现培养三周豆的叶片,在用UV-B光照射一周后,发现NADP-ME活性增加了两倍。Casati等(1998)研究也表明在用UV-B光照射玉米幼苗2~60 min内,NADP-ME蛋白量以及酶活性都有所增加。这些结果表明NADP-ME可能参与了植物的防御功能,并且产生的还原物质NADPH用于木质素等防御物质的合成。
光照也是植物进行光和作用的主要因素,报道表明NADP-ME可以在植物体内参与光和作用,其中C4植物NADP-ME参与光和作用的功能报道研究的最早。Maurino等(1996)报道表明玉米叶片在受到光的诱导后基因表达增加。同样的结果也被Lai等(2002)研究小组的证明,它们通过GUS报告基因证明转基因植株在受到光照后,维管束鞘中的GUS染色活性显著增加。另外,最近Fu等(2009)的研究小组通过RT-PCR的方法表明小麦TaNADPME1和TaNADP-ME2是受光响应的基因,但是是否参与光合成代谢没有给出合理的解释。
4展望
本文对高等植物中NADP-ME的克隆、功能及其表达调控进行了相关叙述,近几年,高等植物的NADP-ME得到较为深入地研究。但对于大多数NADP-ME的了解还处于初步认识阶段,对该基因的分子生物学研究主要集中在植物抗性、防卫反应和果实成熟等方面。但对于NADP-ME在抗性方面的基因表达调控研究还不是很多,特别是在转基因植物功能解析方面报道相对较少。这也使其应用的效果有限,特别是在作物育种方面。随着分子生物学技术的发展,深入了解NADP-ME的生理生化特性、结构及功能、催化机制,将为探讨生物体中苹果酸代谢以及与辅酶再生研究奠定基础。
参考文献
Barkan L., Evans M.A., and Edwards G.E, 2006, Increasing UV-B induces biphasic leaf cell expansion in Phaseolus vulgaris, suggesting multiple mechanisms for controlling plant growth, Photochemistry and Photobiology, 82(6): 1612-1620 doi:10.1111/j.1751-1097.2006.tb09820.x PMid:16930091
Bologna F.P., Andreo C.S., and Drincovich M.F., 2007, Escherichia coli malic enzymes: Two isoforms with substantial differences in kinetic properties metabolic regulation, and structure, Journal of Bacteriology, 189(16): 5937-5946 doi:10.1128/JB.00428-07 PMid:17557829 PMCid:1952036
Casati P., Drincovich M.F., Andreo C.S., Donahue R., and Edwards G.E., 1998, UV-B, red and far-red light regulate induction of the C4 isoform of NADP-malic enzyme in etiolated maize seedlings, Australian Journal of Plant Physiology, 25(6): 701-708 doi:10.1071/PP98004
Casati P., Drincovich M.F., Edwards G.E., and Andreo C.S., 1999, Malate metabolism by NADP- malic enzyme in plant defense, Photosynthesis Research, 61(2): 99-105 doi:10.1023/A:1006209003096
Casati P., Spampinato C.P., and Andreo C.S., 1997, Characteristics and physiological function of NADP-malic enzyme from wheat, Plant Cell Physiology, 38(8): 928-934
Chang G.G., and Tong L., 2003, Structure and function of malic enzymes, a new class of oxidative decarboxylases, Biochemisty, 42(44): 12721-12733 doi:10.1021/bi035251+ PMid:14596586
Cheng Y.X., and Long M., 2007, A cytosolic NADP-malic enzyme gene from rice (Oryza sativa L.) confers salt tolerance in transgenic Arabidopsis, Biotechnology Letters, 29(7): 1129-1134 doi:10.1007/s10529-007-9347-0 PMid:17516134
Chi W., Yang J.H., Wu N.H., and Zhang F., 2004, Four rice genes encoding NADP malic enzyme exhibit distinct expression profiles, Bioscience Biotechnology Biochemistry, 68(9): 1865-1874 doi:10.1271/bbb.68.1865 PMid:15388961
Cushman J.C., 1992, Characterization and expression of a NADP-malic enzyme cDNA induced by salt stress from the facultative crassulacean acid metabolism plant, Mesembryanthemum crystallinum, European Journal of Biochemistry, 208: 259-266 doi:10.1111/j.1432-1033.1992.tb17181.x PMid:1521524
Davies D.D., 1986, The fine control of cytosolic pH, Physiologia Plantarum, 67(4): 702-706 doi:10.1111/j.1399-3054.1986.tb05081.x
Detarsio E., Andreo C.S., and Drincocich M.F., 2004, Basic residues play key roles in catalysis and NADP+-specificity in maize (Zea mays L.) photosynthetic NADP+-dependent malic enzyme, The Biochemical Journal, 382(Pt3): 1025-1030 PMid:15245332 PMCid:1133980
Detarsio E., Maurino V.G., Alvarez C.E., Müller G.L., Andreo C.S., and Drincovich M.F., 2008, Maize cytosolic NADP-malic enzyme (ZmCytNADP-ME): A phylogenetically distant isoform specifically expressed in embryo and emerging roots, Plant Molecular Biology, 68(4-5): 355-367 doi:10.1007/s11103-008-9375-8 PMid:18622731
Drincovich M.F., Casati P., and Andreo C.S., 2001, NADP-malic enzyme from plants: A ubiquitous enzyme involved in different metabolic pathways, FEBS Letters, 490(1-2): 1-6 doi:10.1016/S0014-5793(00)02331-0
Drouet A.G., and Hartmann C.J.R., 1977, Activity of pear fruit malic enzyme; its regulation by metabolites, Phytochemistry, 16(5): 505-508 doi:10.1016/0031-9422(77)80002-2
Franke K.E., and Adams D.O., 1995, Cloning of a full-length cDNA for malic enzyme (EC 1.1.1.40) from grape berries, Plant Physiology, 107(3): 1009-1010 doi:10.1104/pp.107.3.1009 PMid:7716227 PMCid:157218
Fu Z.Y., Zhang Z.B., Hu X.J., Shao H.B., and Xu Ping., 2009, Cloning, dentification, expression analysis and phylogenetic relevance of two NADP-dependent malic enzyme genes from hexaploid wheat, Comptes Rendus Biologies, 332(7): 591-602 doi:10.1016/j.crvi.2009.03.002 PMid:19523599
Fushimi T., Umeda M., Shimazaki T., Kato A., Toriyama K., and Uchimiya H., 1994, Nucleotide sequence of a rice cDNA similar to a maize NADP-dependent malic enzyme, Plant Molecular Biology, 24(6): 965-967 doi:10.1007/BF00014450 PMid:8204833
Goodenough P.W., Prosser I.M., and Young K., 1985, NADP-linked malic enzyme sand malate metabolism in ageing tomato fruit, Phytochemistry, 24(6): 1157-1162 doi:10.1016/S0031-9422(00)81093-6
Holtum J.A.M., and Winter K., 1982, Activity of enzymes of carbon metabolism during the induction of Crassulacean acid metabolism in Mesembryanthemum crystallinum L., Planta, 155(1): 8-16 doi:10.1007/BF00402925
Honda H., Akagi H., and Shimada H., 2000, An isozyme of the NADP-malic of a CAM plant, Aloe arborescens, with variation on conservative amino acid residues, Gene, 243(1-2): 85-92 doi:10.1016/S0378-1119(99)00556-9
Honda H., Shimada H., and Akagi H., 1997, Isolation of a cDNA for an NADP-malic enzyme from Aloe arborescens, DNA Research, 4(6): 397-400 doi:10.1093/dnares/4.6.397 PMid:9501996
Krishnamurthy S., and Patwardhan M.V., 1971, Properties of malic enzyme (decarboxylating) from pulp of mango fruit (Mangifera indica), Phytochemistry, 10(8): 1811-1815 doi:10.1016/S0031-9422(00)86442-0
Lai L.B., Wang L., and Nelson T.M., 2002, Distinct but conserved functions for two chloroplastic NADP-malic enzyme isoforms in C3 and C4 Flaveria species, Plant Physiology, 128(1): 125-139 doi:10.1104/pp.010448 PMid:11788758 PMCid:148954
Lipka B., Steinmuller K., Rosche E., Börsch D., and Westho P., 1994, The C3 plant Flaveria pringlei contains a plastidic NADP-malic enzyme which is ortholous to the C4 isoform of the C4 plant F. pringlei, Plant Molecular Biology, 26(6): 1775-1783 doi:10.1007/BF00019491 PMid:7858216
Liu S.K., Cheng Y.X., Zhang X.X., Guan Q.J., Nishiuchi S., Hase K., and Takano T., 2007, Expression of an NADP-malic enzyme gene in rice (Oryza sativa L.) is induced by environmental stresses; over-expression of the gene in Arabidopsis confers salt and osmotic stress tolerance, Plant Molecular Biology, 64(1-2): 49-58 doi:10.1007/s11103-007-9133-3 PMid:17245561
Martinoia E., and Rentsch D., 1994, Malate compartmentation-responses to a complex metabolism, Annual Review of Plant Physiology and Plant Molecular Biology, 45: 447-467 doi:10.1146/annurev.pp.45.060194.002311
Maurino V.G., Drincovich M.F., and Ndreo C.S., 1996, NADP malic enzyme isoforms in maize leaves, Biochemistry and Molecular Biology International, 38(2): 239-250 PMid:8850519
Ni F.T., Chu L.Y., Shao H.B., and Liu Z.H., 2009, Gene expression and regulation of higher plants under soil water stress, Current Genomics., 10(4): 269-280 doi:10.2174/138920209788488535 PMid:19949548 PMCid:2709938
Rhodes M.J.C., Wooltorton L.S.C., Galliard T., and Hulme A.C., 1968, Metabolic changes in excised fruit tissue-I. Factors affecting the development of a malate decarboxylation system during the ageing of disks of pre-climacteric apples, Phytochemistry, 7(9): 1439-1451 doi:10.1016/S0031-9422(00)88589-1
Rothemel B.A., and Nelson T., 1989, Primary structure of maize NADP-dependent malic enzyme, The Journal of Biology Chemistry, 264(33): 19587-19592 PMid:2584183
Saitou K., Agata W., Masui Y., Asakura M., and Kubota F., 1994, Isoforms of NADP-malic enzyme from Mesembryanthemum crystallinum L. that are involved in C3 photosynthesis and crassulacean acid metabolism, Plant Cell Physiology, 35(8): 1165-1171
Shao H.B., Chu L.Y., Lu Z.H., and Kang C.M., 2008, Primary antioxidant free radical scavenging and redox signaling pathways in higher plant cells, International Journal of Biological Sciences, 4(1): 8-14
Song Y.D., Wynn J.P., Li Y.H., Grantham D., and Ratledge C., 2001, A pre-genetic study of the isoforms of malic enzyme associated with lipid accumulation in Mucor circinelloides, Microbiology, 147(Pt6): 1507-1515 PMid:11390681
van Doorsseleare J., Villaroel R., van Montagu M., and Inze D., 1991, Nucleotide sequence of a cDNA encoding malic enzyme from poplar, Plant Physiology, 96(4): 1385-1386 doi:10.1104/pp.96.4.1385 PMid:16668348 PMCid:1080944
Walter M.H., Grima-Pettenati J., Grand C., Boudet A.M., and Lamb C.J., 1988, Cinnamyl-alcohol dehydrogenase, a molecular marker specific for lignin synthesis: cDNA cloning and mRNA induction by fungal elicitor, Proceeding of the National Academy of Sciences of the United States of America, 85(15): 5546-5550
Walter M.H., Grima-Pettenati J., Grand C., Boudet A.M., and Lamb C.J., 1900, Extensive sequence similarity of the bean CAD4 (cinnamyl-alcohol dehydrogenase) to a maize malic enzyme, Plant Molecular Biology, 15(3): 525-526 doi:10.1007/BF00019173 PMid:2103472
Wheeler M.C.G., Tronconi M.A., Drincovich M.F., Andreo C.S., Flügge U., and Maurino V.G., 2005, A comprehensive analysis of the NADP-malic enzyme gene family of Arabidopsis, Plant Physiology, 139(1): 39-51 doi:10.1104/pp.105.065953 PMid:16113210 PMCid:1203356
Winter K., Foster J.G., Edwards G.E., and Holtum J.A.M., 1982, Intracellular localization of enzymes of carbon metabolism in Mesembryanthemum crystallinum exhibiting C3 photosynthetic characteristics or performing crassulacean acid metabolism, Plant Physiology, 69(2): 300-307 doi:10.1104/pp.69.2.300 PMid:16662197 PMCid:426198
Wynn J.P., Hamid A.A., and Ratledge C., 1999, The role of malic enzyme in the regulation of lipid accumulation in filamentous fungi, Microbiology, 145(8): 1911-1917 doi:10.1099/13500872-145-8-1911 PMid:10463157
Xu W.H., and Yang Q.H., 2003, Advances in the research of cold resistance in sugarcane, Ganzhe (Sugarcane), 10(3): 8-12 (许文花, 杨清辉, 2003, 甘蔗抗寒性研究进展, 甘蔗, 10(3): 8-12)
Xu Y., Xue L., and Qu M., 2007, Physiological and ecological mechanisms of plant adaptation to low temperature, Linye Kexue (Scientia Silvae Sinicae), 43(4): 88-94 (徐燕, 薛立, 屈明, 2007, 植物抗寒性的生理生态学机制研究进展, 林业科学, 43(4): 88-94)
Zhang Z.B., Shao H.B., Xu P., Hu M.Y., Song W.Y., and Hu X.J., 2009, Focus on agricultural biotechnology: Prospective for bio-watersaving theories and their applications in the semi-arid and arid areas, African Journal of Biotechnology, 8(12): 2779-2789