2广东省生物活性药物研究重点实验室, 广州, 510006
 作者
作者  通讯作者
通讯作者
基因组学与医学生物学, 2012 年, 第 1 卷, 第 3 篇 doi: 10.5376/gmb.cn.2012.01.0003
收稿日期: 2012年04月02日 接受日期: 2012年06月28日 发表日期: 2012年07月02日
引用格式(中文):
卢雪梅等, 2012, 肝靶向穿膜肽与家蝇天蚕素基因的融合及其分子特征分析, 基因组学与医学生物学(online) Vol.1 No.3 pp.18-22 (doi: 10.5376/gmb.cn.2012.01.0003)
引用格式(英文):
Lu et al., 2012, Construction and Molecular Characteristics of Fusion Gene HTPP-MDC, Jiyinzuxue Yu Yixue Shengwuxue (online) (Genomics and Medical Biology) Vol.1 No.3 pp.18-22 (doi:10.5376/gmb.cn.2012.01.0003)
本研究利用改进SOE-PCR技术构建肝靶向穿膜肽(HTPP)与家蝇天蚕素(MDC)融合基因并对其分子特征进行了预测和分析。结果表明:成功融合了HTPP与MDC,并构建了HTPP-MDC融合基因的克隆重组质粒HTPP-MDC/pMD20-T。PCR和KpnⅠ/Hind Ⅲ双酶切结果显示获得与预期大小一致的基因片段,测序结果显示获得的基因序列没有发生突变,与预期完全一致。分子特征分析表明,该融合基因编码60个氨基酸,分子量为6 516.2 Da,理论等电点为9.31,二级结构主要由α-螺旋、无规则卷曲、延伸链和β-转角组成。研究结果为HTPP-MDC后续的功能研究奠定了基础,同时也为应用SOE-PCR技术构建融合基因提供了有益借鉴。
肝靶向穿膜肽(hepatocyte-targeted cell penetrating peptide, HTPP)来源于恶性疟原虫环子孢子蛋白。研究发现HTPP可特异性结合肝细胞表面的受体,与人肝癌细胞HepG 2具有高亲和力(80 nmol/L) (Garcia et al., 2006; Suarez et al., 2001)。HTPP还能够有效穿透肝细胞膜,介导环子孢子蛋白进入肝细胞内部,实现细胞亚定位(De Koning-Ward et al., 2009)。
抗菌肽(antimicrobial peptides, AMP)是一类小分子多肽,产生于生物免疫系统用于抵抗外界病原体感染。研究显示AMP可通过阻断病毒粘附侵入(Andersen et al., 2004),抑制病毒DNA复制(Wachinger et al., 1998),以及刺激机体免疫反应(Jenssen, 2009)等机制发挥抗病毒作用。
如能将肝靶向穿膜肽(HTPP)与抗菌肽(AMP)二者融合,构建的融合多肽将利用HTPP靶向肝脏,进入肝细胞内直接发挥抗乙型肝炎病毒(hepatitis B virus, HBV)作用,有望成为新型靶向抗HBV药物,对降低HBV感染引起的发病率和死亡率具有重大意义。
本研究利用改进SOE-PCR技术构建HTPP与来源于家蝇的抗菌肽天蚕素(Musca domestica cecropin, MDC)融合基因,并用相关生物信息学方法对其推导的氨基酸序列进行预测和分析,试图为融合基因的基因工程表达及其生物活性研究奠定基础。
1结果与分析
1.1融合基因的构建和鉴定
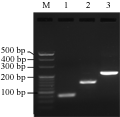
利用SOE-PCR方法,以设计的4条引物进行扩增得到片段HM1、HM2大小分别为82 bp、154 bp,最后拼接的全长HTPP-MDC基因片段大小约为220 bp。结果显示每轮PCR产物的大小与预期相符,无非特异性条带出现(图1)。
|
|
回收获得的全长目的片段并克隆入pMD20-T载体后,进行PCR和KpnⅠ/Hind Ⅲ双酶切鉴定,结果显示PCR扩增和酶切后的基因片段与预期片段大小一致(图2)。测序结果表明该基因序列与预期完全一致,没有突变发生。以上结果表明已成功构建HTPP-MDC融合基因。
|
|
1.2 HTPP-MDC的分子特征分析
ProtParam分析结果显示HTPP-MDC基因编码蛋白含60个氨基酸,其中碱性氨基酸(Arg+Lys) 10个,酸性氨基酸(Asp+Glu) 8个,含量最丰富的氨基酸包括Gly (11.7%)、Asn (10.0%)、Lys (10.0%);其分子量为6 516.2 Da,理论等电点为9.31,分子式为C274H459N91O93;蛋白的脂溶性指数为76.50,GRAVY指数为-0.985,不稳定指数为24.77;在大肠杆菌内的半衰期大于10小时,在体外有机体内的半衰期为3 min,在体外哺乳动物网状细胞中的估计半衰期为1.4 h。
用ProtScale Server进行亲水性/疏水性预测,结果显示HTPP-MDC中亲水氨基酸数量大于疏水氨基酸,且主要位于多肽链的N端(图3)。
|
|
SignalP 3.0 Server分析HTPP-MDC的信号肽分析图谱如图4所示。由分析结果可知,HTPP-MDC中不存在信号肽序列,这与之前融合基因构建的设想完全一致。
|
|
利用PSORTⅡ Prediction蛋白质亚细胞定位分析软件对HTPP-MDC进行预测,结果显示其在各细胞器分布比例为:细胞核65.2%、细胞质17.4%、线粒体17.4%,这证实了HTPP-MDC编码蛋白属于胞质蛋白。
用NPS@对HTPP-MDC基因编码蛋白的二级结构进行预测(图5),结果显示在HTPP-MDC氨基酸序列中,α-螺旋、无规则卷曲、延伸链和β-转角各为37、14、7和2处,分别占总二级结构的61.67%、23.33%、11.67%和3.33%。由此可推测,HTPP-MDC中主要结构元件是α-螺旋,其次为无规则卷曲和延伸链。
|
|
2讨论
HBV主要在肝细胞内寄生和复制,可导致急慢性乙型肝炎发生,甚至引起肝硬化和肝癌,危害极大。目前临床上用于HBV感染的抗病毒药物虽具有一定疗效,但也存在着用药剂量大、疗效不够理想、毒副作用大等问题。将可特异结合肝细胞表面受体的HTPP与具有多重抗病毒机制的抗菌肽融合,有望成为特异性好、用药量少、毒副作用小的新型高效靶向抗HBV药物。本研究利用基因重组技术构建融合基因,试图为获得一定量的HTPP-MDC进行后续的功能研究奠定基础。
目前融合基因的构建方法主要有限制内切酶法和重叠延伸PCR (gene splicing by overlap extension PCR, SOE-PCR) 技术。SOE-PCR技术简便易行,不需要限制性内切酶消化和连接酶处理就可实现任意两个目的基因的连接和扩增,克服了因设计限制性内切酶位点而引入不必要的氨基酸残基的缺点,保证了目的蛋白的真实性和完整性(谷铁军等, 2010)。本研究对传统的SOE-PCR方法进行了改进,通过三个反应步骤(图6)成功地构建了融合基因。在应用改进SOE-PCR技术快速、准确地进行基因融合时,须注意以下几点:
|
|
(1)在传统的SOE-PCR步骤中加入一步:即在不加引物的情况下,将等摩尔的各片段混合先进行10个循环,这种方法可减少非特异性条带的产生,提高产物的特异性。
(2)选择保真性能和扩增效率均高的DNA聚合酶。应用普通的DNA聚合酶进行扩增时,PCR产物的3末端会产生poly A尾,将阻止互补区域退火以后的延伸反应,并且容易造成移码、错配和突变等,无法得到全长的融合产物,因此扩增过程中必须采用具有5′~3′聚合酶和3′~5′外切酶的活性的高保真聚合酶。但传统高保真聚合酶在扩增效率上,一直难以令人满意,本研究使用的为Prime-STAR HS DNA Polymerase (TaKaRa公司),极大地提高了SOE-PCR反应的效率与准确性。本研究对现有SOE-PCR技术的改进之处也在于考虑到下一步的TA克隆,直接在最后一轮PCR反应液中加入普通Taq酶72℃延伸20 min为融合基因加上Poly A尾,不用再一次进行PCR反应,同时也减少了突变的发生。
(3)合理设计引物序列。首先应综合考虑碱基的合成成本和连续反应的次数,寡聚核苷酸链的长度应适宜,过长不利于合成、过短则增加成本和反应次数。我们经过数次重复试验发现引物长度为55~59 bp时,在引物合成成本和扩增效率上的性价比是最高的。其次是引物间重叠区域的长度和选择,现有SOE-PCR技术中相邻引物间的重叠区域一般为25~30个碱基,我们在实验中通过综合考虑引物的Tm值和重叠区的碱基序列等因素,设计的重叠区域短至15~16 bp均获得了有效的扩增。
(4)高质量的模板。由于每轮PCR产物作为下一轮PCR反应的模板,所以PCR产物应进行纯化,以免给后面的反应带来不良后果。同时在回收过程中尽量避免EB及紫外对模板造成的损伤:如用于回收产物的凝胶中应减少EB的加入量(低于0.15 μg/mL),另外切胶过程中应尽量减少紫外线照射时间。
获得的融合基因经测序验证与预期完全一致,在合成过程中没有产生突变。随后我们利用生物信息学软件对融合基因推导的氨基酸序列进行了预测和分析,结果表明HTPP-MDC为阳离子多肽、结构稳定、具有亲水性,二级结构主要由α-螺旋、无规则卷曲、延伸链和β-转角组成,与融合前亲本肽的二级结构基本一致,在一定程度上说明融合多肽保持了与天然多肽类似的空间构象。HTPP-MDC有望开发成新型肝靶向抗HBV药物,成功构建融合多肽并对其分子特征进行了预测和分析,为HTPP-MDC的功能研究和开发利用提供了物质基础和理论依据,同时也为应用SOE-PCR技术快速、准确地构建融合基因提供有益借鉴。
3材料与方法
3.1实验材料
Escherichia coli (E. coli) DH5α、MDC/pMD20-T质粒为本实验室保存。pMD20-T载体,rTaq DNA Polymerase,高保真酶PrimeSTAR HS DNA Polymerase,DNA分子量标准,限制性内切酶KpnⅠ、Hind Ⅲ购自TaKaRa公司;TIANpure Midi Plasmid Kit和TIANgel Midi Purification Kit为北京TIANGEN公司产品;其他化学试剂为国产或进口分析纯。
3.2引物设计
设计4条寡核苷酸引物,其中H1、M1为与目的基因同向部分的引物,H2、M2为与目的基因互补部分的引物,M1和M2用于从MDC/pMD 20-T质粒中扩增MDC片段。H1与H2、H2与M1之间分别有16 bp的重叠互补区。外侧上游引物H1中包含KpnⅠ酶切位点,外侧下游引物M2中设计了终止密码子和Hind Ⅲ酶切位点,各引物在目的基因中的位置如图6所示。引物设计如下(划线部分为酶切位点,斜体为重叠DNA序列部分,粗体为终止密码子):
H1: 5'-CGGGGTACCGACGACGACGACAAGAATAGTCGTTCACTTG-3';H2: 5'-GTAATTTCTCGTTGTCTTCGTTATTTCCATCATCATTTTCTCCAAGTGAACGACTATT-3';M1: 5'-GACAACGAGAAATTACGTGGATGGTTGAAAAAAAT-3';M2: 5'-CCCAAGCTTATTAGTTACCCTTTAATGTGGCGGCA-3';
3.3 SOE-PCR法构建融合基因
将整条序列分成两段(HM1, HM2),首先分别PCR扩增出这两段序列,然后用一轮PCR进行拼接,最后扩增全长目的基因序列。
HM1的扩增:将上述合成的引物H1和H2进行链延伸反应,扩增体系如下:13.75 μL Sterile H2O,2.0 µL dNTP Mixture (2.5 mmol/L each),5.0 µL 5×PrimeSTAR Buffer,2.0 μL H1 Primer (10 μM/L), 2.0 μL H2 Primer (10 μM/L),0.25 µL PrimeSTAR HS DNA Polymerase (2.5 units/μL)。扩增条件为:98℃ 10 s,68℃ 40 s,循环30 次。
HM2的扩增:以pMD 20-T/MDC质粒为模板,M1和M2为引物进行PCR反应,扩增体系如下:14.75 μL Sterile H2O,2.0 µL dNTP Mixture (2.5 mmol/L each),5.0 µL 5×PrimeSTAR Buffer,1.0 μL M1 Primer (10 μM/L),1.0 μL M2 Primer (10 μM/L),1.0 μL pMD 20-T/MDC Plasmid,0.25 µL PrimeSTAR HS DNA Polymerase (2.5 units/μL)。扩增条件为:98℃ 10 s, 68℃ 40 s,循环30次。
全长目的基因的拼接与扩增:第一轮以上一步PCR扩增获得的HM1和HM2互为模板进行链延伸反应,扩增体系如下:5.0 µL 5×PrimeSTAR Buffer,2.0 µL dNTP Mixture (2.5 mmol/L each),15.75 μL Sterile H2O,1.0 μL HM1,1.0 μL HM2,0.25 µL PrimeSTAR HS DNA Polymerase (2.5 units/μL)。扩增条件为:98℃ 10 s,68℃ 30 s,循环10次。第二轮向PCR管内分别加入1.0 μL的外引物H1和M2大量扩增。扩增条件为:98℃ 10 s,68℃ 40 s,循环30次。第三轮向PCR管内加入0.125 μL的rTaq DNA Polymerase (5 units/μL),72℃延伸20 min,为融合基因加上Poly A尾,以利于下一步的TA克隆。用2%琼脂糖凝胶电泳检测PCR产物,凝胶成像系统观察并拍照。
3.4重组质粒HTPP-MDC/pMD20-T的构建及鉴定
用TIANgel Midi Purification Kit回收目的片段后,与pMD20-T载体连接构建重组质粒HTPP-MDC/pMD20-T,转化E. coli DH5a感受态细胞进行蓝白斑筛选,挑取阳性克隆培养后进行菌落PCR鉴定。取初步鉴定为阳性的克隆菌液,用TIANpure Midi Plasmid Kit抽提质粒,抽提的质粒用限制性内切酶Kpn Ⅰ、Hind Ⅲ进行双酶切鉴定,并委托Invitrogen公司进行DNA测序分析。
3.5融合基因的分子特征分析
利用互联网上的在线工具对获得的融合基因编码蛋白进行分析。用蛋白质基本性质分析工具ProtParam Tool (http://www.expasy.org/tools/protparam.html)对HTPP-MDC基因编码蛋白的分子量、氨基酸序列组成和等电点等理化性质进行分析;用疏水性分析软件ProtScal程序(http://web.expasy.org/cgi-bin/protscale.pl)分析其亲水性/疏水性;用SignalP 3.0 Server (http://www.cbs.dtu.dk/services/SignalP)分析其是否存在信号肽,用PSORTⅡ Prediction (http://psort.hgc.jp/form2.html)对其亚细胞定位进行预测分析,用sopma (http://npsa-pbil.ibcp.fr/cgi-bin/secpred_sopma.pl)预测其二级结构和折叠类型。
作者贡献
卢雪梅完成了主要实验操作和文章初稿的写作;黄演婷和汪洁协助细菌培养和质粒提取;金小宝和朱家勇负责文章的修改、审阅和完善。
致谢
本研究由国家自然科学基金(30671832)资助。感谢褚夫江博士对本文的指导。
参考文献
Andersen J.H., Jenssen H., Sandvik K., and Gutteberg T.J., 2004, Anti-HSV activity of lactoferrin and lactoferricin is dependent on the presence of heparan sulphate at the cell surface, Journal of Medical Virology, 74(2): 262-271
http://dx.doi.org/10.1002/jmv.20171 PMid:15332275
De Koning-Ward T.F., Gilson P.R., Boddey J.A., Rug M., Smith B.J., Papenfuss A.T., Sanders P.R., Lundie R.J., Maier A.G., Cowman A.F., and Crabb B.S., 2009, A newly discovered protein export machine in malaria parasites, Nature, 459(7249): 945-949
http://dx.doi.org/10.1038/nature08104 PMid:19536257 PMCid:2725363
Garcia J.E., Puentes A., and Patarroyo M.E., 2006, Developmental biology of sporozoite-host interactions in Plasmodium falciparum malaria: implications for vaccine design, Clinical Microbiology Reviews, 19(4): 686-707
http://dx.doi.org/10.1128/CMR.00063-05 PMid:17041140 PMCid:1592691
Gu T.J., Zhang F.Y., Duan Y., Wei W., Jiang C.L., Wu Y.G., and Kong W., 2010, Synthesis of Anti-Rabies ScFv Fragment Gene FV 57 by SOE-PCR, ShengWu JiShu (Biotechnology), 21(3): 36-39 (谷铁军, 张凤羽, 段冶, 卫巍, 姜春来, 吴永革, 孔维, 2010, SOE-PCR法合成狂犬病毒单链抗体基因Fv57, 生物技术, 21(3): 36-39)
Jenssen H., 2009, Therapeutic approaches using host defence peptides to tackle herpes virus infections, Viruses, 1(3): 939-964
http://dx.doi.org/10.3390/v1030939 PMid:21994576 PMCid:3185547
Suarez J.E., Urquiza M., Puentes A., Garcia J.E., Curtidor H., Ocampo M., Lopez R., Rodriguez L.E., Vera R., Cubillos M., Torres M.H., and Patarroyo M.E., 2001, Plasmodium falciparum circumsporozoite (CS) protein peptides specifically bind to HepG2 cells, Vaccine, 19(31): 4487-4495
http://dx.doi.org/10.1016/S0264-410X(01)00203-1
Wachinger M., Kleinschmidt A., Winder D., Von Pechmann N., Ludvigsen A., Neumann M., Holle R., Salmons B., Erfle V., and Brack-Werner R., 1998, Antimicrobial peptides melittin and cecropin inhibit replication of human immunodeficiency virus 1 by suppressing viral gene expression, Journal of General Virology, 79(Pt 4): 731-740
PMid:9568968
.png)
.png)
.png)
.png)