2 广州医学院卫生职业技术学院, 广州, 510450;
3 萍乡高等专科学校, 萍乡, 337000;
4 中山大学,广州, 510275
 作者
作者  通讯作者
通讯作者
植物药与药理学杂志, 2013 年, 第 2 卷, 第 5 篇
收稿日期: 2013年04月05日 接受日期: 2013年04月21日 发表日期: 2013年04月28日
以PCR产物克隆后测序的方法测定了贵州赤水桫椤国家级自然保护区三个种群59个个体的叶绿体DNA(CpDNA) atpB-rbcL非编码区序列。序列长度介于727~732 bp之间,具长度多态性,A+T百分比含量较高,介于63.27%~63.89%之间。遗传多样性水平较低,表现出较高的单倍型多样性(h=0.639)和低的核苷酸多样性(Dij=0.00028)并存的特征,提示现有种群可能源自一个有效群体规模较小的种群的快速扩张,扩张时间尚短,不足以形成更为复杂的遗传结构。种群间的高基因流Nm,低遗传分化度FST,AMOVA分析以及DNA歧异度结果一致显示各种群间无明显的遗传分化。
桫椤(Alsophila spinulosa(Wall.ex Hook.)Tryon)属桫椤科(Cyatheacea)桫椤属(Alsophila),是古老的冰川孑遗植物,有“活化石”之称,国家濒危保护植物。在距今1.8亿年前的中生代侏罗纪时期桫椤已经存在,且分布范围很广,十分繁盛,经第四纪冰川的侵袭后,其生存受到严重的影响,现今其分布区己大大缩小(Tryon, 1970),仅分布在热带、亚热带地区(Lucansky, 1974; willis, 2002),在我国,桫椤自然种群主要分布在广东、广西、台湾、福建、海南、贵州、四川、云南等地区(夏群, 1989, 中国桫椤科植物的分类, 植物分类学报, 27(1): 1-16)。
桫椤是研究物种形成与植物地理分布关系,地质学及古生物学的理想材料,尽管近些年在生态、形态、人工繁殖、物候等方面对其有不同程度的研究,遗传学方面的研究也取得了一定进展,但其分子群体研究仍嫌不足。
目前用于系统学研究的叶绿体基因间隔区主要有atpB-rbcL、trnC-rpoB、trnL-trnF等非编区,其单亲遗传及快速近中性的进化方式使之适用于种内地理亲缘模式的重建(Ferris et al., 1990),同时DNA测序在技术上回避了RFLP及以PCR为基础的DNA指纹技术无法区分具相同长度但序列不同的片段的局限性。atpB-rbcL间隔区的通用引物被设计出后,这一非编码区被广泛用于种群水平系统发育的分析(Schaal et al., 1998; Huang et al., 2001; Chiang et al., 2001; Trewick et al., 2002; Lu et al., 2002; Hwang et al., 2003)。贵州赤水桫椤国家级自然保护区属中亚热带湿润季风气候,河谷具有类似南亚热带气候特征,冬无严寒,夏无酷暑,湿度大,达90%,年降水量1200~1300mm,云雾雨日多,是全国日照最少地区之一,河谷全年不足650小时,山顶也未超过1000小时,是桫椤生长的理想之地。
本研究以保护区内三个种群为材料,通过分析cpDNA atpB-rbcL非编码序列,检测其遗传多样性及分化水平,探讨与桫椤种群遗传结构形成有关的因素,为桫椤的研究和保护提供分子遗传学方面的依据。
1结果与分析
1.1 DNA提取和PCR产物
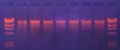
提取的桫椤总DNA片段大小约为20 kb位置(图1),除个别条带有一定程度的降解外,其它条带带型较好。紫外分光光度测定的DNA的A260/A280的值均在1.785-2.203之间,显示总DNA的浓度及纯度符合实验要求。PCR扩增后,回收产物经过电泳检测显示条带清晰,无杂带(图2),可用于测序。
|
图 1 桫椤总DNA |
|
图 2 桫椤atpB-rbcL回收产物检测 |
1.2种群遗传结构及遗传多样性
分样品的cpDNA atpB-rbcL非编码区序列长度介于727~732bp之间,具长度多态性, A+T百分比含量较高,介于63.27%~63.89%之间。59个体共检测到9个单倍型,单倍型多样性(h=0.639)较高,核苷酸多样性(Dij=0.00028)低,且各种群差别不大(表1)。
|
表 1 cpDNA atpB-rbcL非编码序列单倍型多样性(h)和核苷酸多样性(Dij) |
基于桫椤cpDNA atpB-rbcL非编码区序列变异计算出种群间基因流Nm较高,介于5.52~6.64之间,种群分化度FST较低,介于0.0000~0.03448之间(表2)。AMOVA分析显示有85.16%的遗传变异来种群内(P<0.001)。从种群间DNA歧异度角度来分析,核苷酸差异平均数和每位点核苷酸替换平均数均较低,分别介于0.154~0.250之间和0.00021~0.00034之间(表3)。
|
表 2 cpDNA atpB-rbcL非编码区比较桫椤种群间的Nm(对角线上)和FST(对角线下) |
|
表 3 种群间核苷酸差异平均数(对角线上)和每位点核苷酸替换平均数(对角线下) |
2讨论
使研究表明,桫椤种群间有较高的基因流Nm,考虑到桫椤种群现代生境的片断化,桫椤科植物孢子有限的迁移能力以及孢子自身脆弱的生命力(程志英等, 1990),推测统计上的高基因流并不一定表示种群间能够实现高效的基因流,也可能是选择压力相同造成的。桫椤种群的遗传多样性水平低,表现出较高的单倍型多样性和低核苷酸多样性并存的特征,这种式样提示现有种群源自有一个有效群体规模较小的种群的快速扩张(Avise, 2000)。地质研究表明在更新世形成了寒冷的冰期和温暖的间冰期的多次交替(Bennett, 1990),冰期种群的缩小,间冰期种群的增长,能够消除过去积累的核苷酸多样性,但又能快速导致少数碱基位点的变异,单个碱基的变异可以生成新的单倍型,但对核苷酸多样性的影响有限,从而使单倍型多样性相对高,但核苷酸多样性的积累还需更长时间,奠基者效应和瓶颈效应可能是影响桫椤种群遗传结构的一个重要原因。桫椤种群间低的遗传分化度FST,AMOVA分析以及DNA歧异度结果一致显示,基于cpDNA atpB-rbcL非编码区序列分析,各种群之间无明显的遗传分化。
根据桫椤种群遗传结构和遗传多样性水平,建议对保护区内桫椤进行就地保护,重视分布点内尽可能多个体的保护,防止人为的砍伐和生境的退化,采取策略促进这些群体内的幼苗更新,建立在不同生态序列内的桫椤种群永久观察点,定期观察与研究桫椤种群的动态发展。在迁地保护、取样以及育种时,应在较大群体中尽可能多取样,最大限度地保护桫椤的遗传多样性。
3材料和方法
3.1实验材料
提样品采自贵州赤水桫椤自然保护区内三个种群,采样树高0.6~5.0 m不等,每个种群选取个体之间距离大于20 m,将采集的叶片编号,置于硅胶中密封保存。采样种群分布(表4)。
|
表 4 桫椤样品采集资料 |
3.2实验方法
3.2.1总DNA提取
DNA的提取采用改进的CTAB法(苏应娟等, 1998; 张岳峰等, 2008),琼脂糖电泳检测所提取总DNA片段的大小及质量,紫外分光光度计检测DNA样品的浓度和纯度,稀释至50ng/μl,用做PCR扩增的模板。
根据DNA的提取采用改进的CTAB法(苏应娟等, 1998; 张岳峰等, 2008),琼脂糖电泳检测所提取总DNA片段的大小及质量,紫外分光光度计检测DNA样品的浓度和纯度,稀释至50 ng/μl,用做PCR扩增的模板。
3.2.2 PCR扩增与回收
每50 μl反应体系含50 mmol/L的KCl,1.5 mmol/LMgCl2,10 mmol/L的Tris-HCl,0.1%TritonX-100,dNTP各0.1 mmol,模板50 ng,Taq酶1.25U,Pfu酶0.3U,引物atpB:BACATCKARTACKGGACCAATAA,rbcL:AACACCAGCTTTRAATCCAA
引物序列由上海英骏生物技术有限公司合成。PCR扩增程序94ºC预变性7 min,94ºC 45 s,52ºC 50 s,72ºC 90 s,32个循环,72ºC延伸10 min。扩增产物在1.0%的琼脂糖凝胶上进行电泳检测,并在上样孔两端加入与PCR产物检测等量的100 bp DNA Ladder与目的片段对照。产物经浓缩,电泳分离后回收。
3.2.3克隆测序
回收产物与pMD18-T载体连接,转入感受态DH5-α细胞中,于含有50ng/mL Amp 的平板培养基上采用蓝白筛选的方法获得克隆,PCR扩增后电泳检测T载体中插入片段的大小,确定阳性克隆,应用载体两端通用引物M13F和M13R在3730自动测序仪上测序。
3.2.4数据分析方法
用DNAstar将阳性克隆测定的正反向序列拼接,切除两端引物,经Clustal X比对后,用DNAsp分析各种群的单倍型多样性h,核苷酸多样性Dij,基因流Nm和种群分化度FST。用Arlequin软件进行分子变异分析(AMOVA),求算种群间及种群内的变异方差分布。
作者贡献
杨明照负责实验的设计,具体实施,数据处理及论文的撰写,喻君生负责实验材料的采集,参与实验的操作,应站明负责实验材料的采集和论文的修改。
致谢
感谢国家自然科学基金(30771763, 30170101)对本研究的资助,感谢贵州赤水桫椤国家级自然保护区的大力支持,感谢中山大学苏应娟教授的指导,感谢王伯荪教授对桫椤的鉴定,感谢匿名评审专家的意见和修改建议。
Avise J.C., eds., 2000, Phylogeography. Harvard University Press, Cambridge, pp.5-10
Bennett K.D., 1990, Mlankovitch Cycles and their effects on species in ecological and evolutionary time, Paleobiology, 16(1): 11-21
Cheng Z.Y., Tao G.D., and Xu Z.F., 1990, A study on the biological characteristics and the endangering factors of Alsophila spinulosa, ActaBotanica Yunnanica,12(2): 186-190 (程志英, 陶国达, 许再富, 1990, 桫椤濒危原因的探讨, 云南植物研究, 12(2): 186-190)
Chiang T.Y., Schaal B.A., and Peng C.I., 1998, Universal primers for amplification and sequencing a noncoding spacer between atpB and rbcL genes of chloroplast DNA, Bot. BullAcad. Sinica, 39: 245-250
Ferris C., King R.A., Vainola R., Hewitt G.M., 1998, Chloroplast DNA recognizes three refugia sources of European oaks and suggests independent eastern and western immigrations to Finland, Heredity, 80: 584-593
Huang S., Chiang Y.C., Schaal, B.A., Chou C.H., Chang T.Y., 2001, Organelle DNA phylogeography of Cycas taitungensis, a relict species in Taiwan, Mol. Ecol., 10(11): 2669-2681
Hwang S.Y., Lin T.P., Ma C.S., Lin C.L., Chung J.D., Yang J.C., 2003, Postglacial population growth of Cunninghamia konishii(Cupressaceae) inferred from phylogeographical and mismatchanalysis of chloroplast DNA variation, Mol. Ecol., 12: 2689-2695
Lucansky W., 1974, Comparative studies of the nodal and vascular anatomy in the neotropical CyatheaceaeⅡ. Squamate genera, Amer. Jour. Bot., 61: 472-480
Lu S.Y., Hong K.H., liu S.L., Cheng Y.P., Wu W.L,, Chiang T.Y., 2002, Genetic variation and population differentiation of Michelia formosana (Magnoliaceae) based on cpDNA variation and RAPD fingerprints: relevance to post-Pleistocene recolonization. Journal of plants research, 115(3): 203-216
Schaal B.A., Hayworth D.A., Olsen K.Mn., Rauscher J.T., and Smith W.A., 1998, Phylogeographic studies in plants: problems and prospects, Mol. Ecol., 7(4): 465-474
Su Y.J., Wang T., Yang W.D., Huang C., and Fan G.K., 1998, DNA extraction and RAPD analysis of Podocarpus, Zhong-shan Daxue Xuebao, 37(4): 13-18 (苏应娟, 王艇, 杨维东, 黄超, 范国宽, 1998, 罗汉松属植物DNA的提取和RAPD分析, 中山大学学报, 37(4): 13-18)
Trewick S.A., Morgan-RichardsM., Russell S.J., derson S., Rurnsey F.J., Pinter I., Barrett J.A., Gibby M., and Vogel J.C., 2002, Polyploidy, phylogeography and Pleistocene refugia of the rockfern Asplenium ceterach: evidence from chloroplast DNA, Mol. Ecol., 11: 2003-2012
Tryon R.M., 1970, The classification of Cyatheaceae, Contrib Gray Herb, 200: 3-53
Willis K.J., McElwain J.C., eds., 2002, The Evolution of Plants, New York, Oxford University Press, pp.22
Zhang Y.F., Wan L.X., Xu M., and Hou D.B., 2008, A modified CTAB Extraction and inspection for genome of aconitum carmichael, Hubei Agricultural Sciences, 47(1): 12-14 (张岳峰, 万里翔, 徐敏, 侯大斌, 2008, 乌头DNA的改良CTAB提取和ISSR检测, 湖北农业科学, 47(1): 12-14)
.png)
.png)


.png)