2内蒙古自治区中蒙药材规范化生产工程技术研究中心, 呼和浩特, 010021
 作者
作者  通讯作者
通讯作者
植物药与药理学杂志, 2012 年, 第 1 卷, 第 7 篇
收稿日期: 2012年12月06日 接受日期: 2012年12月16日 发表日期: 2012年12月27日
被子植物线粒体cox1基因的GroupI内含子常含有水平基因转移(horizontal gene transfer, HGT)现象。本研究对药用寄生植物锁阳(Cynomorium songaricum)线粒体基因cox1、cox2、nad1序列进行扩增分析,利用最大似然法对cox1和cox2基因内含子和外显子分别进行聚类分析,结果显示cox1内含子聚类结果与其他3个聚类结果不相符,说明该内含子可能存在水平基因转移现象。对比了锁阳6个不同居群间线粒体基因cox1、cox2、nad1外显子与内含子序列平均距离,发现6个不同居群间cox1基因内含子同源性高达100%,说明该基因内含子可能具有功能。结合生物信息学分析表明该内含子很有可能编码核酸内切酶,而DNA核酸内切酶被认为能促进内含子的转移。3'Co-conversion tracts(CCT)是被子植物内含子发生转移的痕迹, 对锁阳cox1基因序列进行检测发现其含有3'CCT序列且长度为35bp,RT-PCR实验验证得到cox1基因内含子可以独立转录mRNA。因此,锁阳cox1基因内含子很可能编码一个核酸内切酶,且促进了其内含子的转移。
水平基因转移(HGT),又称横向基因转移或侧向基因转移(lateral gene transfer, LGT), 是指没有生殖关系的不同物种之间,或叶绿体、线粒体等细胞器之间,以及细胞器和细胞核之间DNA片段的流动(Ochman, 2001; 欧剑虹等, 2003; 李志江等, 2008)。线粒体基因HGT主要在高等植物之间(Chang et al., 2011; Stegemann et al., 2012)发生。最初的研究认为HGT很大程度上只在原核生物中发生,现在发现它也是真核生物基因组进化中的一个重要的力量(Won and Renner, 2003; Davis and Wurdack, 2004)。
大多数的陆地植物和藻类都含有GroupI或GroupII内含子(Hepburn et al., 2012)。GroupI内含子主要存在于细胞器(如线粒体等)、细菌及某些低等真核细胞中,如植物线粒体基因cox1内含子,此类内含子常含有HGT现象。研究发现几乎所有的寄生植物线粒体DNA都被GroupΙ内含子入侵,该内含子可能是由一种真菌转移到被子植物中,然后在植物间进行传播(Barkman et al., 2007)。cox1基因内含子可能编码一个核酸内切酶并促进其内含子的转移(Sanchez-Puerta et al., 2011),但目前这个机制还只是推测,尚未有实验进行过验证。GroupII内含子也常见于细胞器(如线粒体等)及细菌中,如cox2、nad1基因内含子属于GroupII内含子(Keren et al., 2012),大多数GroupII内含子不会自我剪接,它需要蛋白质剪接因子的帮助(Ahlert et al., 2006)。
锁阳是常见的中药和蒙药(岳鑫等, 2013; Liu et al., 2013),常寄生于蒺藜科(Zygophyllaceac)白刺属(Nitraria L.)植物的根部。Sanchez-Puerta等(2011)提到锁阳cox1基因缺乏内含子但存在3'Co-conversion tracts(CCT)序列。研究表明3'CCT很大程度上是被子植物内含子发生转移的痕迹(Vaughn et al., 1995; Sanchez-Puerta et al., 2011)。但目前关于锁阳cox1基因内含子HGT现象的研究均未见报道。所以我们选取锁阳为实验材料,对锁阳的6个不同居群间cox1、cox2、nad1基因序列进行分析,旨在揭示锁阳不同居群间cox1基因内含子的存在情况和GroupI内含子参与水平基因转移的机制。
1结果与分析
1.1系统发育分析
锁阳的分类地位仍未明确,根据形态学和核基因SSU、线粒体基因LSU和叶绿体基因rbcL、atpB和matK的聚类分析表明,锁阳属在分子聚类系统中定位于虎耳草目(Nickrent et al., 2005),而用叶绿体IR区基因聚类分析表明,锁阳在分子聚类系统中位于蔷薇目(Zhang et al., 2009)。我们使用MEGA5软件对线粒体cox1、cox2基因的内含子和外显子序列分别与NCBI数据库收录的相应基因序列构建聚类树(图1)。锁阳cox2基因内含子和外显子聚类结果相一致,均与十字花科的拟南芥属和芸薹属处于同一分支,与大戟属相近。而cox1基因的内含子与其外显子的聚类结果不一致,cox1基因外显子的聚类结果与cox2基因内含子和外显子的聚类结果相似,锁阳与大戟属相近。cox1基因内含子聚类结果显示,锁阳与寄生花属、素馨属处于同一分支,与天南星属相近,其中天南星属为单子叶植物。所以锁阳cox1基因内含子的系统发育关系与cox1外显子、cox2内含子和外显子的系统发育关系不一致。因此,cox1基因内含子聚类结果表明其内含子可能发生了转移。
|
图 1 被子植物cox1和cox2基因内含子和外显子序列使用最大似然法构建的系统发育树 Figure 1 Maximum likelihood phylogeny of angiosperm cox1,cox2 gene introns and exons |
1.2遗传距离比较分析
将锁阳6个不同居群间cox1、cox2、nad1基因各自的外显子与内含子平均遗传距离进行比较。结果如表1所示,cox1基因内含子与外显子序列平均距离值都为0,而cox2基因内含子与外显子序列的平均距离值较大,分别为254.9333和68.9333,nad1基因内含子序列的平均距离值为320.3333(值越小表示基因越保守,越大表示差异性越大)。利用NCBI的Blast在线对6个不同居群间cox1基因内含子序列进行两两同源性检索结果显示均为100%。
遗传距离的大小通常反映遗传多样性的高低,种内个体间遗传距离越小,彼此亲缘关系越近,则遗传多样性越低;反之,遗传距离越大则遗传多样性越高。由表1可知,锁阳6个不同居群间nad1基因内含子和cox2基因的内含子和外显子遗传距离较大,说明cox2和nad1基因的遗传多样性相对较高,在进化过程中其遗传变异性较大,进化较快;而cox1基因的内含子和外显子遗传距离小,说明其遗传多样性较低,该基因在进化过程中的遗传变异性小,进化慢,且6个不同居群间cox1基因的内含子如此高的同源性,表明该基因内含子可能具有功能。
|
表 1 锁阳cox1, cox2和nad1基因6个不同居群间内含子与外显子序列的整体平均距离 Table 1 Compute overall mean distance of intron and exon sequences of cox1, cox2 and nad1 genes between 6 different populations |
1.3生物信息学分析
使用ExPASy-Translate Tool将锁阳线粒体基因cox1基因内含子序列(约900 bp)翻译成氨基酸序列,利用BLAST-protein blast在线对其进行同源性检索显示(图2),cox1基因内含子氨基酸序列与楝科樫木属核酸内切酶、葫芦科西瓜属核酸内切酶、鼠李科马甲子属核酸内切酶、鼠李科欧鼠李核酸内切酶、 姜科舞花姜属核酸内切酶、姜科山柰属核酸内切酶、夹竹桃科玫瑰树属核酸内切酶的同源性分别高达96%、95%、94%、94%、94%、93%和94%,表明cox1基因内含子氨基酸序列与核酸内切酶序列(endonuclease)很相似。
使用Pfam数据库进行比对,发现有两个Pfam-A matches (有意义链)与cox1基因内含子氨基酸序列序列相匹配,分别为LAGLIDADG-1 (Clan: CL0324; Envelope: 52-141; Alignment: 52-140; HMM: 1-101; HMM length:102; Bit score: 47.3; e-value: 2.3e-12)和LAGLIDADG-1 (Clan: CL0324; Envelope:;158-249; Alignment: 158-245; HMM: 1-98; HMM length: 102; Bit score: 36.1; e-value: 6.9e-09),都属于LAGLIDADG endonuclease。Pfam数据库分析表明cox1基因内含子氨基酸序列都与核酸内切酶相匹配。因此认为,锁阳cox1基因内含子很有可能编码核酸内切酶。
|
图 2 锁阳cox1基因内含子的氨基酸序列与不同植物核酸内切酶氨基酸序列的比对 Figure 2 Amino acid sequence alignment of Cynomorium cox1 intron gene and Endonuclease of other plant species |
1.4 3'Co-conversion tracts (3'CCT)分析
一般认为同源内含子很有可能发生转移,由于该位点常常编码DNA核酸内切酶,能促进内含子的转移,途径为:若转移来的序列为含有内含子的基因序列,该内含子编码核酸内切酶,则核酸内切酶识别并剪切不含内含子的同源序列,通过双链断裂修复途径,以含内含子基因序列为模版,在连接酶和解离酶帮助下,完成双链修复,在DNA修复复制时, 内含子两侧的外显子的部分序列也常常被复制到受体DNA中,这部分外显子称为Co-conversion tracts (CCT) (谢君等, 2001; Sanchez-Puerta et al., 2011) (图3)。且3'CCT很大程度上是被子植物内含子发生转移的痕迹(Vaughn, 1995; Sanchez-Puerta, 2011)。我们将cox1基因古老序列、欧洲锁阳和锁阳6个不同居群的部分序列(内含子上下游的部分外显子)进行对比(图4)。结果显示6个不同居群锁阳cox1基因均含有内含子和3' CCT,长度为35 bp,且古老被子植物序列不含内含子和3'CCT,表明其内含子可能携带部分外显子序列发生了水平基因转移。
|
图 3 锁阳cox1部分基因序列及引物示意图 Figure 3 Schematic of Cynomorium songaricum cox1 partial gene sequences and primer |
|
图 4 3' Co-conversion tracts(CCT)分析 Figure 4 Analysis of 3'Co-conversion tracts (CCT) |
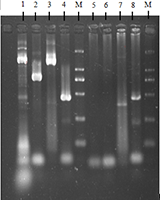
1.5 cox1内含子RT-PCR分析
提取锁阳总RNA,利用紫外分光光度计检测其OD260与OD280的比值为1.86,浓度为297.1 ng/μL,说明RNA纯度较高,可用于后续实验(图5)。以cox2基因内含子为模版设计引物对cox2iF和cox2iR:以DNA为模版,扩增条带约1 400 bp,与该引物所对应的cox2基因序列长度一致;以cDNA为模版,没有扩增出序列,一方面说明cox2基因在反转录成为mRNA的过程中内含子被剪切了,另一方面也证明本实验所使用RNA样品较纯,无DNA污染。因此,该RT-PCR实验中cox2基因的扩增结果可以作为一个对照。
此外,我们在cox1基因外显子处设计引物对cox1eF和cox1eR (图3; 表2),以DNA为模板,扩增序列长度约1 250 bp,与该引物所对应的cox1基因序列长度一致;以cDNA为模版,扩增序列约350 bp序列,这说明反转录过程中内含子被剪切,扩增出的350 bp产物为剪切后剩余的外显子序列。在cox1基因内含子处设计引物对cox1iF和cox1iR,以DNA为模版,扩增条带长约480 bp,与该引物所对应的cox1基因序列长度一致;以cDNA为模版,扩增条带长约480 bp,说明该引物所包含的cox1基因内含子可以转录mRNA。再用上游引物位于cox1基因外显子区域,下游引物位于内含子区域的引物对cox1eF和cox1iR (图3; 表2),以DNA为模版,扩增条带约800 bp,与该引物所对应的cox1基因序列长度一致;以cDNA为模版没有扩增出序列,说明cox1基因内含子部分被剪切掉了,下游引物没有结合上去,所以无扩增产物,说明cox1基因内含子转录的mRNA并不与外显子相连,应该是独立转录。
根据RT-PCR结果,cox1基因内含子可以转录mRNA,并比外显子的扩增条带亮,说明内含子在表达量上与外显子存在差异,更进一步证明cox1基因外显子和内含子的转录是两个独立的过程。我们对扩增出的cox1基因内含子的cDNA进行测序,结果显示它与NCBI数据库中存在的cox1基因内含子序列一样,该结果符合先前的生物信息学预测,即cox1基因内含子可以独立转录mRNA,且很有可能编码具有功能的核酸内切酶。
|
图 5 以锁阳cDNA和DNA为模版进行PCR扩增电泳图 Figure 5 Diagram of used C. songaricum DNA and cDNA as template for PCR amplification |
2讨论
植物线粒体基因组发生水平基因转移是非常普遍的,其中发生最多的是GroupI内含子的转移(Cho and Palmer, 1999; Sanchez-Puerta et al., 2011)。因此线粒体基因cox1内含子的转移现象更成了研究热点。Cho和Palmer (1999)将单子叶植物天南星科(Araceae) 14个属的cox1基因通过Southern杂交技术进行分析,发现6个属的cox1基因中含有GroupI内含子,通过进化树分析表明这6个属的内含子是通过水平转移得到的。
本研究对锁阳cox1、cox2、nad1基因内含子和外显子序列进行了聚类分析,聚类结果表明cox1基因内含子与其系统发育关系不一致,说明cox1基因内含子可能存在转移现象。基因进化速率具有不均一性,与相邻的外显子相比,内含子更容易发生碱基替换(Laroche et al., 1997)。内含子序列在进化过程中变异程度非常高,要高于外显子,由于其不受选择压力作用或选择压力小(欧剑虹等, 2003)。6个不同居群cox1基因的内含子具有如此高的同源性,可能与其内含子具有功能相关。
生物信息学分析预测,该内含子有可能编码核酸内切酶。Vaughn等(1995)表明草胡椒属cox1基因内含子可能是通过水平基因转移从真菌获得。核酸内切酶催化内含子的整合,识别目标序列,通过双链断裂修复途径,3' CCT序列也常常被复制到受体DNA中(谢君等, 2001),成为被子植物内含子发生转移的痕迹(Vaughn et al., 1995; Sanchez-Puerta et al., 2011)。Cho等(1998)和Cusimano等(2008)研究表明cox1基因标准的3' CCT长度为3~21 bp,而Sanchez-Puerta等(2011)对此提出了质疑,他们认为锁阳3' CCT的最小长度为35 bp,欧洲锁阳的3'CCT的最小长度为78 bp,古老被子植物cox1基因无3' CCT序列。而本实验3'CCT检测表明6个不同居群锁阳cox1基因均存在3' CCT序列,且3' CCT序列长度均为35 bp,说明锁阳cox1基因的内含子序列和部分外显子序列很可能是从外源转移而来的,且Sanchez-Puerta等(2011)研究表明锁阳cox1基因不含有内含子,古老被子植物cox1基因也无内含子,而我们的结果表明锁阳cox1基因存在内含子,说明锁阳cox1基因可能处于转移的过程中。RT-PCR实验结果显示内含子可以转录mRNA,因此锁阳cox1基因内含子很可能编码一个核酸内切酶,该核酸内切酶很有可能促进了其内含子的转移。
3材料与方法
3.1材料
本实验所用的锁阳分别采自内蒙古自治区阿拉善盟额济纳旗(101.07'E, 41°97'N)和鄂尔多斯市杭锦旗(108.74'E, 39°84'N)、青海省海西蒙古族藏族自治州(93.37'E, 37°38'N)、宁夏回族自治区石嘴山市平罗县(106.55'E, 38°91'N)、甘肃省武威市民勤县(103.92'E, 38.67'N)和蒙古国南戈壁省Ummugobiaimag (42°24′6″E, 107°34′1″N )6个居群,风干后-80℃储存。
3.2方法
3.2.1锁阳总DNA提取
使用TIANGEN Plant Genomic DNA kit。
3.2.2 PCR及测序方法
在梯度PCR仪上完成聚合酶链式反应(PCR)。反应总体积25 μL,加入TaKaRa LA TaqTM高保真酶扩增。反应体系如下:cox1基因:H2O 12 μL、Buffer 2.5 μL、dNTP 4 μL、Forward primer 2 μL、Reverse primer 2 μL、DNA2 μL、LA Taq 0.5 μL。94℃预变性5 min;94℃变性30 s,58℃退火45 s,72℃延伸2.5 min,35个循环;72℃保温10 min。nad1、cox2基因:H2O 12 μL、Buffer 2.5 μL、dNTP 4 μL、Forward primer 2 μL、Reverse primer 2 μL、DNA 2 μL、LA Taq 0.5 μL。94℃预变性5 min;94℃变性30 s,58℃退火45 s,72℃延伸2 min,35个循环;72℃保温10 min。引物见表2。PCR扩增的产物经纯化后由上海英潍捷基有限公司进行测序。
3.2.3锁阳总RNA提取、纯化及检测
使用TIANGEN RNAprep Pure Plant Kit (多糖多酚植物总RNA提取试剂盒)提取锁阳总RNA,TIANGEN RNAclean Kit及DNaseI纯化RNA。利用分光光度计进行RNA定量及纯度检测,利用甲醛变性凝胶电泳检测RNA的完整性。
3.2.4反转录实验(PT-PCR)
采用TAKARA PrimeScriptTMRT reagent Kit with gDNA Eraser去除基因组DNA,反应条件:42℃ 2 min,4℃保存。然后以RNA为模版反转录为cDNA,反转录反应条件:37℃ 15 min,85℃ 5 s,4℃。再以cDNA为模板扩增目的片段,引物见表2,反应体系:H2O 13 μL、Buffer 2.5 μL、dNTP 4 μL、Forward primer 2 μL、Reverse primer 2 μL、cDNA1 μL、LA Taq 0.5 μL,反应总体积25 μL。反应条件:94℃预变性5 min;94℃变性30 s,58℃退火45 s,72℃延伸2.5 min,35个循环;72℃保温10 min。
|
表 2 基因扩增所用引物 Table 2 Primers used for gene amplification |
3.3序列分析方法
3.3.1系统发育进化分析
使用杭锦旗锁阳DNA为模版,引物cox1F和cox1R扩增cox1基因序列(含内含子序列);引物cox2iF和cox2iR扩增cox2内含子基因序列;引物cox2eF和cox2eR扩增cox2外显子基因序列。测序所得的cox1、cox2、nad1基因序列用BLAST:Basic Local Alignment Search Tool进行比对。从NCBI数据库中选取具有代表性植物的cox1、cox2基因序列(图1),利用ClustalX将序列进行对齐,参数设置使用默认值(Thompson et al., 1997),使用MEGA5软件最大似然法(maximum likelihood tree)构建系统发育树(蒙姣荣等, 2014),参数bootstrap值设置为1 000 (Felsenstein, 1985)。
3.3.2遗传距离比较分析
使用cox1F和cox1R,cox2iF和cox2iR,cox2eF和cox2eR,nad1F和nad1R四对引物扩增锁阳6个居群的cox1、cox2、nad1基因。使用ClustalX软件分别将锁阳6个居群的cox1、cox2、nad1三个基因的内含子和外显子序列进行对齐,使用MEGA5软件分别对6个居群的3个基因内含子和外显子序列进行整体平均距离(compute overall mean distance)预测,分析三个基因的变异程度。
3.3.3生物信息学分析
使用ExPASy-Translate Tool (http://www.expasy.org/tools/dna.html)在线对cox1基因内含子序列进行翻译,利用BLAST在线对其进行同源性检索,ClustalX对比,BoxShade着色,使用Pfam在线对其功能结构域进行预测。
3.3.4 3'Co-conversion tracts (CCT)分析
参照Sanchez-Puerta等(2011)方法,对cox1基因古老序列、欧洲锁阳(Cynomorium coccineum)和锁阳6个不同居群间cox1基因的内含子及内含子邻近的部分外显子序列进行3' CCT分析。
作者贡献
苏小娟是本研究的实验设计和实验研究的执行人,完成数据分析和论文初稿的写作;刘广达参与实验设计和论文的修改;刘英和冯晓宇参与部分实验分析和论文修改;陈贵林是本项目负责人,负责实验设计和论文修改及定稿。全体作者均已阅读并同意提交本稿。
致谢
本研究由国家自然科学基金(31260117)和国家科技支撑计划课题(2011BAI07B07)共同资助。
Ahlert D., Piepenburg K., Kudla J., and Bock R., 2006, Evolutionary origin of a plant mitochondrial group II intron from a reverse transcriptase/maturase-encoding ancestor, Journal of Plant Research, 119(4): 363-371
Barkman T.J., McNeal J.R., Lim S.H., Coat G., Croom H.B., Young N.D., and Depamphilis C.W., 2007, Mitochondrial DNA suggests at least 11 origins of parasitism in angiosperms and reveals genomic chimerism in parasitic plants, BMC Evolutionary Biology, 7(1): 248
Chang S.X., Yang T.T., Du T.Q., Huang Y.J., Chen J.M., Yan J.Y., He J.B., and Guan R.Z., 2011, Mitochondrial genome sequencing helps show the evolutionary mechanism of mitochondrial genome formation in Brassica, BMC Genomics, 12(1): 497
Cho Y., and Palmer J.D., 1999, Multiple acquisitions via horizontal transfer of a group I intron in the mitochondrial cox1 gene during evolution of the Araceae family, Molecular Biology and Evolution, 16(9): 1155-1165
Cho Y., Qiu Y.L., Kuhlman P., and Palmer J.D., 1998, Explosive invasion of plant mitochondria by a group I intron, Proceedings of the National Academy of Sciences of the United States of America, 95(24): 14244-14249
Cusimano N., Zhang L.B., and Renner S.S., 2008, Revaluation of the cox1 group I intron in Araceae and angiosperms indicates a history dominated by loss rather than horizontal transfer, Molecular Biology and Evolution, 25(2): 265-276
Davis C.C., and Wurdack K.J., 2004, Host-to-parasite gene transfer in flowering plants: phylogenetic evidence from Malpighiales, Science, 305(5684): 676-678
Hepburn N.J., Schmidt D.W., and Mower J.P., 2012, Loss of two introns from the Magnolia tripetala mitochondrial cox2 gene implicates horizontal gene transfer and gene conversion as a novel mechanism of intron los, Molecular Biology and Evolution, 29(10): 3111-3120
Felsenstein J., 1985, Confidence limits on phylogenies: an approach using the bootstrap, Evolution, 39(4): 783-791
Keren I., Tal L., des Francs-Small C.C., Araújo W.L., Shevtsov S., Shaya F., Fernie A.R., Small I., and Ostersetzer-Biran O., 2012, nMAT1, a nuclear‐encoded maturase involved in the trans-splicing of nad1 intron 1, is essential for mitochondrial complex I assembly and function, Plant Journal, 71(3): 413-426
Li Z.J., Li H.Q., and Diao X.M., 2008, Methods for the identification of horizontal gene transfer (HGT) events and progress in related fields, Yichuan (HEREDITAS), 30(9): 1108-1114 (李志江, 李还权, 刁现民, 2008, 基因水平转移的评判方法和转移方式的研究进展, 遗传, 30(9): 1108-1114)
Liu G.D., Chen G.L., Li W., and Li C.X., 2013, Genetic and phytochemical diversities of Cynomorium songaricum Rupr. in Northwest China indicated by ISSR markers and HPLC-fingerprinting, Biochemical Systematics and Ecology, 48: 34-41
Laroche J., Li P., Maggia L., and Boudquet J., 1997, Molecular evolution of angiosperm mitochondrial introns and exons, Proceedings of the National Academy of Sciences of the United States of America, 94(11): 5722-5727
Meng J.R., Ouyang Q.F., Cen Z.L., Zhu L.L., Li J.Q., Zhu F.R., and Chen B.S., 2014, Pathogenicity and genetic diversity of the pathogens causing mulberry bacterial wilt in guangxi province, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 33(5): 998-1006 (蒙姣荣, 欧阳秋飞, 岑贞陆, 朱丽玲, 李界秋, 朱方荣, 陈保善, 2014, 广西桑树细菌性枯萎病致病力和遗传多样性分析, 基因组学与应用生物学, 33(5): 998-1006)
Nickrent D.L., Der J.P., and Anderson F.E., 2005, Discovery of the photosynthetic relatives of the ‘‘Maltese mushroom’’ Cynomorium, BMC Evoutionary Biology, 5: 38
Ochman H., 2001, Lateral and oblique gene transfer, Current Opinion in Genetics & Development, 11(6): 616-619
Ou J.H., Xie Z.X., Chen X.D., Ni L.N., and Shen P., 2003, Horizontal Gene Transfer, Yichuan (HEREDITAS), 25 (5): 623-627 (欧剑虹, 谢志雄, 陈向东, 倪丽娜, 沈萍, 2003, 水平基因转移, 遗传, 25(5): 623-627)
Stegemann S., Keuthe M., Greiner S., and Bock R., 2012, Horizontal transfer of chloroplast genomes between plant species, Proceedings of the National Academy of Sciences of the United States of America, 109(7): 2434-2438
Sanchez-Puerta M.V., Abbona C.C., Zhuo S., Tepe E.J., Bohs L., Olmstead R.G, and Palmer J.D., 2011, Multiple recent horizontal transfers of the cox1 intron in Solanaceae and extended co-conversion of flanking exons, BMC Evolutionary Biology, 11(1): 277
Thompson J.D., Gibson T.J., Plewniak F., Jeanmougin F., and Higgins D.G., 1997, The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools, Nucleic Acids Research, 25(24): 4876-4882
Vaughn J.C., Mason M.T., Sper-Whitis G.L., Kuhlman P., and Palmer J.D., 1995, Fungal origin by horizontal transfer of a plant mitochondrial group I intron in the chimeric cox1 gene of Peperomia, Journal of Molecular Evolution, 41(5): 563-72
Won H., and Renner S.S., 2003, Horizontal gene transfer from flowering plants to Gnetum, Proceedings of the National Academy of Sciences of the United States of America, 100(19): 10824-10829
Xie J., Liu C.Q., Huang J.F., and Shao D., 2001, Recent advance in the study of intein, Shengwu Huaxue Yu Shengwu Wuli Jinzhan (Progress in Biochemistry and Biophysics), 28(2): 184-187 (谢君, 刘次全, 黄京飞, 石秀凡, 邵丹, 2001, 蛋白质内含子的特征、转移和演化, 生物化学与生物物理进展, 28(2): 184-187)
Yue X., Duan Y.Y., and Chen G.L., 2013, In vitro callus induction, proliferation and adventitious root organogenesis of Cynomorium songaricum, Zhiwu Shengli Xuebao (Plant Physiology Journal), 49(12): 1421-1426 (岳鑫, 段园园, 陈贵林, 2013, 锁阳愈伤组织诱导和增殖及不定根分化,植物生理学报, 49(12): 1421-1426)
Zhang Z.H., Li C.Q., and Li J.H., 2009, Phylogenetic placement of Cynomorium in Rosales inferred from sequences of the inverted repeat region of the chloroplast genome, Journal of Systematics and Evolution, 47(4): 297-304
.png)
.png)
.png)
.png)
.png)
.png)