 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 46 篇 doi: 10.5376/mpb.cn.2011.09.0046
收稿日期: 2010年12月22日 接受日期: 2011年04月07日 发表日期: 2011年04月15日
黄文静等, 2011, SRAP和RGA标记技术在甘薯的抗茎线虫病遗传多样性中的应用, 分子植物育种 Vol.9 No.46 (doi: 10.5376/mpb.cn.2011.09.0046)
为了评价甘薯种质的抗茎线虫病遗传多样性,选用SRAP和RGA分子标记方法,SRAP和RGA引物设计如下:利用Blast软件,对Genbank数据库中由王钰等克隆的342个甘薯RGA(编号为DQ903322-DQ903664)与抗线虫病基因(RGC1、RX)进行比对,筛选出与抗病基因同源性高于50%的RGA序列11个,以高抗甘薯茎线虫病品种徐78-1和感茎线虫病品种徐薯18的基因组DNA为亲本,基因组DNA采用改良的CTAB法提取得到,然后将没有多态性的RGA结合SRAP组合成SRAP-RGA引物,筛选出五对有多态性的SRAP-RGA引物。将SRAP和RGA技术结合起来研究50个甘薯品种的抗茎线虫病遗传多样性。结果显示,5对引物组合经过PCR扩增,聚丙烯酰胺凝胶电泳检测共产生57条带,平均每对引物组合产生9~13条带,其中多态性条带49条,占总条带数的85.96%。其中多态率最高的引物对是EM4-S2-A07-F,为100%。最低的引物对是EM8-S1-F03-F,为63.6%。根据SRAP-RGA标记结果,应用UPGMA法,采用NTSYS-pc软件对50个甘薯种质进行聚类分析,它们之间的相似系数范围在0.2~0.8之间。UPGMA聚类分析显示,在相似系数为0.58处50份甘薯品种被划分为3大类群。这说明SRAP和RGA标记技术可有效用于甘薯的抗病遗传多样性研究,并为杂交亲本的选择提供理论依据。
甘薯(Ipomoea batatas (L.) Lam.)属于旋花科(Convolvulaceae)双子叶植物(Austin, 1988),广泛种植于世界上100多个国家。中国是世界上最大的甘薯生产国家,每年种植面积和生产量分别占世界的69.4%和84.6%(刘庆昌, 2004)。近年来甘薯病害频发,其中以甘薯茎线虫病最为严重,成为制约甘薯生产的瓶颈问题。抗病品种的选育和推广是控制该甘薯茎线虫病的主要手段。抗病育种主要有常规育种和分子育种。分子育种主要包括抗病基因(候选基因)克隆、分子标记筛选等。
目前,应用于甘薯的以PCR为核心的分子标记技术主要包括RAPD(Randomly Amplified Polymorphic DNA)、AFLP(Amplified Fragment Length Polymorphism)(Kriegner et al., 2003; 张大普等, 2004)、SSR(Simple Sequence Repeat)、SRAP (Sequence Related Amplified Polymorphism) (郝玉民等, 2007)、RGA(Resistance Gene analogue)(林巧玲, 2005)等。由于RAPD技术重复性差、AFLP技术复杂、成本昂贵等的缺点,限制了其在甘薯育种上的广泛应用。
SRAP即序列相关扩增多态性技术(Sequence Related Amplified Polymorphism),是由美国加州大学Li和Quiros博士(Li and Quiros, 2001)于2001年提出来的一种新型的基于PCR的DNA分子标记系统。它利用一个17bp的正向引物和18bp的反向引物对生物全基因组进行扩增,通过分析不同材料的SRAP扩增图谱便可观察分析材料间的遗传差异。SRAP以其高度多态性(张四普等, 2008)、操作简便迅速、成本低、可靠性好、重复性高、易于测序等特点广泛应用于植物遗传多样性分析(Ferriol et al., 2003)、遗传图谱构建(Pan et al., 2010)、基因定位克隆、比较基因组学(Li et al., 2003)等方面。
RGA即抗病基因同源序列(Resistance Gene analogue),是克隆植物抗病基因的一条新途径。其原理是根据己克隆植物抗病基因的保守结构域设计简并引物, 扩增获得RGAs, 然后分析RGAs与抗病基因的关系, 确定候选抗病基因从而获得新的抗病基因(徐兵强等, 2004)。自1996年Kanazin和Yu首次报道用抗病基因产物的保守序列为引物扩增RGA以来,RGA不仅应用于克隆和定位R基因,还用于分子标记辅助育种及种质资源多样性研究等(肖天霞, 2006)。
目前,郝玉民等(郝玉民等, 2007)已利用SRAP技术对36个甘薯品种进行了遗传多样性分析,吴洁等(吴洁等, 2007)也利用SRAP标记技术分析了来自四川省的甘薯种质资源的亲缘关系,但将SRAP和RGA技术结合起来研究甘薯的抗病遗传多样性却鲜有报道。本研究选用SRAP标记技术结合RGA技术对甘薯抗茎线虫病进行分子聚类,为抗茎线虫病育种和优良种质资源的筛选提供一定的理论依据。
1结果与分析
1.1 SRAP-RGA扩增产物的多态性分析
本实验选用筛选出的5对带型清晰、多态性较好的引物组合,利用高感品种徐18和高抗品种徐781为亲本对照,对50份供试甘薯材料进行扩增,所扩增条带的大小一般在250 bp~2 000 bp之间,每对引物组合产生9~13条谱带,共产生谱带57条,其中多态性条带为49条,占总带数的85.96% (表1)。不同的引物对产生的多态率明显不同(Kriegner et al., 2003)。最高是EM4-S2-A07-F引物对的多态率为100%,最低是EM8-S1-F03-F引物对的多态率为63.6%。部分扩增结果如图1所示。
|
表1 各引物对的总带数和多态性条带数 Table 1 The total and polymorphic fragments in all cultivars |
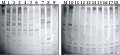 图1 部分SRAP-RGA扩增结果图谱 注: M: 2 000 bp marker; 1: 红冬; 2: 徐55-1; 3: 44104; 4: 徐22; 5: 徐23; 6: 青农2号; 7: 宁紫1号; 8: 潮薯; 9: 渝紫1号; 10: 徐紫糯; 11: 水果甘薯; 12: 徐55-2; 13: 台农69; 14: 559; 15: 胜利百号; 16: 徐55-1; 17: 44104; 18: 徐55-2 Figure 1 The profile of SRAP-RGA amplification Note: M: 2 000 bp marker; 1: Hongdong; 2: Xu55-1; 3: 44104; 4: Xu22; 5: Xu23; 6: Qingnong2; 7: Ningzi1; 8: Chaoshu; 9: Yuzi1; 10: Xuzinuo; 11: Shuiguoganshu; 12: Xu55-2; 13: Tainong69; 14: 559; 15: Shenglibaihao; 16: Xu55-1; 17: 4 04; 18: Xu55-2 |
1.2 聚类分析结果
根据SRAP-RGA标记对50个甘薯品种扩增结果进行数据统计,应用UPGMA法对50个品种进行聚类分析,结果见图2。
 图2 SRAP-RGA技术扩增甘薯基因组的聚类分析图 Figure 2 The dendrogram of the sweetpotato based on SRAP and RGA marker analysis |
结果显示:在相似系数为0.58时,将50个品种划分为3大类群(图2)。第一类包括32个品种:皖702、二黄、199029-1、559、南薯88、美Ⅰ、徐55-2、川山紫、广菜2号、青农2号等;第二类包括12个品种:美国红薯、44104、水果甘薯、潮薯、宁紫1号、徐紫13-4、徐23、济薯18、徐紫薯、徐18等;第三类包括6个品种:徐22、C-14、59、潮薯1号、福薯11号、徐78-1。
1.3 SRAP-RGA扩增产物的抗茎线虫病遗传多样性分析
根据多态性条带统计结果,采用NTSYS软件计算出甘薯种质间的相似系数,结果表明,50份材料间的相似系数范围较广,在0.2~0.8之间。在所有材料中,徐紫薯(48)和二黄(2)、199029-1(3)遗传相似性系数最小,为0.2,说明他们之间的遗传距离较远,而鲁78066(12)和徐781(50)、徐55-2(7)和徐55-1(15)的相似性系数较大,为0.8,说明他们之间的遗传距离较近,而且徐55-2(7)和徐55-1(15)均感甘薯茎线虫病(周忠等, 2005)。
2讨论
2.1甘薯种质资源间的抗茎线虫病遗传多样性
通过SRAP和RGA分子标记技术对甘薯的遗传多样性进行研究,结果显示5对引物扩增出57条多态性条带,多态性比率为85.76% ,不同种质资源间的遗传相似性变异范围较大(0.2~0.8), 这些结果从分子标记的角度进一步说明了我国甘薯主要亲本遗传多样性较丰富(李强等, 2008; 贺学勤等, 2005)。
2.2 SRAP和RGA分子标记技术可有效地应用于甘薯抗茎线虫病遗传多样性研究
目前,SRAP标记技术和RGA技术已广泛应用于多种植物的遗传多样性研究,如SRAP技术应用于荔枝、青蒿、郁金等,RGA技术应用于水稻等的聚类分析。本研究将SRAP和RGA分子标记技术结合起来,选用与抗病基因同源性较高的RGAs和SRAP的引物组合,对50份甘薯的抗茎线虫病遗传多样性进行研究,5对引物扩增出57条清晰的用于多态性分析的谱带,其中多态性条带就有49条,多态性比率为85.76%。这说明SRAP和RGA标记在甘薯抗病遗传多样性研究中具有较高的应用价值。
通过SRAP和RGA技术对甘薯品种进行聚类分析(图2),结果显示在遗传系数0.58处50份甘薯种质资源被划分为三大类。徐18(49)感茎线虫病,徐78-1(50)高抗茎线虫病,和它们的遗传相似系数越高,与它们的遗传距离就越远。如品种徐55-2(7)和徐55-1(15)他们来自于共同的父母本(“苏薯6号”ד萨摩光”),两者均不抗茎线虫病(xushu18.net),聚在第一大类中,且遗传相似系数(GS)为0.8;育种材料美国红薯(18)和川山紫(25)均为国外引进种,它们虽然没有聚在一起,但它们的遗传距离表现为非常接近。品种济薯18(40)、宁紫1号(28)和徐紫薯(48)均为高产紫肉品种,且都中抗茎线虫病,聚在第二大类中。品种鲁78066(12)和徐781(50)都高抗甘薯茎线虫病,虽然没有聚在一起,但相似系数很高,达到了0.8。与前人的研究结果一致(周忠等, 2005; 马代夫等, 1997)。从聚类的结果来看,美国红薯(18)和川山紫(25)等国外引进品种与我国的一些甘薯品种具有较近的亲缘关系。表明国内甘薯育种已经引进了来自国外甘薯品种资源。
本研究采用分子标记技术来研究甘薯品种的遗传多样性,揭示了50个甘薯品种之间的亲缘关系,为甘薯育种者选择杂交亲本提供理论依据。因此,在今后的甘薯育种中,考虑到亲本综合性状优良的前提下,参考聚类分析的结果,将亲缘关系较远的亲本材料杂交组合,可望获得性状优良的杂交后代。
3材料与方法
3.1植物材料
以安徽大学植物组培实验室保存的徐薯18、徐78-1等50个甘薯品种为实验材料(表2),用于基因组DNA的提取。
 表2 供试甘薯材料 Table 2 The list of the trial materials |
3.2甘薯基因组DNA的提取
基因组DNA均采用改良的CTAB法从新鲜的幼嫩叶片中提取得到(李强等, 2007)
提取的DNA经琼脂糖凝胶电泳和紫外分光光度计检测,电泳条带清楚且所提取的基因组OD260/OD280在1.8~2.0之间的DNA,置-20℃冰箱保存并用于PCR分析。
3.3引物设计
利用Blast软件,对Genbank数据库中由王钰等克隆的342个甘薯RGA(编号为DQ903322-DQ903664)(王钰等,2008)与抗线虫病基因(RGC1、RX)进行比对,筛选出与抗病基因同源性高于50%的RGA序列,以高抗甘薯茎线虫病品种徐78-1和感茎线虫病品种徐薯18的基因组DNA为模板进行PCR扩增,进行第一轮筛选。没有多态性的RGA与SRAP组合成SRAP-RGA引物。再以抗茎线虫病品种为模板进行第二轮筛选。从40对SRAP-RGA引物组合中筛选出五对有多态性的引物(表3)为抗茎线虫病聚类引物。SRAP引物序列参考Li and Quiros(2001)(Li and Quiros,2001)。
|
表3 扩增甘薯基因组的引物对 Table 3 Lists of primer pairs used to amplify genomic DNA of sweet potato |
3.4 PCR扩增和检测
根据优化实验的结果,PCR反应体系为20 μL,包括:模板DNA 100 ng,2.5×PCR Buffer 2 μL,0.5 mmol/L dNTPs,0.375 μmol/L引物,Taq DNA聚合酶1 U (上海生工生物),用ddH2O调整体系,使反应终体积为20 μL。PCR反应程序如下:94℃预变性5 min;反应前5个循环为94℃变性45s,45℃复性45s,72℃延伸l min;随后的35个循环退火温度提高到50℃,72℃延伸7 min (Wang et al., 2009)。
PCR扩增产物94℃变性3 min后,6%变性聚丙烯酰胺凝胶电泳,95V电泳1.5 h,银染检测。
3.5数据分析
对不同引物在各甘薯材料中的SRAP-RGA扩增情况做记录,统计清晰的条带,有带的记为1,无带的记为0,得到二元数据资料,形成0,1矩阵。50个品种间的遗传相似性根据Nei和Li描述的方法(Nei and Li, 1979)由Dice相似性系数计算获得。遗传距离由公式GD=1-GS计算得到。根据NTSYS-Pcversion2.1软件应用UPGMA(Xiao et al., 2008)法进行聚类分析,并采用软件处理,建立聚类图。
作者贡献
黄文静、陈达伟、季必霞是本研究的实验设计和实验研究的执行人;黄文静完成数据分析,论文初稿的写作;蒋琳参与实验设计,试验结果分析;王钰是本项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由安徽省自然科学基金(11040606M84)资助。作者感谢安徽大学提供了优越的实验条件以及仪器设备。本文中提到了我们实验中涉及的有关试剂供应商,这并非我们为这些试剂供应商的产品和服务提供推荐。
参考文献
Austin D.F., 1988, The taxonomy, evolution and genetic diversity of sweetpotato and related wild species exploration, maintenance and utilization of sweet potato genetic resources, International Potato Center, Lima, Peru., 27-61
Ferriol M., Pico B., and Nuez F., 2003, Genetic diversity of a germplasm collection of cucurbita pepo using SRAP and AFLP markers, Theor. Appl. Genet., 107(2): 271-282 doi:10.1007/s00122-003-1242-z PMid:12845442
Hao Y.M., Guo L., Han Y.C., Diao Y., Yang X.S., and Hu Z.L., 2007, Genetic diversity analysis of sweet potato based on SRAP markers, Wuhan Zhiwuxue Yanjiu (Journal of Wuhan Botanical Research), 25(4): 406-409 (郝玉民, 郭兰, 韩延闯, 刁英, 杨新笋, 胡中立, 2007, 甘薯品种的SRAP遗传多样性分析, 武汉植物学研究, 25(4): 406-409)
He X.Q., Liu Q.C., Wang Y.P., and Zhai H., 2005, Analysis of genetic diversity of sweetpotato landraces in China, Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 38(2): 250-257 (贺学勤, 刘庆昌, 王玉萍, 翟红, 2005,中国甘薯地方品种的遗传多样性分析, 中国农业科学, 38(2): 250-257)
Kriegner A., Cervantes J.C., Burg K., Mwanga R.O.M., and Zhang D.P., 2003, A genetic linkage map of sweetpotato [Ipomoea batatas (L.) Lam.] based on AFLP markers, Mol. Breed., 11: 169-185 doi:10.1023/A:1022870917230
Li G., Gao M., Yang B., and Quiros C.F., 2003, Gene for gene alignment between the brassica and a robidopsis genomes by direct transcriptome mapping, Theor. Appl. Genet., 107: 168-180 PMid:12835942
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103: 455-461 doi:10.1007/s001220100570
Li Q., Jie Q., Liu Q.C., Wan X., Ma D.F., Zhai H., and Wang Y.P., 2007, An efficient and rapid method for sweetpotato genomic DNA extraction, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 5(5): 743-746 (李强, 揭琴, 刘庆昌, 王欣, 马代夫, 翟红, 王玉萍, 2007, 甘薯基因组DNA高效快速提取方法, 分子植物育种, 5(5): 743-746)
Li Q., Liu Q.C., Zhai H., Ma D.F., Wang X., Li X.Q., and Wang Y.P., 2008, Genetic diversity in main parents of sweetpotato in China as revealed by ISSR marker, Zuowu Xuebao (Acta Agronomica Sinica), 34(6): 972-979 (李强, 刘庆昌, 翟红, 马代夫, 王欣, 李雪琴, 王玉萍, 2008, 中国甘薯主要亲本遗传多样性的ISSR分析, 作物学报, 34(6): 972-979)
Lin Q.L., 2005, Cloning and analysis of NBS-LRR type resistance gene analogs in sweetpotato (Ipomoea batatas), Thesis for M.S., South China University of Tropical Agriculture, Supervisor: Zeng H.C., pp.8-11 (林巧玲, 2005, 甘薯NBS-LRR类抗病基因同源序列的克隆与分析, 硕士学位论文, 华南热带农业大学, 导师: 曾会才, pp.8-11)
Liu Q.C., 2004, Importance of sweetpotato in the security of food and energy in China, Keji Daobao (Sci. Tech. Rev.), 9: 21-22 (刘庆昌, 2004, 甘薯在我国粮食和能源安全中的重要作用, 科技导报, 9: 21-22)
Nei M., and Li W., 1979, Mathematical model for studying genetic variation in terms of restriction endonucleases, Proceeding of the National Academy of Sciences of the USA, 76: 5256-5273 doi:10.1073/pnas.76.10.5269
Pan D., Yu M.W., Guo Y.C., Wei L., Ji R.W., Eviatar N., and You L.Z., 2010, Sequence-related amplified polymorphism (SRAP) of wild emmer wheat (Triticum dicoccoides) in Israel and its ecological association, Biochemical Systematics and Ecology, 38(1): 1-11
Wang Y., Rosen B., Scoffield J., Egnin M., Steiner S., Cook D.R., and He G.H., 2009, Isolation and analysis of resisitance gene homologues in sweetpotato, Plant Breeding, 129(5): 519-525
Wang Y., Wang R.F., and He G.H., 2008, Cloning of resistance gene analog (RGA) from sweetpotato (Ipomoea batatas (L.) Lam), Nanjing Nongye Daxue Xuebao (Journal of Nanjing Agricultural University), 31(3): 81-86(王钰, 王荣富, 何国浩, 2008, 甘薯抗病基因同源序列的克隆与分析, 南京农业大学学报, 31(3): 81-86)
Wu J., Tan W.F., Yan W.Z., Zhong C.S., and Wang D.Y., 2007, Genetic relationship of sweetpotato cultivars analysised by sequence-related amplified polymorphism, Journal of Sichuan University (Natural Science Edition), 44(2): 878-882 (吴洁, 谭文芳, 阎文昭, 钟昌松, 王大一, 2007, 甘薯种质资源亲缘关系SRAP标记分析, 四川大学学报, 44(2): 878-882)
Xiao P.F., Guo G.N., Li P.G., Man Z.B., 2008, Genetic diversity of dianthus accessions as assessed using two molecular marker systems (SRAPs and ISSRs) and morphological traits, Scientia Horticulturae, 117: 263-270 doi:10.1016/j.scienta.2008.04.001
Xiao T.X., 2006, Development and application of PCR-based RGA markers in rice, Thesis for M.S., Agricultural and biotechnology institute, Zhejiang University, Supervisor: Zhu J., Wu W.R., pp.15-22 (肖天霞, 2006, 基于PCR的水稻RGA标记开发和利用, 硕士学位论文, 浙江大学农业与生物技术学院, 导师: 朱军, 吴为人, pp.15-22)
Xu B.Q., Du Z.J., and Huang J.S., 2004, Advances in cloning candidate disease resistant genes with the RGA cloning method, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 2(3): 421-428 (徐兵强, 杜中军, 黄俊生, 2004, RGA法克隆候选抗病基因的研究进展, 分子植物育种, 2(3): 421-428)
Zhang D.P., Genoveva R., Albert K., and Robert H., 2004, AFLP assessment of diversity in sweetpotato from Latin America and the Pacific region: Its implications on the dispersal of the crop, Genetic Resources and Crop Evolution, 51: 115-120 doi:10.1023/B:GRES.0000020853.04508.a0
Zhang S.P., Wang L.J., Cao S.Y., and Liu T., 2008, Analysis of genetic diversity of 23 pomegranate genotypes by SRAP, Guoshu Xuebao (Journal of Fruit Science), 25(2): 655-660 (张四普, 汪良驹, 曹尚银, 刘涛, 2008, 23个石榴基因型遗传多样性的SRAP分析, 果树学报, 25(2): 655-660)
Zhou Z., Wang X., Ma D.F., Li H.M., Xie Y.P., and Li X.Y., 2005, Genetic diversity analysis based on RAPD for resistance and susceptibility of sweetpotato varieties to stem-namatode, Jiangsu Nongye Xuebao (Jiangsu J. of Agr. Sci.), 21(1): 35-39 (周忠, 王欣, 马代夫, 李洪民, 谢逸萍, 李秀英, 2005, 甘薯抗、感茎线虫病品种遗传多态性的RAPD分析, 江苏农业学报, 21(1): 35-39)