2.中国科学院遗传与发育生物学研究所, 北京, 100101
3.辽宁省果树科学研究所, 营口, 115009
4.北京市农林科学院林业果树研究所, 北京, 100093
5.中国科学院武汉植物园, 武汉, 430074
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 76 篇 doi: 10.5376/mpb.cn.2011.09.0076
收稿日期: 2011年03月12日 接受日期: 2011年05月16日 发表日期: 2011年06月16日
刘硕等, 2011, SRAP技术在桃(Prunus persica L.)遗传多样性研究上的应用, 分子植物育种 Vol.9 No.76 (doi: 10.5376/mpb.cn.2011.09.0076)
序列相关扩增多态性标记(SRAP)是一种基于PCR技术的新型分子标记,具有多态性高、重复性好、操作技术简单、成本低等优点,被广泛用于遗传图谱构建、比较基因组学、基因定位和遗传多样性分析等领域。本研究以48个桃品种为试材,采用SRAP技术对桃品种的遗传多样性进行分析,筛选出28对多态性丰富的引物组合,共产生864条扩增带,其中有379条多态性条带;每对引物平均产生31条扩增带,其中有14条多态性带,平均多态性比例为44.1%,平均PIC指数为0.28。对不同类群和来源的遗传距离分析发现,黄肉桃的平均遗传距离最大而硬肉普通桃最小,中国地方品种平均遗传距离最大而中国选育品种的遗传距离只有0.28,说明我国选育品种的遗传基础比较狭窄,需要在育种过程中广泛地使用各种类型的桃种质材料,丰富培育品种的遗传基础。经NT-sys聚类,将48个桃品种材料划分为9个层次,14个类群,平均相似系数为0.57,分析结果较好地反应了参试桃资源间的遗传、地理起源及果型、果肉颜色等生物学性状的遗传多样性,表明SRAP标记可以用于桃品种的鉴定、遗传多样性及遗传图谱构建的研究中。
桃(Prunus persica L.)属蔷薇科(Rosaceae)李属(Prunus L.)植物,为多年生乔木,是世界范围内重要的果树种类之一。我国作为桃的原产地,约在3000年前就有栽培(汪祖华和庄恩及, 2001),具有丰富的种质资源。19世纪中期以来,世界范围内桃种质材料交流频繁。欧美及日本以我国“上海水蜜”桃为基础育成众多的优良品种,在生产上得到了大面积的种植(汪祖华和庄恩及, 2001)。近年来,我国利用优质的地方品种和国外品种进行杂交也育成了许多品种,如“华玉”、“瑞光3号”、“瑞蟠20号”等。
序列相关扩增多态性标记(sequence related amplified polymorphism, SRAP)是由美国加州大学Li和Quiros (2001)发现的一种基于PCR技术的新型分子标记。其上游引物特异性地和外显子结合,下游引物特异性地和启动子区域结合,同时对多个开放阅读框(open reading frames, ORF)进行扩增,因个体不同以及物种的内含子、启动子与间隔长度不等而产生多态性扩增产物。SRAP标记是一种无需任何序列信息可直接PCR扩增的新型分子标记技术,与随机引物扩增多态性DNA (random amplified polymorphic DNA, RAPD)、简单重复序列(simple sequence repeat, SSR)、扩增片段长度多态性(amplified restriction fragment polymorphism, AFLP)等标记比较,具有多态性高、重复性好、操作技术简单、成本低等优点,近年来被广泛应用在苜蓿(沈程文等, 2009)、姜黄(Leng et al., 2009)、鸡冠花(Feng et al., 2009)、茄子(Mutlu et al., 2008)、甜瓜(陈芸等, 2009)和枣(李莉等, 2008)等植物上的遗传图谱构建、比较基因组学、基因定位、遗传多样性分析和标定基因等领域。
DNA分子标记是种质资源鉴定、评价的重要工具,程中平等(2001)利用RAPD技术对新疆桃分类地位的探讨;陈巍等(2007)利用SSR技术对中国、日本和美国桃育成品种进行遗传多样性研究,通过计算Nei’s和Shannon指数揭示欧美品种群遗传变异最高,其次为中国,最后为日本。Yoon等(2006)利用SSR技术对国内、日本、韩国和美国等地区96个桃品种进行遗传多样性研究,并通过PIC指数检测SSR标记利用效率。国内利用SRAP标记开展桃多样性评价的研究报道仅见史红丽等(2009),但其试材全部为国内品种,并且多为陕西、宁夏等西北地区品种。本实验取48个桃品种为试材,包括2个桃近缘种(新疆地区),31个国内地方品种(东北、华北、西北、东南和西南等地区),15个国外品种(美国、欧洲, 日本等地区)。应用SRAP分子标记技术,筛选可以用于桃遗传多样性分析的引物组合,揭示不同引物组合的扩增位点数、多态性位点数及多态信息含量(polymorphism information content, PIC),展现SRAP标记不同引物组合的利用效率及其和性状的相关性,以期为桃种质资源多样性的分析鉴定以及分子标记辅助育种奠定基础。
1结果与分析
1.1 SRAP引物多态性分析
SRAP扩增结果经检测发现,88个引物组合均能扩增出多态性条带,其中多态性较高的组合有28个,每个组合能产生16~53条清晰条带(图1; 表1)。28个引物组合共扩增出863条清晰条带,平均每对引物组合扩增出31个条带,引物组合Me1Em9扩增的条带最多,为53条,而Me7Em10只扩增出16条带(表1)。在所有的扩增条带中,有379条在48个品种中表现出多态性,每个组合的多态性条带数为4~25,平均每个引物组合产生13.5个多态性条带。在多态性比例方面,每个引物组合在12.5%~86.2%之间,平均多态性比例为44%,组合Me4Em10扩增的29条带中有25条带具有多态性,多态性比例高达86.2%,而组合Me7Em11尽管也扩增出32条带,但只有4条带具有多态性,其多态性比例只有12.5%。每个引物组合的多态信息含量也不尽一致,最小的组合只有0.07 (Me7Em11),最大的为0.50 (Me4Em7),大多数的组合在0.30以下,平均多态信息含量为0.28(表1)。进一步分析发现,上游引物中以Me4较好,由其组合的引物PIC值大多在0.30以上,下游引物中Em4组合具有较高的PIC值(表1)。在28个引物组合中,PIC最高的5个引物组合(Me4Em7, Me4Em10, Me4Em11, Me5Em4, Me7Em4)共能扩增出134条带,其中有98条带具有多态性,多态性比例高达74%,因此这些引物组合可以优先用于桃品种鉴定、遗传多样性及遗传作图的研究中。
![]()
图1 SRAP引物组合Me1Em6电泳图(编号同表1)
注: M: 100 bp (BM019)
Figure 1 Profile of Me1Em6 combination in 48 peach cultivars
Note: M: 100 bp (BM019)

表1 SRAP引物组合及多态性信息
Table 1 SRAP primer combinations and polymorphism information
1.2不同类群遗传距离分析
供试的48份种质按照来源可分为中国地方品种25份,国外品种(日本, 美国和欧洲)15份和中国选育品种8份(表1);按照品种特性分为黄肉桃15份,蟠桃8份,溶质普通桃12份,硬肉普通桃6份和油桃5份(程中平等, 2001)。对SRAP扩增结果按照不同品种特性类群进行分析发现,不同类群中多态性位点数最多的是黄肉桃,高达355个;最少为硬肉普通桃,仅210个;稀有位点数方面(只在一个品种中出现的条带),黄肉桃最多(27个),其次是溶质普通桃(26个),蟠桃及油桃均为1个,硬肉普通桃则无稀有位点。而在平均遗传距离方面,黄肉桃最大(0.48)而硬肉普通桃只有0.27(表2),表明在所供试的黄肉桃具有较高的遗传多样性,黄肉桃的遗传来源广或在长期的育种过程中遗传变异较大,硬肉普通桃遗传基础可能比较狭窄,来源比较单一,亲缘关系较近。对国内外(地方)品种比较发现,国外品种多态性位点数最多,为347个(其中稀有位点26个),中国地方品种为315个(其中稀有位点4个),而中国选育品种最少(237个)。中国地方品种平均遗传距离最大(0.40)而中国选育品种最小(0.28)(表2),说明我国地方品种由于分布广、来源不一,具有丰富的遗传多样性,而选育品种由于频繁地使用某些优良亲本,造成了遗传基础比较狭窄,因此需要在育种进程中广泛地使用各种类型的桃种质材料,尤其是地方品种,丰富我国桃品种培育的遗传基础,提高培育品种的遗传多样性。

表2 SRAP引物扩增的多态性位点数,稀有位点数及平均遗传距离
Table 2 Number of alleles and rare alleles assayed by 28 SRAPs, and index of genetic distance between different groups in peach (Prunus persica)
1.3桃品种聚类分析
48个品种SRAP标记分析的数据经NT-SYS2.10e软件分析,通过UPGMA聚类,得到SRAP标记的聚类图(图2)。不同品种之间的相似系数在0.27~0.89之间,平均相似系数为0.57。分析结果在相似系数0.27处,可将参试材料分Ⅰ、Ⅱ两大类群,其中Ⅰ类群只包含来自于保加利亚的保1一份材料;Ⅱ类群又分为A,B两个类群,其中A类群所含的材料(“张黄7号”, “临黄9号”和“肉蟠桃”)均来自甘肃,三者间的遗传背景较近;B类群除乌桃外分为C、D两类群,C类群包括“新疆大甜仁”和“新疆小甜仁”均来源于新疆,为桃近源种。D类群可以进一步分为E、F两类群:E类群除‘兴津油桃’外均为以云南品种为主的地方品种;F类群含两个类群,其中G类群含25品种,除4个日本品种及美国品种“Vivian”外,均为中国地方品种;H类群分为L、K两类群,其中具有国外血统的“华玉”、“瑞光3号”和“瑞蟠20号”三个国内育成品种和两个国外品种(“Dixon”和“Frederic”)以及中国地方品种“吊枝白”形成K类群,L类群中除了地方品种“早黄金”外均为国外品种,本结果表明“吊枝白”和“早黄金”和国外品种具有较近的亲缘关系,推测“吊枝白”和“早黄金”或其相似的地方品种(祖先种)可能作为了最初的桃育种的种质材料被国外利用。此外,聚类分析结果在一定程度上也反映了供试材料的某些生物学性状,如J类群中“撒花红蟠桃”、“白芒蟠桃”、“黄金蟠桃”和“晚熟大蟠桃”均为蟠桃,L类群中除“Robin”外均为黄肉桃,而K类群中除“Dixon”和“Frederic”外均为白肉桃。分类结果表明SRAP分子标记聚类分析的结果不但与资源的地域分布相一致,也与部分生物学性状相吻合,所以SRAP分子标记技术可以用于桃遗传多样性的分析。
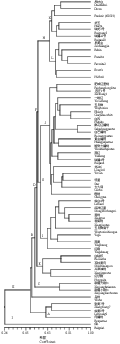
图2 桃品种遗传多样性聚类图
Figure 2 Dendrogram of 48 peach cultivars based on SRAP markers with UPGMA method
1.4与桃重要性状显著相关的标记位点
为了研究SRAP标记和桃品种性状之间的相关性,将桃重要性状包括风味、果肉颜色、果实形状等8种性状按照性状的有无进行数字化处理(如果实圆形记为1, 其它记为0),所得性状数据矩阵利用SPSS12.0与SRAP标记数据进行相关性的检测(t0.01,45=2.69, 相关系数r在0.37以上显著)。结果显示,有26对引物组合的109个标记位点和桃不同性状之间存在显著的相关关系(结果未列出),其中有12个引物组合的26个标记位点分别和多个性状(3个以上)有显著的相关关系(表3),如引物组合Me1Em11的不同位点分别和果实风味、果皮底色、果肉颜色、花型及树型之间存在着显著的相关性。每一个性状也存在着多个相关的标记,组合Me1Em11、Me6Em6、Me3Em4和Me4Em2均与果实风味(酸)显著相关;组合Me1Em11、Me6Em6和Me4Em2与白色果肉性状显著相关;组合Me4Em1、Me4Em3和Me9Em4与粘核性状之间存在相关(表3)。说明利用这些引物组合的分子标记有可能对有关性状进行有效地鉴定,特别是一些性状的早期选择,可以有效地提高育种效率,缩短育种进程。

表3 与桃重要性状相关的SRAP标记
注: 相关系数在0.37以上即为P=0.01水平上的相关显著
Table 3 Preferable SRAP primer combinations correlated with peach important traits
Note: the correlation coefficient over 0.37 was significantly related to the level at 0.01 (p)
2讨论
多态信息含量(PIC)直接反应出一个遗传标记所能提供的遗传信息总量。本研究把一个引物组合所有扩增出的条带作为一个整体进行引物组合多态信息含量(PIC)的计算,得到PIC值介于0.07~0.50之间,平均PIC值为0.28,属于中度水平(Botstein et al., 1980)。Yoon等(2006)利用SSR标记检测96个桃材料,其PIC值范围为0.4~0.98,平均PIC值为0.8,高于本实验结果0.28。其原因为本实验在计算PIC值时把无多态性的条带也一并统计在内,因此PIC值会低于其它单位点检测标记(如SSR)的结果。一般来说多态性的条带数越少(即多态性比例越低),PIC值越低,但我们也发现有些多态性高的引物组合,其PIC值并不高,如引物组合Me4Em10多态性条带最多,比例高达86.2%,其PIC值仅为0.46,说明这些多态性条带在整个群体中并不是均匀分布的,很多多态性条带只在少数品种中出现(如保1)。在28个引物组合中,PIC值最高的5个引物组合(Me4Em7, Me4Em10, Me4Em11, Me5Em4, Me7Em4)能扩增出98条多态性条带,揭示了品种间丰富的遗传多样性,因此这些引物组合可以优先用于桃品种的鉴定、遗传多样性及遗传作图的研究中。
遗传多样性是物种长期进化的结果,是种群生存和发展的基础。本研究利用SRAP技术对48个桃品种进行遗传多样性的研究,品种的相似系数在0.27~0.89之间,平均相似系数为0.57(图2),与史红丽等(2009)对47个桃材料聚类相似系数(0.501~0.842),陈巍等(2007)对56份桃材料聚类相似系数(0.2~0.79)结果相近。此外,本试验结果高于柑橘、柿子等其它木本果树植物的SRAP聚类结果,说明桃具有更高的遗传多样性(Guo and Luo, 2006; Uzun et al., 2009),这可能与材料中包含有大量的亲缘关系较远的地方品种有关。通过聚类分析,发现日本的溶质普通桃和中国的蟠桃及部分溶质普通桃聚在一起(J类群),说明蟠桃品种虽然在果型上和其它类型的桃之间有显著的差别,但其它性状的遗传基础和普通桃类似;日本桃品种培育最早是以中国上海的优异水蜜桃为基础的,和中国的溶质普通桃应该具有较高的遗传相似性。我国的溶质普通桃分布在不同的类群中(J和I类群),说明我国的溶质普通桃遗传背景比较复杂,起源可能是多元的(陈巍等, 2007)。“兴津油桃”和来源于日本的其它品种(普通桃)并没有聚在一起,说明日本油桃品种的选育和其它桃的遗传基础不尽一致。其它小类群(如A, C和I)和以上这4大类群因相似系数较低,但所含的品种来源一致,如来自新疆的“新疆大甜仁”和“新疆小甜仁”,这两个品种是桃的近缘种,和桃具有比较远的亲缘关系(程中平等, 2001)。甘肃具有独特的生态环境,“张黄7号”、“张黄9号”和“肉蟠桃”经过长期的驯化形成了特殊的生态类型,故聚在一起。对所有品种聚类结果分析发现,来自保加利亚的品种“保1”和其它桃品种的相似系数只有0.27,而其生物学性状与其它品种之间并无显著差异,国内外的研究报道并中无该品种的研究介绍,该材料也是首次在本实验中被利用,在SRAP电泳图谱上也发现该品种存在大量稀有位点,说明该品种存在着特殊的遗传基础,有可能作为重要的种质材料进行桃品种的选育,丰富选育品种的遗传多样性。
本实验不足之处是仅凭借SRAP一种分子标记通常很难真实反映资源间的相互关系,只有通过多种分子标记综合分析,并结合遗传起源、地理分布、植物学和生物学性状调查分析,才能更准确地揭示品种资源间的遗传差异,丰富与桃生物学性状紧密相关的标记数量。本实验室也在将多种分子标记方法同时应用于桃品种资源的扫描,目的在于借助不同的分子标记方法,弥补单一标记的不足,更全面更有效的揭示桃遗传多样性。
总之,本研究证明SRAP标记含有较高的多态性信息,可以较好的对桃品种材料进行遗传多样性分析,其聚类结果能够反映资源间的遗传起源、地理分别、植物学和生物学性状等方面的相互关系。从统计学角度对桃生物学性状进行相关分析,找到一些与桃果实颜色、形状等重要性状相关度高的SRAP引物组合,为SRAP标记的应用及丰富桃品种遗传多样性分析提供较好的实验结果。
3材料与方法
3.1试验材料
试验材料于2009年夏季取自北京市农林科学院林业果树研究所桃国家资源圃,共48份(表4),其中中国地方品种25份,国外品种(日本、美国和欧洲)15份,中国选育品种8份。取当年生枝条的幼嫩叶片0.2 g,放入2.0 mL 离心管中,液氮速冻,置于-80℃ 低温冰箱保存。

表4 试验材料及类群划分
Table 4 Peach cultivars, their origins and groups used in this study
3.2 DNA提取及SRAP分析
3.2.1 DNA提取
实验材料经液氮浸泡后使用Geno/Grinder 2000研磨机磨碎。基因组总DNA的提取采用改良CTAB法(Doyle and Doyle, 1990),提取后稀释至20 ng/μL备用。
3.2.2引物设计及组合
SRAP引物参考Li和Quiros (2001)的引物加以补充,正向引物(forward primer) 8种,反向引物(reverse primer) 11种,形成88个组合(表5)。引物由北京赛百盛生物工程公司合成。
![]()
表5 SRAP正反向引物序列
Table 5 The forward and reverse SRAP primers in this study
3.2.3 SRAP-PCR反应体系及程序设定
扩增反应在Bio-RAD C1000型PCR仪上进行,反应程序在Li和Quiros (2001)基础上优化为95℃ 预变性5 min;94℃ 60 s,34℃ 60 s,72℃ 60 s,循环5次;95℃ 60 s,50℃ 60 s,72℃ 60 s,循环35次;72℃延伸10 min。PCR反应总体积为20 μL,包括100 ng DNA模板、1.5 mmol MgCl2、0.2 mmol dNTPs、0.2 umol引物、1.0 U Taq DNA聚合酶和1X Buffer。以上试剂全部来自北京天根生化科技有限公司。
3.2.4 PCR产物检测
PCR扩增产物经95℃变性处理10 min,冰上冷却后用4.5%变性聚丙烯酰胺凝胶电泳分离,恒功率75W电泳50 min (Bio-rad 38×30 cm电泳系统),电泳后利用银染法进行条带的展示(韩永亮和常金华, 2006)。电泳胶板晾干后进行条带的统计,并拍照记录图像。
3.2.5数据分析计算
SRAP图谱仅统计条带清晰、片段大小为80~600 bp的条带,有带记为1,无带记为0,形成数字化矩阵数据。用NT-sys2.10e软件,采用Jacard相似系数计算群体间遗传距离(genetic distance, GD),利用非加权分组平均法(unweighted pair group method with arithmetic mean, UPGMA)进行聚类分析。相似系数计算公式为Sij=a/(a+b+c),其中a为两份样品共有带数,b为i样品的特有条带数,c为j样品的特有条带数,遗传距离GD=1-Sij。利用SPSS12.0软件分析SRAP标记与桃重要性状之间的相关性。计算各个引物组合的多态性比例(多态性比例%=多态性条带数/扩增总条带数×100%)和多态信息含量(polymorphism information content, PIC) (Botstein et al., 1980)。
作者贡献
刘硕、刘冬成、刘威生、章秋平和刘有春是本研究的实验设计和实验研究的执行人;刘硕、刘冬成和章秋平完成数据分析,论文初稿的写作;赵剑波、李绍华和张爱民参与实验设计,试验结果分析及论文修改;李宝江是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由公益性行业(农业)科研专项(3-37)资助。本文中提到了我们实验中涉及的有关试剂供应商和测序服务商,这并非我们为这些试剂供应商和测序服务商的产品和服务提供推荐或背书。
参考文献
Botstein D., White R.L., Skolnick M., and Davis R.W., 1980, Construction of a genetic linkage map in man using restriction fragment length polymorphisms, Am. J. Hum. Genet, 32(3): 314-331
PMid:6247908 PMCid:1686077
Chen W., Wang L.R., Zhang S.L., Chen C.W., and Cao K., 2007, Genetic diversity analysis of peach (Prunus persica) cultivars introduced from different countries by SSR, Guoshu Xuebao (Journal of Fruit Science), 24(5): 580-584(陈巍, 王力荣, 张绍铃, 陈昌文, 曹珂, 2007, 利用SSR研究不同国家桃育成品种的遗传多样性, 果树学报, 24(5): 580-584)
Chen Y., Wang X.L., Xia X.Q., Chang J.Q., and Li G., 2009, Establishment and optimization for SRAP-PCR system of Muskmelon, Xinjiang Nongye Kexue(Xinjiang Agricultural Sciences), 46(4): 764-771(陈芸, 王贤磊, 夏雪琴, 常菊芹, 李冠, 2009, 甜瓜SRAP-PCR程序和反应体系的优化, 新疆农业科学, 46(4): 764-771)
Cheng Z.P., Chen Z.W., Hu C.G., Deng X.X., and Luo Z.R., 2001, Classification study of xinjiang peach using RAPD markers, Yuanyi Xuebao (Acta Horticulturae Sinica), 28(3): 211-217(程中平, 陈志伟, 胡春根, 邓秀新, 罗正荣, 2001, 利用RAPD技术对新疆桃分类地位的探讨, 园艺学报, 28(3): 211-217)
Doyle J.J., and Doyle J.L., 1990, Isolation of plant DNA from fresh tissue, Focus, 12: 13-15
Feng N., Xue Q., Guo Q., Zhao R., and Guo M., 2009, Genetic diversity and population structure of Celosia argentea and related species revealed by SRAP, Biochem Genet, 47(7-8): 521-532
http://dx.doi.org/10.1007/s10528-009-9257-z PMid:19521763
Guo D.L., and Luo Z.R., 2006, Genetic relationships of some PCNA persimmons (Diospyros kaki Thunb.) from China and Japan revealed by SRAP analysis, Genetic Resources and Crop Evolution, 53(8): 1597-1603
http://dx.doi.org/10.1007/s10722-005-8717-5
Han Y.L., and Chang J.H., 2006, Comparative analysis of two kinds of methods to detect SSRs on polyacrylamide gel electrophoresis systems, Zaliang Zuowu (Rain Fed. Crops), 26(3): 176-177(韩永亮, 常金华, 2006, 聚丙烯酰胺凝胶电泳的两种染色方法对SSR标记的影响, 杂粮作物, 26(3): 176-177)
Leng C.H., Tao Z.M., Wu Z.G., Lin X.C., Lou Y.F., and Jiang C.X., 2009, Study on genetic diversity of curcuma wenyujin from various habitats by SRAP, Zhong Yao Cai, 32(10): 1507-1510
PMid:20112709
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: it′s application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103(2-3): 455-461 http://dx.doi.org/10.1007/s001220100570
Li L., Peng J.Y., and Zheng B.Q., 2008, Optimization of SRAP-PCR system with orthogonal design in zixiphus jujube, Nongye Shengwu Jishu Xuebao (Journal of Agricultural Biotechnology), 16(2): 361-362(李莉, 彭建营, 郑宝强, 2008, 枣SRAP-PCR体系的正交优化, 农业生物技术学报, 16(2): 361-362)
Mutlu N., Boyaci F.H., Göçmen M., and Abak K., 2008, Development of SRAP, SRAP-RGA, RAPD and SCAR markers linked with a fusarium wilt resistance gene in eggplant, Theor Appl Genet, 117(8): 1303-1312
http://dx.doi.org/10.1007/s00122-008-0864-6 PMid:18712340
Shen C.W., Ning Z.X., Huang J.A., Chen D., and Li J.X., 2009, Genetic diversity of camellia sinensis germplasm in guangdong province based on morphological parameters and SRAP markers, Yingyong Shengtai Xuebao (Chinese Journal of Applied Ecology), 20(7): 1551-1558(沈程文, 宁正祥, 黄建安, 陈栋, 李家贤, 2009, 基于表型参数及SRAP标记的广东茶树种质遗传多样性, 应用生态学报, 20(7): 1551-1558)
Shi H.L., Han M.Y., and Zhao C.P., 2009, Genetic diversity analysis of prunus persica using SRAP and SSR markers, Huabei Nongxuebao (Acta Abricuturae Boreali Sinica), 24(6): 187-192(史红丽, 韩明玉, 赵彩平, 2009, 桃遗传多样性的SRAP和SSR标记分析, 华北农学报, 24(6): 187-192)
Uzun A., Yesiloglu T., Aka-K.Y., and Gulsen O., 2009, Genetic diversity and relationships within citrus and related genera based on sequence related amplified polymorphism markers (SRAPs), Sci. Hort, 121(3): 306-312
http://dx.doi.org/10.1016/j.scienta.2009.02.018
Wang Z.H., and Zhuang E.J., 2001, Chinese monograph of fruit trees, Beijing : China Forestry Press, pp.41-94(汪祖华, 庄恩及, 2001, 中国果树志-桃卷, 北京: 中国林业出版社, pp.41-94)
Yoon J.H., Liu D.C., Song W.S., Liu W.S., Zhang A.M., and Li S.H., 2006, Genetic diversity and ecogeographical phylogenetic relationships among peach and nectarine cultivars based on simple sequence repeat (SSR) markers, J. Am. Soc. Hortic. Sci., 131(4): 513-521
Yoon J.H., Yang D.C., Liu D.C., Song W.S., Zhang A.M., and Li S.H., 2009, Phylogenetic relationships among cultivars of Prunus persica based on internal transcribed spacer (ITS) sequences of nuclear ribosomal DNA, The journal of horticultural science & biotechnology, 84(2): 167-174