1.中国海洋大学, 海洋生物遗传育种研究室, 青岛, 266003
2.全国水产技术推广总站苗种处, 北京, 100125

作者

通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 95 篇 doi: 10.5376/mpb.cn.2011.09.0095
收稿日期: 2011年06月28日 接受日期: 2011年07月21日 发表日期: 2011年07月27日
推荐引用:
引用格式(中文):
孙晓宇等, 2011, 不同保存条件下五种大型海藻的DNA提取和PCR分析, 分子植物育种(online) Vol.9 No.95 pp.1680-1691 (doi: 10.5376/mpb.cn.2011.09.0095)
引用格式(英文):
Sun et al., 2011, DNA extraction and PCR analysis of five kinds of large seaweed under different preservation conditions, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.9 No.95 pp.1680-1691 (doi: 10.5376/mpb.cn.2011.09.0095)
高质量DNA的提取纯化是进行核酸分子生物学研究的基础与前提。大型海藻中不仅含有丰富的多糖、酚类、萜类等次生物质,而且这些物质在不同藻类、不同组织部位以及不同生长发育阶段的含量和比例不同,这些物质易导致DNA难以溶出或产生不同程度的氧化,严重影响DNA的纯度和得率。与陆生高等植物所含有的中性多糖相比,大型海藻中含有的硫酸基多糖更易水溶,使溶液高度粘稠,在DNA提取中很难去除,同时,这种酸性多糖会抑制限制性酶切反应和PCR扩增 (Hong et al., 1995)。海带、马尾藻等藻类的DNA提取难度甚至超过了烟草、龙眼、梨等顽拗植物。尽管近年来,藻类学工作者已分别在紫菜、龙须菜等红藻,浒苔、石莼等绿藻以及海带、裙带菜等褐藻中已建立了一系列的DNA提取方法,但仍主要集中在鲜活样品的提取(王萍等, 2007; 隋正红等, 2000; 杨君等, 2000; 胡远皆和周志刚, 2001; 张东等, 2009)。
随着分子标记及基因组学研究在海藻遗传学和育种学研究中不断深入发展,DNA的大批量、高效提取是进行高通量研究的必备手段。同时,由于大型海藻分布环境特殊性,样品储运条件复杂、运输时间长也对材料储存工作提出了更高的要求。如何结合样品储存与研究需要,建立大型海藻样品保存以及适用的DNA提取方法,是海藻分子遗传学研究面临的主要问题。本文选择了红藻、褐藻、绿藻门5种主要常见的大型海藻为研究对象,分别进行了活体培养、干燥、冷冻保鲜、低温处理等10种不同的样品储存条件处理,以期摸索出一种经济、有效的样品保存与DNA提取方法,应用于大型海藻分子遗传学分析等领域。
1结果与分析
1.1不同保存方法对DNA提取的影响
5 种大型海藻在10种保存条件下分别保存2个月,统一采用CTAB法提取DNA,用琼脂糖凝胶电泳检测提取情况,部分DNA电泳结果(图1)。不同干燥方法保存样品提取DNA结果显示除高温干燥后保存的样品外,其它样品均可提取到DNA;仅浒苔活体样品、硅胶干燥样品及龙须菜活体样品观察到点样孔有残留,可能是糖胶类物质残留造成;亨氏马尾藻恒温干燥样品及压标本法保存样品观察到DNA带拖尾,可能有少量降解;不同干燥方法保存的海带及亨氏马尾藻样品均观察到RNA残留,恒温保存及压标本法保存的龙须菜样品也观察到有少量RNA残留。不同低温保存样品提取DNA结果显示5种海藻均能提取到DNA;亨氏马尾藻和浒苔4℃保存样品观察到点样孔有残留,龙须菜-20℃和-80℃保存样品也观察到点样孔有残留;仅海带-20℃和-80℃保存样品有DNA带拖尾现象;龙须菜、浒苔和亨氏马尾藻4℃保存样品及所有海带冻存样品中均观察到RNA残留(表1; 表2)。

图1 不同保存方法提取的DNA电泳图
Figure 1 Electrophoresis figure of genomic DNA extracting from different methODs preserving sample |

表1 不同干燥方法样品提取DNA结果
Table 1 The result of DNA extracting from different methods desiccating sample |

表2 不同冻存方法提取DNA结果
Table 2 The result of DNA extracting from sample preservation at differrent low temperature |
1.2不同保存方法对DNA质量的影响
1.2.1不同干燥方法对DNA质量的影响
比较不同干燥方法保存样品所提取的DNA的浓度、纯度及得率结果(表3)。不同的大型海藻分别适合于不同的干燥保存条件。海带、龙须菜样品用恒温干燥法保存提取的DNA得率最高,其中海带的浓度为171.8 ng/μL,OD260/OD280为1.70,得率为34.36 μg/g,龙须菜的浓度为220.5 ng/μL,OD260/OD280为1.72,得率为44.10 μg/g;亨氏马尾藻样品用压标本法干燥保存提取的DNA得率最高,浓度为246.3 ng/μL,OD260/OD280为1.67,得率为49.26 μg/g;长心卡帕藻样品用冷冻干燥法保存提取的DNA得率最高,浓度为307.7 ng/μL,OD260/OD280为1.69,得率为61.54 μg/g;而浒苔样品用硅胶干燥法保存提取的DNA得率最高,浓度为245.8 ng/μL,OD260/OD280为1.93,得率为49.16 μg/g。

表3 不同干燥方法保存样品提取DNA的浓度、纯度及得率
Table 3 The concentration, purity and quantity of DNA extracting from different methods desiccating sample |
1.2.2不同冻存方法对DNA质量的影响
比较不同冻存方法提取的DNA溶液的浓度、纯度和得率(表4)。海带样品在-80℃冻存提取的DNA得率最高,浓度为250.7 ng/μL,OD260/OD280为1.80,得率为50.14 μg/g;亨氏马尾藻样品4℃、-20℃、-80℃保存提取的总DNA得率、纯度均较高,其中-80℃保存样品提取的总DNA得率最高,浓度为446.6 ng/μL,OD260/OD280为1.71,得率为88.32 μg/g;龙须菜样品4℃、-20℃保存提取的总DNA得率、纯度较高,其中以-20℃保存提取的DNA得率最高,浓度为510.9 ng/μL,OD260/OD280为1.81,得率为102.18 μg/g;长心卡帕藻样品在-20℃、-80℃保存提取的总DNA得率、纯度高,其中以-20℃保存提取的DNA得率最高,浓度为491.3 ng/μL,OD260/OD280为1.84,得率为98.26 μg/g;浒苔样品在4℃保存提取的DNA得率最高,浓度为554.2 ng/μL,OD260/OD280为1.96,得率为100.85 μg/g。综上表明海带、亨氏马尾藻、龙须菜、长心卡帕藻、浒苔在4℃、-20℃、-80℃保存后,均可获得完整的DNA并且DNA纯度、得率较高,但在4℃条件下长期保存的样品易出现部分组织腐烂的情况,因此低温储存样品选择在-20℃或-80℃较好。

表4 不同冻存方法保存样品提取DNA的浓度、纯度及得率
Table 4 The concentration, purity and quantity of DNA extracting from sample preservation at differrent low temperature |
1.3不同保存方法提取DNA对SRAP遗传标记扩增结果的影响
利用2对SRAP(ME1/EM3; ME3/EM2)引物对不同保存条件下所提取的DNA进行扩增(图2; 图3)。M1/E3引物扩增结果表明不同条件下保存的样品,能提取得到DNA的经过扩增均有与对照组相同的产物带型(高温干燥条件下保存的样品未检测得到DNA, 因此无扩增条带);E2/M3引物扩增结果表明,除龙须菜、长心卡帕藻、浒苔在高温干燥(45℃, 75℃)条件下保存的样品以外,均获得与对照组相同的扩增产物电泳图谱,同时也说明高温干燥(45℃, 75℃)条件下虽未能在琼脂糖凝胶上检测到DNA,溶液中仍存在浓度较低的DNA,但由于其在扩增中的不稳定性,此种样品制备方法不予以采用。而其他保存方法下样品提取的DNA溶液得率相对较高,在SRAP中的扩增具有稳定性,可用于分子标记的研究。

图2 利用SRAP引物ME1/EM3对不同保存方法DNA的扩增结果
Figure 2 SRAP results with different storing sample methods (primer: ME1/EM3) |
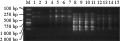
图3 利用SRAP引物ME3/EM2对不同保存方法DNA的扩增结果
Figure 3 SRAP results with different storing sample methods (primer: ME3/EM2) |
1.4不同保存方法提取DNA对rbcL、coxⅠ基因扩增结果的影响
利用rbcL 引物对不同保存条件下所提取的DNA进行扩增结果表明,以新鲜材料提取的DNA扩增产物的带型为对照,海带除在高温(45℃)干燥条件下保存的样品未有扩增之外,其他样品扩增产物的带型均与新鲜材料相同,此结果也说明高温(75℃)干燥所提取的DNA溶液中含有少量DNA;亨氏马尾藻、龙须菜、长心卡帕藻在各种条件下保存的样品扩增产物带型均与新鲜材料相同;浒苔除高温(75℃)干燥样品没有获得扩增产物,其他几种样品制备方法均获得与新鲜材料提取的 DNA扩增产物相同的带型(图4)。
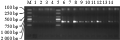
图4 不同保存样品的DNA的rbcL扩增结果
Figure 4 rbcL results with different storing sample methods |
利用coxⅠ引物对不同保存条件下所提取的DNA进行扩增结果表明,所有条件下保存的样品均有与对照样品相同的扩增(图5)。
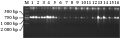
图5 不同保存样品的DNA的coxⅠ扩增结果
Figure 5 coxⅠ results with different storing sample methods |
综上两种基因扩增的结果显示,样品在高温干燥保存条件下扩增结果不稳定,其他保存条件下样品所提DNA,扩增结果稳定,可用于基因的扩增。
2讨论
2.1各种样品保存方法的评价
快速、经济地从大型海藻样品中提取高产量、高纯度的基因组DNA已成为藻类分子生物学研究的首要问题,其中藻类组织材料的采集与保存对提取DNA的产量和质量有很大影响。本研究探讨了5种大型海藻分别在6种保存方法处理后的DNA提取效率,除高温干燥样品外均得到了较完整的DNA。藻类样品含水量非常高,较长时间的高温干燥过程极易使样品的组织结构及化学物质发生变化,对样品的损害较大,因此影响了DNA提取效果,不适合作为藻类样品的干燥保存手段。冷冻干燥法和恒温干燥法需要有相应的干燥设备对新鲜样品及时进行干燥处理,不适合海藻材料采集现场保存;低温储存法可以作为样品短期(4℃)和长期(-20℃, -80℃)保存手段。相对而言,压标本干燥法和硅胶干燥法非常适用于采集地点多、相距远、野外采集时间长、所采样品的种类和数目多的遗传多样性研究,另外压标本干燥法比硅胶干燥法更简单、方便,而且可以更好地保存样品的形态特征,有助于样品的后期分类鉴定。
2.2不同保存方法样品提取DNA的PCR扩增检测评价
样品DNA的质量会对分子遗传分析结果的可靠性和稳定性产生影响,本研究提取的为大型海藻总DNA,可用于核基因组和细胞质基因组的研究。除高温干燥保存样品的DNA扩增结果不稳定外,其它不同条件下保存样品的DNA在经过扩增(2对RSAP引物, 1对rbcL引物和1对coxⅠ引物)均得到了与对照组(新鲜样品)相同的产物带型。
综上几种PCR扩增的结果显示,样品在高温干燥保存条件下扩增结果不稳定,其它保存条件下样品所提DNA,扩增结果稳定并且PCR扩增检测结果与对照组DNA无明显差别,可用于核基因组、线粒体及叶绿体基因组的分子遗传学研究。
2.3 DNA提取方法评价
从顽拗植物中提取高质量DNA的难度较大,科研工作者对各种顽拗植物的DNA提取方法进行了大量的探索工作。目前报道的藻类DNA提取方法有改良的CTAB 法(沈晓岚等, 2009),试剂盒法(韩静等, 2008),氯化锂法(Hong et al., 1995),SDS法,NaHSO3法, NaOH法(沈晓岚等, 2009)等多种方法。改良后的CTAB法即能裂解细胞又能有效地沉淀多糖,对于多糖类物质含量高的材料较使用。沈晓岚等(沈晓岚等, 2009)对不同属的观赏凤梨叶片用多种方法提取DNA并进行了比较,结果显示CTAB法所得DNA得率及质量均为最优。韩静等(韩静等, 2008)比较了改良的CTAB法、LiCI法及试剂盒法在三种海藻材料中抽提基因组DNA的效率,改良的CTAB法相对于试剂盒法得率要高,相对于氯化锂法等质量要好。
大型海藻中普遍富含蛋白质,但不同种属藻类含有的胶类物质不同,绿藻中果胶含量高,红藻中富含琼胶或卡拉胶,褐藻中褐藻胶含量丰富,且各类海藻均含有较多的酚类物质。本实验中涉及的5种大型海藻采用统一的改良CTAB法均取得了良好的DNA提取效果,5种海藻的活体样品平均得率为56.7 μg/g,制作成标本的样品在标本制作过程中可能受到部分影响,DNA提取难度相对较大,标本样品平均得率为39.4 μg/g,但相对于SDS法、LiCI法、试剂盒法等在多种藻类和高等植物的得率仍处于较高水平,充分显示了改良CTAB法在不同物种中较高的适用性,同时CTAB法操作相对简便,成本较低,是大型海藻样品理想的DNA提取方法。
3材料与方法
3.1实验材料
实验所用活体大型海藻由中国海洋大学大型海藻种质库提供(表5)。

表5 大型海藻样品信息
Table 5 The information of seaweed sample |
3.2主要试剂
CTAB提取缓冲液:100 mmol/L Tris-HCl (pH8.0),20 mmol/L EDTA,1.4 mol/L NaCl,2% CTAB,3% PVP,2% (v/v);β-巯基乙醇;氯仿:异戊醇(24:1);酚:氯仿:异戊醇(25:24:1)。
PCR中所用引物由博尚生物工程有限公司合成,MgCl2、PCR Buffer、dNTP、Taq DNA聚合酶购自TAKARA宝生物工程(大连)有限公司。其他药品购自上海国药集团,均为分析纯。
3.3样品制备
(1)对照组:新鲜的活体样品。
(2)实验组:藻体用消毒海水冲洗干净后,用吸水纸吸干表面水分,每组样品称取2份,每份1 g,处理条件如下:
a.硅胶干燥:将藻体放入10 g硅胶中,用密封塑料袋装好。在常温下进行干燥。
b.恒温干燥:将藻体放于培养皿中,放于电子防潮柜内进行干燥。
c.压标本法干燥:将藻体放于多层吸水纸间,用铁夹固定,在常温下放置。每隔24 h更换一次吸水纸。
d.高温干燥:将藻体放于培养皿中,在烘箱内分别进行45℃,75℃烘干处理至脱水。
e.冷冻干燥:将样品用低温冷冻干燥仪进行干燥,至脱水。
f.低温储存:将样品密封在样品袋中,分别在4℃、-20℃、-80℃冰箱内进行保鲜储存。
恒温干燥、高温干燥、冷冻干燥法处理样品至干燥后,用保鲜膜密封,放置于-20℃冰箱保存。样品保存时间均为2个月。
3.4 DNA提取方法
参照Lichtenstein和Draper (1985)的CTAB法稍作调整。利用0.2% (v/v) β-巯基乙醇抑制样品中酚类物质的氧化,并利用两个浓度的CTAB (2%和10%)与多糖等杂质结合。
3.5 DNA的质量检测
提取的DNA经1%琼脂糖凝胶电泳并用JS-380A型凝胶成像系统检测所得DNA的完整性,用Thermo Scientific NANODROP 1000分光光度计测定DNA浓度及OD260/OD280比值,并计算得率。
3.6 PCR分析
以提取的DNA为模板,分别利用rbcL、coxⅠ引物进行核外基因的扩增,同时利用2对SRAP引物进行扩增。
rbcL PCR扩增引物、反应程序和反应体系参考Lane等(Lane et al., 2006),所用引物为KL2 (5'-GATGCTGATTATAACGTTAAAG-3');KL8 (5'-GTTGGTGCATTTGACCACA-3')。
coxⅠ PCR扩增引物、反应程序及反应那个体系参照Folmer 等(Folmer et al., 1994),所用引物为LCO1490 (5'-TAAACTTCAGGGTGACCAAAAAATCA-3');HCO2198 (5'-GGTCAACAAATCATAAAGATATTGG-3')。
参考Qiao等(Qiao et al., 2000)的两对引物组合进行SRAP PCR扩增,其引物分别为ME1 (5'-TGAGTCCAAACCGGATA-3'),EM3 (5'-GACTGCGTACGAATTGAC-3');ME3 (5'-TGAGTCCAAACCGGAAT-3'),EM2 (5'-GACTGCGTACGAATTTGC-3')。两对引物组合反应体系相同,为20 μL,内含模板DNA 1 μL、引物2 μL、Buffer (10×) 2 μL、dNTP 1 μL、Taq DNA聚合酶0.5 μL。扩增程序相同为:95℃预处理5 min后,94℃变性1 min,35℃复性1 min,72℃延伸2 min的条件下5个循环后进行94℃变性1 min,48℃复性1 min,72℃延伸2 min的条件下进行35个循环,最后72℃延伸10 min。
扩增的PCR产物用1%的琼脂糖凝胶电泳(90V, 30min),用JS-380A型凝胶成像仪观察电泳结果并拍照。
作者贡献
孙晓宇、罗丹、赵翠、李巍和刘涛是本研究的实验设计和实验研究的执行人;孙晓宇、罗丹和赵翠完成实验操作和分析,论文初稿的写作;李巍参与了论文内容后期修改补充;刘涛是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家公益性行业专项(200903030, 200805075),山东省科技攻关计划(2006GG3205001),山东省科技兴海项目(2007GG10005018),转基因生物新品种培育科技重大专项(2009ZX08009-100B)资助,感谢罗丹,赵翠在实验数据整理中提供的帮助,感谢李巍在论文写作及修订中提供的帮助,感谢刘涛对本研究提供的指导。
参考文献
Folmer O., Black M., Hoeh W., Lutz R., and Vrijenhoek R., 1994, DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates, Molecular Marine Biology and Biotechnology, 3(5): 294-299
Han J., Yu S.W., and Luo L.J., 2008, Studies on efficient methods of algae DNA isolation, Journal of Shanghai Fisheries University, 17(5): 530-534 (韩静, 余舜武, 罗利军, 2008, 高效海藻DNA提取方法研究, 上海水产大学学报, 17(5): 530-534)
Hong Y.K., Kim S.D., Polne-Fuller M., and Gibor A., 1995, DNA extraction conditions from Porphyra perforata using LiCl, Journal of Applied Phycology, 7: 101-107 http://dx.doi.org/10.1007/BF00693055
Hu Y.J., and Zhou Z.G., 2001, Optimization of random amplified reaction conditions for Laminaria japonica (Phaeophyta) sporophyte DNA, Journal of Shanghai Fisheries University, 10(3): 193-198 (胡远皆, 周志刚, 2001, 海带孢子体DNA随机扩增反应条件优化, 上海水产大学学报, 10(3): 193-198)
Lane C.E., Mayes C., Druehl L.D.D., and Sauders G.W., 2006, A multi-gene molecular investigation of the kelp (Laminariales, Phaeophyceae) supports substantial taxonomic re-organization, Journal of Phycology, 42(2): 493-512 http://dx.doi.org/10.1111/j.1529-8817.2006.00204.x
Lichtenstein C.P., and Draper J., 1985, Genetic engineering of plants, In: DNA Cloning: A Practical Approach, Vol.Ⅱ, ed. by Glover, D.M., IRL Press, Oxford, UK, pp.67-119
Qiao L.X., Liu H.Y., Guo B.T., Weng M.L., Dai J.X., Duan D.L., and Wang B., 2007, Molecular identification of 16 Porphyra lines using sequence-related amplified polymorphism markers, Aquatic Botany, 87: 203-208 http://dx.doi.org/10.1016/j.aquabot.2007.06.006
Shen X.L., Ge Y.Y., Yu X.Y., Wang W.Y., and Mao B.Z., 2009, Explore about the method for extracting DNA of ornamental Bromelia, Northern Gardening, 12: 114-117 (沈晓岚, 葛亚英, 俞信英, 王炜勇, 毛碧增, 2009, 观赏凤梨DNA提取方法研究, 北方园艺, 12: 114-117)
Sui Z.H., Zhang X.C., and Kong J., 2000, Cloning and detecting of subvunit gene of phycoerythrin of Gracilaria kemaneiformis, Marine Sciences, 24(2): 9-11 (隋正红, 张学成, 孔杰, 2000, 龙须菜藻红蛋白亚基基因的克隆及监测, 海洋科学, 24(2): 9-11)
Wang P., Ni W.Z., and Meng X.P., 2007, Study on methods of DNA extraction from thallus of Porphyra yezoensis, Journal of Jilin Agricultural University, 29(6): 640-642, 646 (王萍, 倪伟镯, 孟学平, 2007, 条斑紫菜叶状体DNA提取方法的研究, 吉林农业大学学报, 29(6): 640-642, 646)
Yang J., An L.J., Wang Q., Wang H.W., Su Q., and Kang X.H., 2000, Application of RAPD in Ulva and Enteromorpha (Chlorophyta), Oceanologia Etlimnologia Sinica, 31(4): 408-413 (杨君, 安利佳, 王茜, 王宏伟, 苏乔, 康晓慧. 2000, 石莼属(Ulva)和浒苔属(Enteromorpha)绿藻的RAPD分析, 海洋与湖沼, 31(4): 408-413)
Zhang D., Sui Z.H., Yi H., Tong Z., and Bao Z.M., 2009, The Genetic diversity analysis of the sporophyte s of Undaria pinnatifida (Phaeophyceae) for Qingdao population by AFLP markers, Periodical of Ocean University of China, 39(4): 664-668 (张东, 隋正红, 易恒, 仝舟, 包振民, 2009, 青岛裙带菜孢子体野生种群遗传多样性的AFLP分析, 中国海洋大学学报, 39(4): 664-668)
 作者
作者  通讯作者
通讯作者
![]()




![]()
![]()
![]()
![]()
![]()