2云南农业大学农学与生物技术学院, 昆明, 650201
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 8 篇 doi: 10.5376/mpb.cn.2012.10.0008
收稿日期: 2012年02月17日 接受日期: 2012年02月29日 发表日期: 2012年03月19日
引用格式(中文):
田璐等, 2012, 苍山奇蝶捧瓣蝶化MADS基因筛选, 分子植物育种(online) Vol.10 No.8 pp.1061-1066 (doi: 10.5376/mpb. cn.2012.10.0008)
引用格式(英文):
Tian et al., 2012, Screening of MADS genes for llip-like petals in Cymbidium tortisepalum ‘Cang Shan Qi die’, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.8 pp.1061-1066 (doi: 10.5376/mpb.cn.2012.10.0008)
为了克隆兰花捧瓣蝶化的相关基因,揭示蝶化形成的分子机理,本试验用总RNA提取试剂盒提取‘苍山奇蝶’蝶化捧瓣、正格花捧瓣的总RNA,经mRNA纯化试剂盒纯化后,采用Smart技术逆转录成cDNA,经RsaI酶切后,以蝶化捧瓣酶切cDNA作为检测子,正格花酶切cDNA作为驱赶子,成功构建了‘苍山奇蝶’蝶化捧瓣与其正格花捧瓣之间差异表达cDNA文库。生物信息学分析表明:文库包含MADS、假设蛋白、GenBank中未匹配蛋白、储存蛋白以及其它等5类差异表达基因。其中4个基因归为MADS家族中的B亚族。系统进化树分析表明:2个MADS基因归入DEF基因同亚家族,另2个MADS归为PI基因。这些MADS基因的获得为‘苍山奇蝶’捧瓣蝶化相关基因克隆和功能解析奠定了基础。
兰花是一个大家族,总称兰科植物(Orchldaceae)。兰花花结构高度特化,具有花瓣状的萼片和特异的唇瓣以及各种奇异状的花型,尤以蝶花、奇花为著,并于90年代后期开始逐步取代正格的瓣型花、素花成为兰花市场的主流,常是每株售价上百逾万。环境破坏和高价位的兰花使得来源于农村广袤无垠的林地的国兰资源变得极度濒危或十分稀有(吴应祥, 1993; 江泽慧, 2000),因此,探索奇花形成的分子机理等遗传学基础研究,对保护国兰野生资源,培育优良国兰品种具有重要意义。
在兰花中蝶化程度的高低是区别蝶瓣花品质优劣的主要依据,‘苍山奇蝶’为莲瓣兰(Cymbidium tortisepalum)蝶瓣奇花,1990年始发现于云南省大理州境内。此品种捧瓣蝶化面积达90%以上,具有重要的观赏价值和经济价值(刘振龙, 2007)。
研究人员在对拟南芥和金鱼草的各类花器官同形突变体的研究中,发现了在花的发育中调控各类花器官形成的A、B、C、D、E基因。这几类基因中的一类或几类发生突变就会导致萼片变成花瓣,花瓣变成雄蕊等(朱永平等, 2011)。这些基因称为同形异位基因,它们编码的蛋白质均含有一个保守性很强的DNA结合序列——MADS盒,因此属于MADS盒基因家族。该家族每一类基因通过基因复制而产生亚家族, 而后又独自地发生了基因复制事件,产生多个平行进化同源基因(Coen et al., 1991; Pelaz et al., 2000)。
现有的研究揭示了MADS-box等基因在兰花成花转换及花器官发育过程中的重要作用(Lu et al., 1993; Mariana et al., 2008)。近年来,关于兰科植物开花调控的研究取得了一定的进展,已分离鉴定出一些花发育调控基因,包括花器官特异基因及一些花分生组织特异基因,但主要集中在热带兰上,而对中国兰的研究少有报道。对于蝶化奇花基因的研究几乎没有报道。
本试验利用消减抑制杂交技术对‘苍山奇蝶’蝶化捧瓣与其正格花捧瓣之间的差异表达基因进行研究,以期探索中国兰花花型瓣化变异机理,为利用植物基因工程改良中国兰花的观赏性状提供理论依据。
1结果与分析
1.1总RNA及mRNA的质量检测
提取‘苍山奇蝶’蝶化捧瓣和正格花捧瓣的总RNA后,在紫外呈像仪上观察RNA条带。结果显示(图1)总RNA具有28S、18S两条明显的带谱,其完整性较好,且28S和18S的亮度比例约为2:1,表明提取的总RNA可用构建文库。
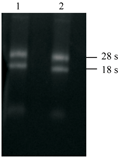 图1 莲瓣兰总RNA Figure 1 Identification of total RNA in Cymbidium tortisepalum |
1.2酶切效果分析
电泳结果图2A表示蝶化捧瓣合成cDNA的片段大小分布与8 000~100 bp之间,经RsaⅠ酶切后,条带明显下移,集中于100~1 000 bp之间,图2B表示正格花捧瓣合成cDNA的片段大小分布与5 000~500 bp之间,经RsaⅠ酶切后,条带明显下移,集中于100~1 000 bp之间,大片段减少、小片段增多。说明酶切效果好,适用于后续试验。
 图2 蝶化捧瓣(A)及正格花捧瓣(B)的酶切和未酶切cDNA Figure 2 The products of undigested and digested cDNA in lip-like petals (A) and normal petals (B) |
1.3抑制消减cDNA文库中cDNA片段大小的检测
构建的正向文库大小为3 000个克隆,反向文库大小为500个克隆。从正向文库中随机挑选15个菌落进行菌落PCR,外源片段的长度分布在100~750 bp左右,平均片段长度约500 bp左右(图3)。以上结果显示,成功构建消减抑制文库。
|
图3 蝶化捧瓣SSH文库的cDNA片段大小分析 Figure 3 Analysis of the length of cDNA fragments in lip-like petals library |
1.4 ESTs功能分类
将唯一序列NCBI在线Blastx进行同源比对及功能分析。114个序列按功能分类为MADS、假设蛋白、GenBank中未匹配蛋白、储存蛋白以及其它等5类(图4)。其中MADS基因所占比例为5%、假设蛋白所占比例为58%、GenBank中未匹配蛋白所占比例为18%、储存蛋白所占比例为6%以及其它所占比例为13%。
.png) 图4 ESTs功能分类图 Figure 4 Functional classifications of all ESTs |
1.5 MADS系统进化树分析
用MEGA软件对筛选到的上调表达的MADS克隆构建系统发育树。从图5中可知,代号为b109、b9的序列与细茎石斛、胡颓子、油菜的PI基因归为一类;代号为b95和b107的序列与凤仙花DEF1和DEF2聚为一类;DEF和PI都属于B类基因,与控制花瓣形成有关。
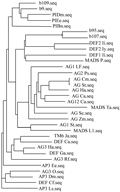 图5 蝶化捧瓣中上调表达MADS基因聚类分析 Figure 5 Clustering analysis of up-regulate expression genes in lip-like petals of peloria flower |
2讨论
本试验利用抑制消减杂交技术,从‘苍山奇蝶’突变捧瓣中分离特异表达的序列,获得4个MADS基因,聚类分析表明:4个MADS基因归为B亚家族基因中的PI基因和DEF基因。
DEF和PI基因为植物花器官发育B类基因,控制植物花瓣的发育,近年来,不同实验室已从多种兰花组织中分离到多个B类基因的平行进化同源基因(Pelaz et al., 2000)。德国学者( Mondragón-Palomino and Theissen, 2008)对已发表的包括兰花在内的所有单子叶植物的DEF基因进行了系统进化及发育方面的分析后,提出了兰科植物花器官属性决定的分子模型,并将之称为“兰花密码”,认为兰花的DEF类基因在早期经过两轮复制,形成了四类基因,第一和第二类DEF基因独立控制花瓣状的萼片形成,第一、第二和第三类DEF基因共同控制捧瓣的形成,第四类DEF基因控制唇瓣的形成。“兰花密码”的提出是一个创新,但兰科植物种类繁多,这一模型并不适用于所有兰科植物(Tsai et al., 2004)。如郑至勤(1996)从姬蝴蝶兰中克隆到两个E类基因PeMADS8和PeMADS,特异地在唇瓣中表达,并在具有三唇瓣突变体的唇瓣中也有表达。郭滨(郭滨等, 2006)从蝴蝶兰(P. hybrida)中克隆到三个PI-like基因,RT-PCR与原位杂交分析表明phP19、PhPI10和PhPI15均在蝴蝶兰的生殖发育阶段表达,在营养,生长阶段不表达,PhPI15在所有花器官中都表达,而PhPI9和phPI10仅仅在唇瓣表达。
本实验中有2个上调表达序列与DEF基因聚为一类,2个上调表达序列与PI基因聚为一类,为平行进化同源基因。这4个基因同时在蝶化捧瓣中上调表达,与“兰花密码”分子模型有不一致性。由此可以推测,兰花的高度特化的花部结构,涉及到比较复杂的分子遗传机制。莲瓣兰名品‘苍山奇蝶’捧瓣突变,有可能与这4个基因的过量表达有关,从而表现出捧瓣蝶化现象。
近年来,不同实验室已从多种兰花中分离到多个MADS基因的平行进化同源基因。说明MADS等基因在兰花的成花转换及花器官形成过程中起重要作用,与兰花结构的特异性及多样性有关。然而,基因表达与花形态相关的试验证据还不够充足,基因表达模式的保守性与特异性还需要深入研究(田敏等, 2011)。本文对序列功能的预测是基于表达序列标签分析,由于这些表达序列标签序列比较短,不能代表基因的全长,所以只能得到部分的基因信息。为了揭示‘苍山奇蝶’蝶化捧瓣变异分子机理,为今后利用植物基因工程改良中国兰花的观赏性状,我们还必须进行后续试验,根据这些EST序列设计引物,通过race技术获得全长目的基因,以便获得目的基因的结构、功能及其表达产物的特性和完整的基因信息。这将为中国兰花花器官高度进化的形态学发育提供分子基础,并有助于人们对其分子调控的理解。
3材料与方法
3.1植物材料
从云南大理购买‘苍山奇蝶’植株,带回实验室后,分别取尚未完全开放的‘苍山奇蝶’及其正格花捧瓣,用干净的锡箔纸包裹后置于液氮4 h后,放入-80℃冰箱保存待用。
3.2主要试剂
RNAiso-mate for Plant Tissue、pMD18-T Vector购自Takara公司;PCR cDNA Synthesis Kit、消减杂交试剂盒购自Clontech公司;Oligotex mRNA Mini Kit 购自QIAGEN公司;2K PlusII DNA Maker购自全式金生物技术有限公司。其它试剂均为国产分析纯产品。
3.3总RNA的提取及mRNA的纯化
按照RNAiso-mate for plant Tissue试剂盒的方法,进行总RNA的提取。参照Oligotex mRNA Mini Kit说明进行Ploy A+ mRNA的分离、收集和纯化。
3.4消减抑制杂交
3.4.1 cDNA的合成及纯化
以‘苍山奇蝶’蝶化捧瓣为检测子,其正格花捧瓣为驱赶子进行差减杂交。以mRNA为模版,SMART 3' CDS Primer II A为引物,在PowerScript Reverse Transcriptase的作用下反转录出cDNA一链。二链的合成参照Advantage 2 PCR Kit的说明进行,循环参数为:95℃ 1 min;95℃ 15 s,65℃ 30 s,27个循环后68℃延伸6 min。二链PCR产物用酚氯仿抽提纯化,纯化产物放于-20℃冰箱保存。
3.4.2酶切及酶切产物的纯化
用Rsa I进行酶切。酶切体系为:43.5 μL cDNA、5 μL 10× Rsa Restriction Buffer、1.5 μL Rsa I (10 U)。37℃培养箱中孵育3 h。用2.5 uL EDTA/Glycogen混合物终止反应。酶切产物用酚氯仿抽提纯化。
3.4.3消减杂交
在1.5 μL的EP管中分别加入0.5 μL driver cDNA、1.5 μL tester cDNA和1 μL杂交缓冲液,将混合液进行第1轮的差减,循环参数为:98℃变性1.5 min,68℃退火8h。第1轮杂交结束后,混合2份杂交液,加入新鲜变性的driver cDNA和1 μL杂交缓冲液,68℃反应12 h。第2轮杂交结束后,将溶液稀释于200 μL稀释缓冲液中,72℃保温7 min,-20℃保存。
3.4.4 Nested PCR
以两轮杂交产物为模板,进行两轮PCR。第一轮PCR条件为:75℃ 5 min,94℃ 25 s;94℃ 10 s,66℃ 30 s,72℃ 1.5 min 27个循环。第二轮PCR条件为:94℃ 10 s,68℃ 30 s,72℃ 1.5 min 27个循环。引物为:Nested PCR primer 1、Nested PCR primer 2R。
3.5消减抑制文库的构建
3.5.1连接
pMD18-T Vector与第2轮PCR产物进行连接,总体系10 μL,在16℃的条件下孵育16 h。
3.5.2转化
取连接产物2 μL,加入6 μL的灭菌水,稀释3倍与Takara公司提供的感受态细胞DH5ɑ (50 μL)结合,混匀后冰上静置30 min、42℃热击90 s,冰上2 min后加入800 μL的SOC,37℃摇床培养1 h (230 r/min)后取80 μL涂于含氨苄青霉素的LB培养基上,37℃过夜培养。
将生长出来的白色菌斑,用已灭菌的牙签挑取菌落,置于装有2 000 μL LB培养液的试管中,37℃摇床培养过夜(转速为230 r/min),保存菌液。
3.5.3菌落PCR
进行菌落PCR,用巢式PCR引物扩增插入片段,检测片段的大小及有无。反应条件为:95℃ 10 min;94℃ 30 s,68℃ 30 s,72℃1 min 27个循环后,72℃延伸10 min。
3.6克隆片段DNA同源比对及进化树构建
挑取150个克隆送上海生工测序,经过去除载体和引物序列的净化处理后,得到135个唯一序列。利用NCBI在线Blast同源比对并利用MEGA软件构建进化树。
作者贡献
和凤美、田璐是本研究的实验设计和实验研究的执行人;武芸芸完成数据分析;朱永平参与实验设计,试验结果分析;和凤美是项目构思者和负责人,指导实验设计,数据分析,论文写作与修改;全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学基金(30860178)资助。作者感谢云南农业大学农业生物多样性应用技术国家工程研究中心全体工作人员在本实验过程中的帮助。感谢同行评审人的评审建议和修改建议。
参考文献
Coen E.S., and Meyerowitz E.M., 1991, The war of the whirls: genetic interactions controlling flower development, Nature, 353: 31
http://dx.doi.org/10.1038/353031a0
Guo B., Chen D.H., Dai W., Wei X., and Ming F., 2006, Cloning and expression analysis of a Phalaenopsis flowering-related gene pPI9, Fudan Daxue Xuebao (Journal of fudan university), 45(3): 277-82 (郭滨, 陈东红, 戴薇, 魏星, 明凤, 2006, 蝴蝶兰花发育相关基因PI9的克隆与表达分析, 复旦大学学报, 45(3): 277-82)
Jian Z.H., 2000, Study on peloria flower of chinese orchid and appreciation of famous variety, Chinese Forestry Press, Beijing, China, pp.87-123 (江泽慧, 中国兰花奇花艺研究及奇花名品鉴赏, 2000, 中国林业出版社, 中国, 北京, pp.87-123)
Lin X., Li B.J., Qin D.H., Guo F.Q., Wu C., and Sun C.B., 2011, Advances in Molecular Biology of orchid Floral Development, Zhongguo Xibao Shengwu Xuebao (Chinese Journal of CellBiology), 33(5): 554-563 (向林,李伯钧, 秦德辉, 郭方其, 吴超, 孙崇波, 2011, 兰花花发育的分子生物学研究进展, 中国细胞生物学学报, 33(5): 554-563)
Liu Z.L., 2007, Appreciation and cultivation of famous varieties in Chinese orchid, Guangxi Science and Technology Press, Nanning, China (刘振龙, 2007, 中国兰花名品新品鉴赏与栽培, 广西科学技术出版社, 中国, 南宁)
Lu Z.X., Wu M., and Lob C.S., 1993, Nucleotide sequence of a flower specific MADS box cDNA clone from orchid, Plan t Mol. Biol., 23(4): 901-904
http://dx.doi.org/10.1007/BF00021545
Mariana M., Palominoa, and Günter T.ß., 2008, MADS about the evolution of orchid flowers, Trends in Plant Science, 13(2): 51-59
http://dx.doi.org/10.1016/j.tplants.2007.11.007
Mondragon P.M., and Theissen G., 2008, MADS about the evolution of orchid flowers, Trends Plant Sci, 13: 51- 59
http://dx.doi.org/10.1016/j.tplants.2007.11.007
Pelaz S., Ditta G.S., Baumann E., 2000, B and C floral organ identity functions require SEPA LLA TA MADS‐box genes, Nature, 405(6783): 200-203
http://dx.doi.org/10.1038/35012103
Tian M., Xu X.Y., and Wang C.X., 2011, Research progress on gene regulation in the flower development of orchid, Zhejiang Nonglin Daxue Xuebao (Journal of Zhejiang Forestry College, 28(3): 494-499 (田敏, 徐小雁, 王彩霞, 2011, 兰花植物花发育的基因调控研究进展, 浙江农林大学学报, 28(3): 494-499)
Tsai W. C., Kuoh C. S., Chuang M. H., Chen W. H., and Chen H.H., 2004, Four DEF like MADS-box genes displayed distinct floral morphogenetic roles in Phalaenopsis orchid, Plant Cell Physiol, 45(7): 831-844
http://dx.doi.org/10.1093/pcp/pch095
Wu Y.X., 1993, Chinese orchid, Chinese Forestry Press, pp.26-64 (吴应祥, 1993, 中国兰花, 中国林业出版社, 26-64)
Zheng Z.Q., 1996, Identification of transcription factors involved in labellum development in Phalaenopsis orchids, Thesis for M.S., Department of Life Sciences, National Cheng Gong University, Supervisor: Cheng H.J., pp.34-35 (郑至勤, 1996, 参与蝴蝶兰唇瓣发育之转录因子研究硕士学位论文, 国立成功大学生命科学院, 导师: 陈虹榉, pp.34-35)
Zhu Y.P., Yang D., Yang X.H., Guan W.L., Wu H.Z., and He F.M., 2011, Screening specific expression gene of multiple labellum of peloria flower and analysis in Cymbidium sinense, Xibei Zhiwu Xuebao (Acta Botanica Boreali-Occidentalia Sinica), 30(11): 1090-1096 (朱永平,杨德, 杨晓虹, 关文灵, 吴红芝, 和凤美, 2011, 墨兰多舌奇花MADS特异表达基因筛选及分析, 西北植物学报, 30(11): 1090-1096)