 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 10 篇 doi: 10.5376/mpb.cn.2012.10.0010
收稿日期: 2012年02月17日 接受日期: 2012年03月06日 发表日期: 2012年04月23日
引用格式(中文):
韩睿等, 2012, 菊芋SRAP-PCR反应体系优化及引物筛选, 分子植物育种(online) Vol.10 No.10 pp.1080-1086 (doi: 10.5376/mpb. cn.2012.10.0010)
引用格式(英文):
Han et al., 2012, Optimization of SRAP-PCR Reaction System and Selection of Primers for Jerusalem Artichoke, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.10 pp.1080-1086 (doi: 10.5376/mpb.cn.2012.10.0010)
本研究以菊芋为材料,通过单因子优化试验和L16(45)正交试验设计对影响SRAP-PCR扩增结果的重要参数进行探讨,建立了适合菊芋SRAP-PCR分析的反应体系,即20 μL反应体系中含10×PCR 扩增缓冲液,2.5 mmol/L Mg2+,0.24 mmol/L dNTPs,0.25 μmol/L正反引物,0.8 U TaqDNA聚合酶和80 ng模板DNA。利用该优化体系进行稳定性检测,并从110 个SRAP 引物组合中筛选出100个具有扩增条带清晰丰富且重复性好的引物组合和22个清晰且多态性高的引物组合,表明该体系稳定可靠,并且能够有效地用于菊芋种质资源鉴定及遗传多样性分析等研究。
菊芋(Helianthus tuberosus L.)俗称洋姜、鬼子姜,为菊科向日葵属多年生草本植物。菊芋具有很强的生态适应性,耐寒、耐旱、耐贫瘠、耐盐碱,地下块茎和地上茎叶除了可做优良的饲料外,块茎富含菊糖,有特殊的保健和抗癌作用,茎秆富含纤维可以做成高密度纤维板(薛延丰等, 2006),具有很高的推广应用和研究价值。目前国内外对菊芋的相关研究大多集中在新品种选育与生态治理(孔涛等, 2009)、食品加工(周正等, 2008)、栽培技术、生理生化基础(钟启文等, 2009; 黄高峰等, 2011)等方面,在分子水平上对其种质资源进行遗传多样性的研究尚未见报道。
相关序列扩增多态性(Sequence—related amplified polymorphism, SRAP)是一种新型的基于PCR的标记系统,为显性标记,是由美国加州大学Li与Quiros博士于2001年提出的(Li and Quiros, 2001)。它针对基因外显子GC含量丰富而启动子、内含子AT含量丰富的特点来设计引物对ORFs (openreading frames)进行扩增,具有简便稳定、重复性好、产率高、便于克隆目标片段等优点,已广泛应用于图谱构建、遗传多样性分析等方面的研究,得到了较满意的结果(张俊卫等, 2011; 李怀志等, 2011)。本研究通过对菊芋SRAP-PCR反应系统中的重要参数先后进行单因子和正交优化,并通过稳定性检测,成功建立其最佳反应体系,同时为后续应用该分子标记技术开展菊芋种质资源遗传多样性等方面的研究奠定基础。
1结果与分析
1.1 Mg2+浓度对SRAP-PCR的影响
Mg2+浓度显著影响着SRAP的扩增效率以及产物的量和特异性,浓度过低会影响PCR扩增产量甚至导致扩增失败,浓度过高则会降低PCR扩增的特异性。本试验通过比较5个Mg2+浓度梯度发现不同浓度间扩增结果存在显著差异。由图1可见,当Mg2+浓度为1.5 mmol/L时,扩增条带较弱;当浓度等于或大于3.0 mmol/L时,扩增条带较少且较弱;当浓度在2.0 mmol/L~2.5 mmol/L之间时,扩增条带较多,谱带清晰,且特异性条带和非特异性条带对比鲜明,因此Mg2+浓度范围应该选择大于等于2.0 mmol/L且小于3.0 mmol/L。
.png)
图1 不同Mg2+浓度的SRAP反应结果 Figure 1 Results of SRAP at different Mg2+ concentrations |
1.2 dNTPs浓度对SRAP-PCR的影响
dNTPs作为SRAP扩增反应的原料,显著影响合成效率和扩增的忠实性。由图2可见,当dNTPs浓度为0.1 mmol/L时,扩增产物谱带较弱;当dNTPs浓度在0.15 mmol/L~0.2 mmol/L时,扩增的条带完整,且清晰明亮;当dNTPs浓度高于0.25 mmol/L时,扩增效果明显减弱,且随着浓度的增加,特异性条带减少。因此dNTPs浓度范围应该在0.15 mmol/L~0.25 mmol/L。
.png) 图2 不同dNTPs浓度的SRAP反应结果 |
1.3引物浓度对SRAP-PCR的影响
引物是PCR特异性反应的关键,在退火时与模板DNA结合,启动DNA扩增。本试验采用相同的上、下游引物浓度,当浓度为0.1 μmol/L时,扩增条带相对较弱;随着引物浓度的增加,条带渐清晰和稳定,且扩增结果相差不大,考虑到节约引物的用量,故引物适宜的浓度范围为0.2 μmol/L~0.4 μmol/L。(图3)
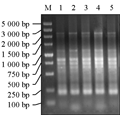 图3 不同引物浓度的SRAP反应结果 |
1.4 Taq DNA聚合酶对SRAP-PCR的影响
TaqDNA聚合酶是SRAP扩增反应的重要参数。本试验中Taq DNA聚合酶浓度对扩增结果影响不大,谱带的扩增效果较一致(图4)。在用量为1.25 U时,扩增条带最为清晰和明亮,其余都有模糊现象,分离界限不够清晰。因此从经济因素考虑,可供选择的Taq DNA聚合酶适宜浓度范围为0.5 U~1.25 U。
.png) 图4 不同Taq DNA聚合酶浓度的SRAP反应结果 |
1.5模板浓度对SRAP-PCR的影响
模板DNA浓度是制约扩增产量及特异性的一个因素,选择适宜的模板量是保证扩增的重要前提。本试验通过比较不同模板浓度对扩增结果的影响,结果显示:在设定的DNA模板质量范围内,都能扩出完整条带(图5),但随着模板浓度的增加,谱带逐渐清晰和明亮,当浓度大于80 ng时,扩增基本相同,为节约模板,我们选用模板浓度的范围为40 ng~80 ng。
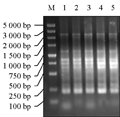 图5 不同DNA浓度的SRAP反应结果 |
1.6 PCR正交试验设计的直观分析
按表3设计的16个处理以me9和em1组合作为引物,对青芋1号菊芋基因组DNA进行SRAP扩增。从图6的电泳结果可以看出,不同组合扩增的结果存在明显差异。13、14、15和16处理扩增条带少且弱,特异性条带缺失,重复不一致,可能由于这四个处理相比其它处理Mg2+浓度较高所致。1、2号处理扩增条带弱,可能由于这两个体系中PCR主要因素的浓度都较低。4、5、6、8和9号处理扩增条带有的很弱,有的有不同程度的弥散现象,也有的特异性条带缺失,可能是这些组合由于正交设计各个因素梯度的不同,其偏离最优组合比较远,导致扩增结果不显著。3、7和10号处理两个重复不一致。11和12号处理,条带清晰明亮,重复一致,但12号处理特异性条带缺失,可能是该处理相对dNTPs浓度过大所致。根据扩增谱带的特异性、数量、亮度的强弱和有无弥散现象进行综合性评价,11号处理扩增清晰度高、条带数多、且特异性条带和非特异性条带对比鲜明,因此可以将其定为最优组合,即20 μL反应体系中含10×PCR 扩增缓冲液,2.5 mmol/L Mg2+,0.24 mmol/L dNTPs,0.25 μmol/L 正反引物,0.8 U TaqDNA聚合酶和80 ng模板DNA。
 图6 正交设计SRAP-PCR反应体系扩增结果 |
1.7稳定性检测
根据上述优化试验的最终结果,以me9/em1引物组合对不同菊芋种质资源模板DNA进行SRAP-PCR扩增,结果表明,10种菊芋材料均能扩增出清晰、多态性好的谱带(图7)。同时,以青芋1号菊芋DNA为模板随机选择10个SRAP引物组合进行扩增,也得到了较好的指纹图谱(图8)。由此可见,优化确定的菊芋SRAP-PCR体系具有稳定可靠、重复性好、多态性较强等特点,可用于菊芋种质资源的分子鉴定和遗传多样性分析等研究。
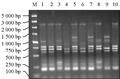 图7 引物me9/em1对10个模板DNA的扩增结果 |
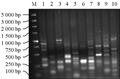 图8 10个SRAP引物组合的扩增结果 |
1.8 SRAP引物筛选
以青芋1号、青芋2号及青芋3号菊芋DNA为模板,利用优化的反应体系对表2设计的所有SRAP引物进行扩增。结果表明:110对引物组合均能扩增出条带,其中100对扩增出的谱带清晰稳定且丰富,所占比率为90.91%,说明SRAP分子标记有很强的适用性;本试验共以3个菊芋DNA为模板能够筛选出22对具有多态性的引物组合,多态率为20%,相对多态性较高。其中,部分引物组合扩增结果见图9。
 图9 部分引物组合扩增结果 |
2讨论
影响SRAP扩增结果的因素有很多,不同物种的反应条件各不相同,因此,应用SRAP标记时应该首先对其反应体系进行优化。国内有关SRAP-PCR反应体系优化的研究大多仍采用单因素试验方法,需要进行多次梯度试验,过程繁琐且不能兼顾到各因素间的交互作用(杨珺等, 2010; 李双双等, 2007)。为了提高分析的准确性,并且获得清晰、稳定性好、重复性高的SRAP-PCR谱带,本试验首先采用单因素方法确定各因素的浓度范围,降低单个因素浓度对试验结果的影响,再通过正交设计的方法,对菊芋SRAP-PCR 反应体系从Mg2+、dNTPs、引物、Taq DNA 聚合酶及模板5 种因素在4个水平上进行优化,进一步降低各因素间互作效应对结果的干扰,从而提高试验的精确性。
在菊芋SRAP体系优化的过程中,发现Mg2+、dNTPs及模板DNA浓度会显著影响SRAP 扩增效果,而引物浓度和Taq DNA聚合酶浓度对扩增无显著影响,优化时可以考虑经济因素等原因选择适宜的浓度。但本试验优化体系中各组分的用量与菊苣(罗燕等, 2010)、芦笋(盛文涛等, 2010)、苎麻(刘立军等, 2006)等植物的用量均有不同,这可能是由于不同植物所用试剂产地及基因组大小不同所致。通过优化,本课题组成功建立了菊芋SRAP-PCR最佳反应体系,即20 μL反应体系中含10×PCR 扩增缓冲液,2.5 mmol/L Mg2+,0.24 mmol/L dNTPs,0.25 μmol/L 正反引物,0.8 U TaqDNA聚合酶和80 ng模板DNA,该体系在各菊芋材料和不同的引物组合中均表现出良好的稳定性、重复性及丰富的多态性。同时,利用该体系进一步筛选出一批带型清晰、稳定性强且重复性好的SRAP引物对,表明该优化体系能够满足菊芋SRAP-PCR 扩增的要求,同时也能够很好的进行后续实验。目前,本课题组正在利用该体系开展菊芋种质资源遗传多样性的研究,已经获得了较好的研究结果。
3材料与方法
3.1材料
本试验选用的材料为“青芋1号”、“青芋2号”、“青芋3号”菊芋及相关菊芋种质资源,种植于青海省农林科学院园艺所菊芋资源圃。相关菊芋种质资源材料见表1。
.png) 表1 供试菊芋种质资源的名称及来源 |
3.2试剂
110对SRAP引物参考Li等提出的原则设计合成,具体序列见表2,由南京金斯瑞生物科技有限公司合成。Taq DNA 聚合酶及dNTPs购自北京全式金生物技术有限公司,DNA Marker购自广州东盛生物科技有限公司。
.png) 表2 SRAP所用引物序列 |
3.3 DNA提取
在研钵中加入少许石英砂和少许PVP干粉研磨30 mg硅胶干燥的菊芋叶片,迅速移入装有已65℃预热的700 uL 2×CTAB提取液(100 mmol/L Tris-HCl PH 8.0, 20 mmol/L EDTA, 1.4 mol/L NaCl, 2% CTAB, 使用前加入0.5% β-巯基乙醇)的1.5 ml离心管中,充分混匀后于65℃水浴保温30 min,不时将离心管上下颠倒摇匀。冷却至室温,加入等体积的氯仿/异戊醇(24:1),颠倒混匀10 min,使内含物形成乳浊液,10 000 r/min,离心10 min 取上清转入新的1.5 mL的离心管中,再次加入等体积的氯仿/异戊醇抽一次。取上清加入2/3体积预冷的(-20℃)异丙醇,于-20℃的冰箱中放置30 min或过夜。13 000 r/min,离心10 min后倒出上清液收集沉淀,加入75%的乙醇500 μL清洗沉淀,10 000 r/min,离心6 min,收集沉淀,同样的方法再洗一遍后将沉淀于室温下自然风干,加入100 μL的TE缓冲液(10 mmol/L , Tris-HCl, pH 8.0, 1.0 mmol/L EDTA)溶解DNA,用2 μL RNaseA 于37℃保温30 min,得到的DNA用0.8%琼脂糖凝胶电泳,以λDNA HindⅢ为标准,检查所得DNA的分子量、含量、纯度及完整性,作为母液4℃或-20℃保存备用。
3.4 SRAP-PCR基本反应体系及反应程序
20 uL PCR反应体系包括:10×扩增缓冲液2 μL;MgCl2 (25mmol/L) 2 μL;dNTPs (2.5 mmol/L) 2.0 μL;引物(10 μmol/L) 0.6 μL,模板DNA (50 ng/μL) 1 μL;DNA聚合酶(2.5 U/L) 0.5 μL;加入ddH2O 补齐到20 μL。反应程序:94℃预变性5 min;进入5个循环,94℃变性1 min,35℃复性1 min,72℃延伸1 min;再进入35个循环,94℃变性1 min,50℃复性1 min,72℃延伸1 min;然后 72℃延伸10 min,4℃保存。
3.5 SRAP-PCR反应体系优化
3.5.1单因素优化
在基本反应体系中分别将Mg2+、dNTPs、引物、Tag DNA聚合酶及模板 DNA各因子浓度作如下调整:Mg2+浓度分别为1.5 mmol/L、2.0 mmol/L、2.5 mmol/L、3.0 mmol/L和3.5 mmol/L;dNTPs浓度分别为0.1 mmol/L、0.15 mmol/L、0.2 mmol/L、0.25 mmol/L和0.3 mmol/L;引物用量分别为0.1 μmol/L、0.2 μmol/L、0.3 μmol/L、0.4 μmol/L和0.5 μmol/L;Tag DNA聚合酶用量分别为0.5 U、1.0 U、1.25 U、1.5 U和2.0 U;模板DNA用量分别为20 ng、40 ng、60 ng、80 ng和100 ng,从而进行单因子多水平试验。
3.5.2正交试验设计
采用L16(45)正交试验设计,按照表3的方案将Mg2+浓度、dNTP浓度、引物浓度、TaqDNA聚合酶浓度以及DNA模板浓度进行正交组合。
根据表3配制20 μL总体积的PCR反应体系,每管除表中的变化因素外还含有2 μL 10×PCR buffer,其余用ddH2O补足20 μL,每处理设2次重复。PCR反应结束后,产物在2.0%琼脂糖凝胶中电泳,经EB染色后,凝胶成像系统进行拍照分析。
.png) 表3 SRAP-PCR反应体系的正交试验设计 |
3.6菊芋SRAP-PCR反应体系稳定性检测
利用优化的体系,以me9/em1为引物选取10种菊芋DNA模板分别进行扩增,并以“青芋1号”菊芋DNA为模板随机选择10种SRAP引物,进行菊芋SRAP-PCR反应体系的稳定性检测。
作者贡献
韩睿、赵孟良、马胜超和李莉是本研究的实验设计和实验研究的执行人;韩睿、赵孟良及马胜超完成数据分析,论文初稿的写作;李莉参与实验设计,试验结果分析,指导论文修改;韩睿是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家大宗蔬菜产业技术体系西宁综合试验站项目(CARS-25-G-49)资助。作者感谢青海省农林科学院菊芋研发中心各位老师在本实验过程中的帮助。
Huang G.F., Wang L.H., Fang Y.H., and Li L., 2011, Effects of drought stress on protective enzyme activity and membrane lipid peroxidation of leaf in Helianthus tuberosus L. seedling, Xinan Nongye Xuebao (Southwest China Journal of Agricultural Sciences), 24(2): 552-555 (黄高峰, 王丽慧, 方云花, 李莉, 2011, 干旱胁迫对菊芋苗期叶片保护酶活性及膜脂过氧化作用的影响, 西南农业学报, 24(2): 552-555)
Kong T., Wu X.Y., Liu L.L., and Yan H., 2009, Comparison study on growth rhythm and photosynthesis characteristics for two varieties of jerusalem artichoke in wind-sandy land, Shanxi Nongye Kexue (Journal of Shanxi Agricultural Sciences), 37(7): 40-43, 47 (孔涛, 吴祥云, 刘玲玲, 闫晗, 2009, 风沙地2种菊芋生长节律及光合特性的比较研究, 山西农业科学, 37(7): 40-43, 47)
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103(2-3): 455-461
http://dx.doi.org/10.1007/s001220100570
Li H.Z., Zhang J., Li X., and Chen H.Y., 2011, Genetic diversity analysis and fingerprinting of eggplant cultivars with SRAP markers, Nanjing Nongye Daxue Xuebao (Journal of Nanjing Agricultural University) 34(4): 18-22 (李怀志, 张峻, 李翔, 陈火英, 2011, 应用 SRAP 标记对茄子品种进行遗传多样性分析与指纹图谱构建, 南京农业大学学报, 34(4): 18-22)
Li S.S., Zhao R.H., Yan G.Q., and Hao G., 2007, Establishment & optimization of SRAP analysis system of potato genome, Shanxi Shifan Daxue Daxue Xuebao (Journal of Shanxi Normal University), 21(4): 79-83 (李双双, 赵瑞华, 闫桂琴, 郜刚, 2007, 马铃薯基因组SRAP反应体系的建立和优化, 山西师范大学学报, 21(4): 79-83)
Liu L.J., Meng Z.Q., Xing X.L., and Peng D.X., 2006, Optimization for SRAP reaction system in ramie (Boehmeria nivea L. Guad), Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 4(5): 726-730 (刘立军, 蒙祖庆, 邢秀龙, 彭定祥, 2006, 苎麻基因组SRAP扩增体系的优化研究, 分子植物育种, 4(5): 726-730)
Luo Y., Bai S.Q., Peng Y., Zhang Y., and Ma X., 2010, Genetic diversity of cichorium intybus germplasm resources using sequence-related amplified polymorphism (SRAP) molecular markers, Caoye Xuebao (Acta Prataculturae Sinica), 19(5): 139-147 (罗燕, 白史且, 彭燕, 张玉, 马啸, 2010, 菊苣种质资源遗传多样性的SRAP研究, 草业学报, 19(5): 139-147)
Sheng W.T., Zhou J.S., Tang Y.P., Luo S.S., and Chen G.Y., 2010, Optimization of SRAP reaction system and selection of primers for Asparagus officinalis L., Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 8(3): 612-618 (盛文涛, 周劲松, 汤泳萍, 罗绍春, 陈光宇, 2010, 芦笋SRAP反应体系优化及引物筛选, 分子植物育种, 8(3): 612-618)
XueY.F., Liu Z.P., Zheng Q.S., ChenM.D., Liu L, and Yan Y.N., 2006, Abating effect of calcium ion on seawater-stressed jerusalem artichoke (Helianthus tuberosus L.), Xibei Zhiwu Xuebao (Acta Botanica Boreali-Occidentalia Sinica), 26(6): 1267-1271 (薛延丰, 刘兆普, 郑青松, 陈铭达, 刘玲, 严一诺, 2006, 钙离子对菊芋海水胁迫的缓解效应研究, 西北植物学报, 26(6): 1267-1271)
Yang J., Ren Y., Yang G.S., and Yin J.M., 2010, Establishment and optimization of sequence-related amplified polymorphism system for Phalaenopsis, Redai Nongye Kexue (Chinese Journal of Tropical Agriculture, 30(3): 21-24) (杨珺, 任羽, 杨光穗, 尹俊梅, 2010, 蝴蝶兰SRAP反应体系的建立与优化, 热带农业科学, 30(3): 21-24)
Zhang J.W., Mao Q.S., and Bao M.Z., 2011, Analysis of genetic diversity among germplasm of Prunus mume Sieb. et Zucc. using SRAP markers, Yuanyi Xuebao (Acta Horticulturae Sinica), 38(1): 117-124 (张俊卫, 毛庆山, 包满珠, 2011, 梅遗传多样性的SRAP分析, 园艺学报, 38(1): 117-124)
Zhong Q.W., Liu S.Y., Wang L.H., Wang Y., and Li L., 2009, Absorption, accumulation and allocation of nitrogen, phosphorus and potassiumof jerusalem artichoke, Zhiwu Yingyang Feiliao Xuebao (Plant Nutrition and Fertilizer Science), 15(4): 948-952 (钟启文, 刘素英, 王丽慧, 王怡, 李莉, 2009, 菊芋氮, 磷, 钾吸收积累与分配特征研究, 植物营养与肥料学报, 15(4): 948-952)
Zhou Z., Cao H.L., Zhu Y., Li S.G., Bai X.F., Zhao X.M., and Du Y.G., 2008, The primary study of ethanol production by fermenting of jerusalem artichoke instead of corn, Xibei Nongye Xuebao (Acta Agriculturae Boreali-occidentalis Sinica), 17(4): 297-301, 305 (周正, 曹海龙, 朱豫, 李曙光, 白雪芳, 赵小明, 杜昱光, 2008, 菊芋替代玉米发酵生产乙醇的初步研究, 西北农业学报, 17(4): 297-301, 305)