 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 16 篇 doi: 10.5376/mpb.cn.2012.10.0016
收稿日期: 2012年03月01日 接受日期: 2012年03月24日 发表日期: 2012年04月30日
孙祖东等, 2012, SRAP分子标记在大豆抗豆卷叶螟研究中的应用, 分子植物育种(online) Vol.10 No.16 pp.1122-1126 (doi: 10.5376/mpb. cn.2012.10.0016)
引用格式(英文):
Sun et al., 2012, Application of SRAP Marker in Soybean Resistance to Bean Pyralid (Lamprosema indicata Fabricius), Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.16 pp.1122-1126 (doi: 10.5376/mpb.cn.2012.10.0016)
相关序列扩增多态性(SRAP)是一种新型的分子标记,该标记操作简便,具有高度共显性,在基因组中分布均匀,便于克隆测序目标片段,可用于基因定位,基因克隆,遗传图谱构建等。SRAP分子标记能成功应用到大豆作物研究中前提,必需要建立起一个优良稳定有效的SRAP-PCR反应体系,以便确定SRAP标记结果是有效可信的。本研究建立一个有效稳定优良的SRAP-PCR体系,即20 μL的反应体系中各组分为:Mg2+浓度为2.0 mmol/L,各dNTP的浓度0.20 mmol/L,Taq酶为1.5 U,上下游引物的浓度均为0.5 μmol/L,DNA为50 ng。本研究设计了27个正向引物,40个反向引物,两两组合后有1080对引物组合,研究结果显示在12份抗感大豆材料具有多态性的引物组合有516对,是所有引物组合的47.8%,在可扩增出产物的引物组合中占60.6%。
SRAP即相关序列扩增多态性(sequence-related amplified polymorphism),是一种基于PCR反应的新型分子标记系统,于2001年由美国加州大学蔬菜系Li和Quios (Li and Quios, 2001)开发出来的,也被称为SBAP即基于序列扩增多态性(sequence-based amplified polymorphism)。
这种新型分子标记系统(sequence-related amplified polymorphism)的分子标记引物由一段核心序列、“填充”序列和选择性碱基组成,基因外显子中的GC含量高则启动子与内含子中的AT含量高,引物就是根据这个理论进行设计的。
基于PCR的分子标记技术SRAP,通过对引物的独特设计来扩增ORFs,正反向引物的长度分别为17 bp,18 bp,正向引物用来扩增外显子,反向引物特异扩增启动子区域、内含子区域。个体不同导致启动子、内含子和间隔区长度的不同,从而产生了SRAP的多态性。SRAP在对分子遗传图谱的构建、遗传多样性研究及种质资源的鉴定、作物起源与进化关系研究上以及作物目标性状连锁标记研究上都有广泛的应用(李严和张春庆, 2005; 柳李旺等, 2005)。
SRAP的操作简便,具有高度共显性,遍布整个基因组,在基因组中分布均匀,便于克隆测序目标片段,可用于基因定位、基因克隆、遗传图谱构建等。SRAP标记最早是应用于芸薹属作物的研究,随后被迅速应用到各种作物的研究中,目前在水稻、油菜、番茄、棉花、马铃薯、大蒜、柑橘、辣椒、小麦、拟南芥等(林忠旭等, 2003; 王凤涛等2009; 王刚等,2004, 中国科学C 辑, 34 (6): 510-516; 杜晓华等, 2006; 张丽等, 2007; 张安世等, 2010)作物研究中成功应用。本文将SRAP应用于大豆抗豆卷叶螟研究上。
1结果与分析
1.1 Mg2+浓度对扩增反应的影响
试验设置Mg2+在反应体系中的浓度梯度为五个:1 mmol/L、1.5 mmol/L、2.0 mmol/L、2.5 mmol/L、3.0 mmol/L。反应产物经丙烯酰胺凝胶电泳分离后结果表明,当Mg2+浓度在1.0 mmol/L~3.0 mmol/L时,均能扩增出产物。而当Mg2+浓度在1.0 mmol/L~2.0 mmol/L时,产物量多,条带越来越清晰;而Mg2+浓度在2.5 mmol/L~3.0 mmol/L时,产物量下降,带型模糊。反应体系中Mg2+浓度直接影响体系中Taq酶活性,体系中Mg2+不足则Taq酶活性不足,反应产物少;若体系Mg2+过量,则过量的Mg2+会与dNTP络合,反应产物同样会减少。因此,本试验反应体系中Mg2+浓度2.0 mmol/L时为最佳反应浓度。
1.2 dNTP浓度对扩增反应的影响
dNTP在反应体系中的浓度直接或间接影响扩增产物量得多少,体系中dNTP不足,扩增产物少,而过多的dNTP则会与Taq酶竞争Mg2+,从而影响Taq酶的活性,Taq酶活性降低则产物量减少。本试验设置各dNTP的浓度梯度为:0.05 mmol/L、0.10 mmol/L、0.15 mmol/L、0.20 mmol/L、0.25 mmol/L。结果表明,当体系中各dNTP的浓度从0.05 mmol/L~0.20 mmol/L梯度上升时,条带清晰明显;当dNTP浓度达到0.25 mmol/L时反应产物量下降。因此在dNTP的浓度选择上,本试验最佳浓度为0.20 mmol/L。
1.3 Taq酶浓度对扩增反应的影响
Taq酶在PCR中起到关键作用。在反应中加入过多的Taq酶,会产生非特异性扩增;过少则直接导致产物量合成少。本试验设置的Taq酶梯度为:0.5 U、1.0 U、1.5 U、2.0 U、2.5 U。由图3可见,随着Taq酶的增加,扩增产物量明显增多,当Taq酶加入量达到2.0 U时出现了非特异性扩增,且泳带拖尾现象明显。因此,从本试验的结果可得出,体系20 μL时,Taq酶加入量为1.5 U时为反应最佳条件。
1.4稳定性检验
由以上试验结果可知,本研究建立大豆最优SRAP-PCR反应体系,即20 μL的反应体系中各组分为:Mg2+浓度为2.0 mmol/L,dNTP的浓度0.20 mmol/L,Taq酶为1.5 U,正、反向引物的浓度均为0.5 μmol/L,DNA为50 ng。利用该体系对12份大豆对卷叶螟具有抗感性材料进行扩增,经6%聚丙烯酰胺凝胶上进行电泳分离,谱带清晰重复性好如图1所示。这说明该优化SRAP体系稳定可靠,可用于研究大豆分子标记分析。
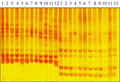 图1 引物Me19Em1及Me19Em2扩增产物SRAP图谱 Figure 1 SRAP finger print of 12 soybean varieties genetated by primers Me19-Em1 and Em19-Em2 |
1.5引物扩增多态性
反应体系经优化后,以12份抗感大豆材料为模板,用27个正向引物,40个反向引物的1 080对引物组合进行扩增,PCR扩增产物经凝胶电泳,银染结果显示:具有清晰条带的引物有866对,占所有标记数的80.2%;其中具有多态性的引物为516对,是所有引物组合的47.8%,在可扩增出产物的引物组合中占60.6%,无法扩增出产物的引物组合为214对,占所有引物组合的19.8%,见表1。
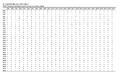 表1 抗感材料扩增出PCR产物的引物组合 Table 1 The primer combinations producd on every two soybean varieties |
2讨论
新型分子标记SRAP有很多特点,如高多态性、好的重复性、高产率、操作方法简单、均匀分布于基因组中、扩增得到的目的片段测序容易、引物有一定的通用性,而且其正反向引物可以相互搭配组合,用少量引物能组配成多个引物对,使引物的使用效率提高。SRAP标记已在油菜、马铃薯、水稻、生菜、大蒜、苹果、樱桃、柑橘与芹菜等(Zudong et al., 2007; Mukhlesur et al., 2008; 王凤涛等, 2009; 林忠旭等, 2003; 杜晓华等, 2006; 张丽等, 2007;张安世等, 2010; 王刚等,2004, 中国科学C 辑, 34 (6): 510-516)作物的分子标记研究中,广泛用于种质资源的鉴定评价、遗传图谱的构建、基因组的转录图谱绘制、重要性状的基因标记、基因克隆与标记测序。
目前,SRAP标记在大豆中应用的报道还比较少。周春娥等使用SRAP标记进行了大豆种质多样性分析(周春娥等, 2012, 江苏农业科学, 40(1): 40-42)。王贤智等(2008)利用SRAP标记与SSR、AFLP标记对大豆重组自交系群体进行大豆多态性标记构建遗传连锁图谱,其中SRAP标记的引物为170对,亲本间有多态性条带的有23对,获得了关于大豆含油量QTL SRAP分子标记14个。此外还获得了2个关于叶型和茸毛色的形态标记。但是该报道称SRAP标记应用于大豆扩增效果不理想,重复性差。因此,在大豆研究中应用SRAP标记需要进行反应体系的优化,建立一个稳定优良有效的SRAP-PCR反应体系是十分必要的。
本研究中,进行多次的体系优化,在反应时间上也进行了多次的校正,反复进行了稳定性与重复性试验,最后获得了较理想的SRAP-PCR反应条件与反应体,即20 μL的反应体系中各组分为:Mg2+浓度为2.0 mmol/L,各dNTP的浓度0.20 mmol/L,Taq酶为1.5 U,引物浓度为0.5 μmol/L,DNA为50 ng。在引物筛选上,用27条正向引物和40条反向引物组成1 080对引物组合,经过筛选和验证,获得516对具有多态性的引物组合,这些引物组合可用于分子标记或进行遗传图谱的构建,SRAP分子标记在大豆研究上应用少,为以后的研究工作节时省力,提高研究效率提供帮助。
3材料与方法
3.1实验材料
用于本研究的材料是经过多年鉴定,对豆卷叶螟高抗的大豆品种牛黄豆,栾川城关春小黑豆,吴江青豆3,赶泰-2-2,东兴青皮,丰平黑豆和高感觉的大豆品种Morsoy,Bethel,皖82-178,大浪中团黄豆,山东大豆,河洲黄豆金龙黑豆,那坡黑眼豆。
3.2试验方法
3.2.1总DNA的提取
在苗期随机选取新鲜叶片,采用CTAB法提取总DNA,用6%琼脂糖凝胶电泳检测DNA质量,RNA/DNA浓度电度测定仪-20℃保存备用。
3.2.2 PCR扩增引物
根据SRAP标记特性进行引物设计,共设计有正向引物有27个,反向引物有40个,共组成1 080对SRAP引物组合,见表2,所有引物均是由上海捷瑞生物工程有限公司合成。
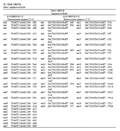 表2 SRAP引物序列 Table 2 sequences of SRAP |
3.2.3 PCR扩增
以丰平黑豆与Bethel为模板,对Mg2+、dNTP、Taq酶、引物分别设置5个不同的浓度梯度,进行20 μL PCR反应体系扩增优化。反应过程为:预变性94℃ 4 min,变性94℃ 50 s,复性35℃ 50 s,延伸72℃ 1 min,循环数是5个;变性94℃ 50 s,退火50℃ 50 s,延伸72℃ 1 min,共35循环,循环后,继续72℃延伸10 min,产物保存于4℃。
3.2.4产物检测
以丰平黑豆(抗)与Bethel (感)等(孙组东等, 2005)为父母亲本作对照进行SRAP分析,检测各引物组合进行SRAP-PCR的有效性。产物中加入Loading Buffer 3.2 μL,电泳检测用8%非变性聚丙烯酰胺凝胶,200V电压,0.5×TBE电泳缓冲液,1 h电泳,银染显影后,拍照保存。
作者贡献
孙祖东是项目的构思着和负责人,指导实验设计,数据分析,论文写作与修改;龙继凤、唐兴富、杨微是本实验研究的执行人。龙继凤负责分子标记实验,与第一作者同等贡献;唐兴富负责田间试验;杨微参加田间试验,负责实验数据整理与分析。全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学基金项目(30871545),广西回国基金项目(桂科回0832020)和广西农业科学院基本业务费项目(201004Z(基))共同资助。
参考文献
Du X.H., Gong Z.H., Wang D.Y., and Yin Q.M., 2006, Genetic differences among well-performing inbred lines of hot pepper (Capsicum annuum L.), Xibei Zhiwu Xuebao (Acta Botanica Boreali-Occidentalia Sinica), 26(12): 2445-2452 (杜晓华, 巩振辉, 王得元, 殷秋妙, 2006, 辣椒优良自交系间遗传差异的分子分析, 西北植物学报, 26(12): 2445-2452)
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: Its application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103: 455-461
http://dx.doi.org/10.1007/s001220100570
Li Y., and Zhang C.Q., 2005, A molecular marker-SRAP technique optimization and application analysis, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 21(5): 108-112 (李严, 张春庆, 2005, 新型分子标记—SRAP技术体系优化及应用前景分析, 中国农学通报, 21(5): 108-112)
Lin Z.X., Zhang X.L., Nie Y.C., He D.H., and Wu M.Q., 2003, Construction of SRAP genetic linkage map in cotton, Kexue Tongbao (Chinese Science Bulletin), 8(15): 1676-1679 (林忠旭, 张献龙, 聂以春, 贺道华, 吴茂清, 2003, 棉花SRAP遗传连锁图构建, 科学通报, 48(15): 1676-1679)
Liu L.W., Gong Y.Q., Huang H., and Zhu X.W., 2005, Novel molecular marker systems——SRAP and TRAP and their application, Yichuan (Hereditas), 26(s): 777-751 (柳李旺, 龚义勤, 黄浩, 朱献文, 2005, 新型分子标记—SRAP与TRAP及其应用, 遗传, 26(5): 777-781)
Rahman M., Sun Z., McVetty P.B., and Li G., 2008, High throughput genome-speciWc and gene-speciWc molecular, markers for erucic acid genes in Brassica napus (L.) for marker-assisted selection in plant breeding, Theor. Appl. Genet., 117(6): 895-904
http://dx.doi.org/10.1007/s00122-008-0829-9 PMid:18633592
Sun Z.D., Wang Z.N., Tu J.X., Zhang J.F., Yu F.Q., McVetty P.B., and Li G.Y., 2007, An ultradense genetic recombination map for Brassica napus, consisting of 13551 SRAP markers, Theor. Appl. Genet., 114(8): 1305-1317
http://dx.doi.org/10.1007/s00122-006-0483-z PMid:17426959
Wang F.T., Lin R.M., Ouyang H.Y., and Xu S.C., 2009, The application of SRAP-PCR in genetic diversity study of Henan wheat cultivars, Zhiwu Yichuan Ziyuan Xuebao (Journal Of Plant Genetic Resources), 10(4): 517-521 (王凤涛, 蔺瑞明, 欧阳宏雨, 徐世昌, 2009, 利用SRAP标记分析河南小麦栽培品种的遗传多样性, 植物遗传资源学报, 10(4): 517-521)
Wang X.Z., Zhou R., Zhang X.J., Shan Z.H., Sha A.H., Chen H.F., Qiu D.Z., Li P.W., and Zhou X.A., 2008, Map construction and QTL analysis of oil content in soybean, Zhongguo Youliao Zuowu Xuebao (Chinese Journal of Oil Crop Sciences), 30(3): 272-278 (王贤智, 周蓉, 张晓娟, 单志慧, 沙爱华, 陈海峰, 邱德珍, 李培武, 周新安, 2008, 大豆遗传图谱的构建和含油量的QTL分析, 中国油料作物学报, 30(3): 272-278)
Zhang A.S., Xu J.W., Xin C.J., Zhang L.M., Xing Z.F., and Wei H.Y., 2010, Analysis of genetic diversity of new middle-late lines of japonica rice in henan province by srap, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 26(2): 50-54 (张安世, 徐九文, 辛长永, 张利民, 邢智峰, 韦慧彦, 2010, 河南省水稻中晚粳新品系遗传多样性的SRAP分析, 中国农学通报, 26(2): 50-54)
Zhang L., Jiang S.K., Zhang X.J., Li L., Guo Z.F., Ma H., Chen L.J., Wang X.Y., Xu Z.J., and Zhong M., 2007, Construction and analysis of a SRAP molecular genetic map of japonica rice (Oryza sativa L. sp. japonica), Zhiwu Shenglixue Tongxun (Plant Physiology Communications), 43(3): 443-447 (张丽, 姜树坤, 张喜娟, 李丽, 郭志富, 马慧, 陈丽静, 王学英, 徐正进, 钟鸣, 2007, 粳稻SRAP分子标记遗传群的构建与分析, 植物生理学通讯, 43(3): 443-447)