 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 38 篇 doi: 10.5376/mpb.cn.2012.10.0038
收稿日期: 2012年06月02日 接受日期: 2012年06月14日 发表日期: 2012年08月07日
引用格式(中文):
吕运舟等, 2012, 参与杉木次生壁合成调控的转录因子ClMYB4的克隆及在大肠杆菌中表达, 分子植物育种(online) Vol.10 No.38 pp.1278-1286 (doi: 10.5376/mpb.cn.2012.10.0038)
引用格式(英文):
Lv et al., 2012, Cloning Transcription Factor ClMYB4 Involving in Secondary Cell Wall Biosynthesis from Cunninghamia lanceolata (Lamb.) Hook and expressing in E. Coli, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.38 pp.1278-1286 (doi: 10.5376/mpb.cn.2012.10.0038)
植物次生壁及木质素生物合成过程在转录水平上受到某些R2R3-MYB基因家族成员的调控,如AtMYB46和PtMYB4。本研究在杉木发育木质部中克隆了一个全长为1 453 bp的R2R3-MYB基因cDNA序列,编码413个氨基酸残基的预测蛋白。生物信息学分析发现它与火炬松PtMYB4的序列相似性最高,且与AtMYB46、PtMYB4和EgMYB2次生壁合成调控基因在系统进化上聚为一类。因此,命名为ClMYB4。本实验构建了ClMYB4基因的原核表达载体pET-30b-ClMYB4,并在大肠杆菌Rosetta中实现了高效表达和纯化,为进一步研究转录因子ClMYB4在调控杉木次生壁发育过程的下游靶基因及相关顺式作用元件提供基础。
研究木材发育过程是了解木材的品质和产量形成机制的重要途径。木材发育过程是一个高度有序的过程,在生物学上可以分为形成层细胞分裂,细胞伸展增大,次生壁加厚,木质化及细胞程序化死亡5个事件(Hertzberg et al., 2001)。这些生物学事件是在很多转录因子的交互调控下完成的。根据已有的研究报道,主要有以下几类转录因子参与了木质部的发育:生长素应答因子(ARF),同源盒亮氨酸拉链Ⅲ (HD-ZIP Ⅲ),MYB (MYB domain gene),NAC (NAC domain gene)和WOX (WUS homeobox gene) (Ohashi-Ito and Fukuda, 2010)。模式植物拟南芥中鉴定的转录因子AtMYB46被证明是次生细胞壁化学组分纤维素、木质素和木酮糖生物合成的共有调控基因(Zhong et al., 2007),而AtMYB58/AtMYB63是木质素合成通路的直接调控转录因子(Zhou et al., 2009)。在林木的木材发育过程中也发现一些MYB类转录因子参与调控次生壁生物合成及木质素的生物合成,如火炬松中的PtMYB1、PtMYB4和PtMYB8 (Patzlaff et al., 2003a; 2003b; Bomal et al., 2008),桉树中的EgMYB2 (Goicoechea et al., 2005),杨树中的PtrMYB3、PtrMYB20 (McCarthy et al., 2010)和PtrMYB28 (Zhong and Ye, 2009)。它们可以与木质素生物合成基因的顺势调控元件AC序列相结合,正向调节木质素单体的生物合成过程。火炬松PtMYB1、PtMYB4、PtMYB8和桉树EgMYB2基因都在植株次生壁发生加厚的发育木质部中表达,这些基因的过量表达都可以激活转基因植株次生壁生物合成过程,造成相关基因的异位表达和木质素的沉积(Patzlaff et al., 2003a; 2003b; Bomal et al., 2008; Goicoechea et al., 2005)。
杉木是我国南方最重要的造林树种,以生长迅速、材质优良而著称,尤其是心材棕红色的木材尤为珍贵。但是对杉木优质木材形成过程,如次生壁加厚及木质素生物合成的分子机理研究未见报道。编号为“6421”的杉木优树无性系,于1964年选自福建省洋口国有林场的一片约30年生的人工林。不同时期的遗传测定表明,杉木无性系“6421”不同性状的一般配合力和特殊配合力,均具有突出的表现。围绕着从分子水平上解析该无性系优良特性的形成机理这一目标,在前期常规育种的基础上,项目组用该无性系进行了转录组分析。本文首次报道,在杉木发育木质部中克隆了一个MYB基因,其序列及编码蛋白预测显示ClMYB4属于典型的植物R2R3-MYB转录因子,推测其可能与杉木次生壁加厚和木质素合成的调控过程相关。此外,本文通过构建原核表达载体pE-30b-ClMYB4,转化大肠杆菌菌株Rosetta,SDS-PAGE电泳观察不同浓度IPTG (异丙基-β-D-硫代吡喃半乳糖苷)对重组蛋白表达的诱导效果,利用His-tag亲和技术成功纯化了ClMYB4重组蛋白。ClMYB4及其重组蛋白的成功表达,为下一步研究杉木木材发育次生壁加厚过程的转录调控机理奠定了基础。
1结果与分析
1.1 ClMYB4全长cDNA的获得
通过本地Blast分析,我们获得了一条1 200 bp左右的contig,与火炬松PtMYB4同源。分析发现其5'序列不完整。经过5'RACE PCR扩增,我们获得了一条约600 bp的特异条带(图1A)。克隆后分析测序结果,得到与以上contig部分重叠的400 bp 长的DNA片段,并含有起始密码子ATG,表明该片段为目的基因的5'端序列。以杉木发育木质部cDNA第一链为模板,采用特异引物MYB4-F/MYB4-R扩增目的基因全长,回收特异条带(图1B, 1 500 bp左右)。测序结果表明,该cDNA全长1 453 bp,编码413个氨基酸残基的多肽。在NCBI上Blast比对分析,与火炬松等植物R2R3-MYB基因有较高的相似性,命名为ClMYB4。
 图1 ClMYB4基因的PCR产物 |
1.2 ClMYB4基因的生物信息学分析
ClMYB4基因编码一条392个氨基酸残基的多肽,理论分子量为44.5 KDa,等电点为5.72。基于BioEdit软件的Pairwise alignment (ClustalW BLOSUM62)功能分析表明,ClMYB4蛋白与PtMYB4蛋白序列的一致性为40.5%,相似性为47.66%。但在MYB蛋白的DNA结合结构域(DNA Binding Domain, DBD)序列上,它们的一致性和相似性均为88.03%,ClMYB4与被子植物拟南芥MYB46的序列相似性也高达79.66%。利用ClustalX比对分析ClMYB4与火炬松PtMYB1、PtMYB4,白云杉PgMYB1、PgMYB2、PgMYB4和拟南芥AtMYB46、AtMYB58、AtMYB63蛋白发现,它们在蛋白N端的MYB-DBD序列上高度保守,均具有典型的【W-(X19)-W(X19)-W…F/I-(X18)-W-(X19)-W】氨基酸基序(Dubos, 2010),如图2A。MEME在线分析这些蛋白发现了两个保守的氨基酸基序,它们代表着MYB基因的R2和R3重复序列,在各个物种中具有高度保守的氨基酸基序,如图2B,说明我们克隆的ClMYB4基因具有典型的R2R3结构域氨基酸基序。
 图2 ClMYB4是典型的R2R3-MYB基因 |
用SWISS-MODEL对ClMYB4蛋白的三级结构进行同源建模,见图3。结果表明在其MYB结构域区域构成两个螺旋-转角-螺旋(HTH)结构,如箭头所示,分别在空间上形成两个环状的沟槽,为参与DNA识别及结合的部位;ClMYB4蛋白与AtMYB46和PtMYB4蛋白空间结构的比较发现,这三个蛋白在N端DNA结合结构域上的空间结构基本一致,只是参与的氨基酸数量不同,分别为103、112和107个氨基酸残基。与同源建模的模板1H8CC相比缺少R1-repeat,因此这些蛋白都少一个与DNA结合的空间结构,说明ClMYB4蛋白是典型的R2R3-MYB转录因子。
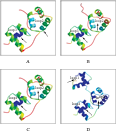 图3 ClMYB4蛋白的三维空间结构预测及与AtMYB46和PtMYB46蛋白构象比较 |
利用MEGA 5.0软件的Neighbor-Joining法,构建了基于蛋白氨基酸序列的系统进化树,如图4。所研究的蛋白序列一部分为裸子植物火炬松和白云杉的R2R3-MYB基因,一部分为桉树和拟南芥中与次生壁和木质素合成转录调控相关的MYB基因。结果显示,ClMYB4与白云杉PgMYB4,火炬松PtMYB4,桉树EgMYB2和拟南芥AtMYB46聚为一个亚组,拟南芥AtMYB46已被证实是维管发育决定基因SND1 (Secondary cell wall related NAC Domain1)的直接下游靶基因(Zhong et al., 2007),负责次生壁合成过程的转录调控。其他裸子植物MYB基因中,PgMYB2与PtMYB2、PtMYB3与PtMYB7、PtMYB1、PtMYB8和PgMYB1各自聚为一个小类,说明ClMYB4与白云杉PgMYB4,火炬松PtMYB4的亲缘关系最近。此外,拟南芥AtMYB58和AtMYB63单独聚为一类,而桉树EgMYB2和火炬松PtMYB14聚为一类。
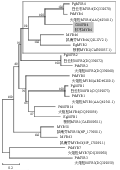 图4基于氨基酸序列构建的ClMYB4系统发育树 |
1.3杉木MYB基因的原核表达条件优化
将构建正确的原核表达载体质粒pET-30b-ClMYB4转化大肠杆菌Rosetta菌株。挑选阳性克隆子在37℃条件下采用IPTG浓度0.01 mmol/L,0.1 mmol/L,1 mmol/L诱导后,SDS-PAGE电泳显示目的蛋白ClMYB4均可以表达,但是ClMYB4蛋白(70 kD, 如图5)的大小与预测蛋白分子量大小(54 kD)不太一致。不同的IPTG对重组蛋白表达的诱导影响不明显,0.1 mmol/L,1 mmol/L IPTG诱导的效果较好,因此,我们选择0.1 mmol/L IPTG作为大量诱导重组蛋白表达的使用浓度。
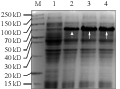 图5 pET-30b-ClMYB4 (Rosetta)诱导条件优化电泳图 |
1.4杉木MYB基因原核表达蛋白的纯化
分析发现ClMYB4重组蛋白不是以可溶性蛋白形式存在,而是主要存在于大肠杆菌的包涵体中,包涵体蛋白不溶于水,从包涵体中纯化重组蛋白,需要先将沉淀蛋白溶解。重组蛋白ClMYB4的包涵体沉淀分别用5%的Triton和不同浓度的尿素溶液洗涤,洗涤液经SDS-PAGE电泳检测溶解效果。结果如图6所示,0.5%的Triton不能溶解包涵体蛋白,包涵体蛋白在4 mol/L或8 mol/L的尿素溶液中溶解度较高,可以得到较多的ClMYB4重组蛋白,我们选用8 mol/L尿素溶液为Rosetta-ClMYB4菌株包涵体的最佳溶解洗涤溶液。
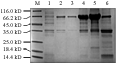 图6 ClMYB4蛋白包涵体尿素洗涤SDS-PAGE电泳图 |
使用Ni-Agarose His标签蛋白纯化试剂盒纯化经过8 mol/L尿素溶液溶解的ClMYB4重组蛋白,该镍柱纯化系统对6×His-tag蛋白具有显著特异吸附能力,能够高效一步纯化带有6个组氨酸亲和标签的蛋白。本实验所得重组蛋白在两端都标记有6×His-tag,所以可以直接利用此方法纯化。将8 mol/L尿素洗涤液过Ni柱吸附ClMYB4重组蛋白,再用不同浓度的咪唑缓冲液洗脱纯化,SDS-PAGE电泳检测回收液。电泳结果显示(如图7),咪唑缓冲液浓度越高,得到的重组蛋白ClMYB4浓度和纯度越好,其中500 mmol/L咪唑洗脱液洗脱纯化的重组蛋白的纯度最高。
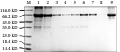 图7 ClMYB4纯化过程SDS-PAGE电泳图 |
2讨论
MYB类转录因子家族是一类含有一个或多个MYB结构域(MYB-repeat)的转录因子(Bedon et al., 2007),其高度保守的DNA结合结构域基序为我们分离克隆和鉴别MYB家族成员提供理论基础。本实验克隆的杉木ClMYB4蛋白具有两个保守的MYB结构域,序列及蛋白结构分析发现它们分别属于典型的R2和R3结构域,可以独立形成螺旋-转角-螺旋二级结构,构成两个与DNA结合的三维空间构象。同时在氨基酸水平上,这段保守序列中每隔约18个氨基酸残基便规则的出现色氨酸残基(W),它们构成MYB蛋白的疏水中心。比较杉木ClMYB4蛋白与松科植物和被子植物R2R3-MYB蛋白的DBD结构域发现,它们在氨基酸序列上保守性均为80%左右,而且在核心氨基酸(如色氨酸)的数量和位置上也高度保守,说明MYB基因的DNA结构域在物种之间是高度保守的。
虽然R2R3-MYB在N端的DNA结合结构域的氨基酸基序高度保守,但是与MYB基因的转录激活作用相关的C端氨基酸基序的变异率很高,保守性极低(Stracke et al., 2001; Wilkins et al., 2009),这些特点与MYB基因家族在真核生物发挥着重要且广泛的调节作用相关(Bedon et al., 2007)。植物中的R2R3-MYB参与了植物初生和次生代谢过程,细胞形态建成,发育过程及各种应激反应(Stracke et al., 2001)。所以,分析杉木ClMYB4蛋白与其它蛋白的进化关系有助于我们了解其生物学功能。根据编码蛋白全长序列的聚类分析,我们发现ClMYB4蛋白与其它参与植物次生壁加厚过程转录调控的R2R3-MYB蛋白聚为一个亚族。2003年,Patzlaff等首次发现火炬松PtMYB4的异源表达会导致木质素在转基因烟草植株的异位沉积,证明PtMYB4基因在火炬松木材发育过程发挥重要作用。拟南芥AtMYB46被证实是其次生壁加厚过程的重要调控基因,不但木质素会在过量表达植株中发生异位沉积,同时导致某些细胞发生不正常的次生壁加厚现象(Zhong et al., 2007)。AtMYB46和PtMYB4在桉树中的同源基因EgMYB2也具有相似的生物学功能(Goicoechea et al., 2005)。本研究发现ClMYB4与以上基因的系统发生关系最近,杉木中的ClMYB4是与他们一类的同源基因。因此笔者认为ClMYB4基因同样可能在杉木木材发育过程中发挥重要的调控作用。
以往的研究还发现,一些次生壁合成基因和木质素合成功能酶基因的顺式调控元件AC序列(AC Ⅰ:ACCTACC)是其转录调控的分子基础(Hatton et al., 1995; Lacombe et al., 2000; Zhong et al., 2010)。参与次生壁合成调控的AtMYB46,PtMYB4和EgMYB2蛋白,以及木质素合成调控转录因子AtMYB58/AtMYB63,PtMYB1等蛋白均可以在体外与AC序列结合。本实验在原核表达菌株Rosetta中系统优化表达了杉木ClMYB4蛋白,进一步根据重组蛋白的His标签纯化获得了ClMYB4重组蛋白。一方面,可以通过EMSA (Electrophoretic Mobility Shift Assay,电泳迁移率实验)分析ClMYB4重组蛋白是否可以通过与AC序列相互作用来调控杉木木质素合成基因的转录;另一方面可以结合其它分子生物学方法如染色质免疫共沉淀等方法(O'Neill et al., 2006)研究ClMYB4基因如何调控木质素之外的其它次生壁组分生物合成的分子机理。迄今为止,木质素合成过程的功能酶遗传改良已在多种林木品种中开展了研究(章霄云等, 2006; 李桢等, 2009),但是在转录调控水平上改良木质素和次生壁合成过程的研究没有报道,本研究克隆的ClMYB4基因为杉木在转录调控水平遗传改良木材品质奠定了理论基础。
3材料与方法
3.1材料
3.1.1实验材料及RNA提取
由福建洋口林场引进的杉木6421无性系的嫁接植株,于2008年春定植于南京林业大学苗圃。实验剪取两年生枝条的茎段,液氮速冻用于RNA提取。CTAB法提取总RNA,具体方法参考Chang等(1993),杉木发育木质部转录组数据库由本项目组保存。
3.1.2菌株及质粒
大肠杆菌菌株(E. coli) JM109由本实验室保存;Rosetta购自康为世纪生物公司;克隆载体pMD19-T购自TaKaRa公司;质粒pET-30b (+)由中国林科院生物所馈赠。
3.1.3工具酶及试剂
限制性内切酶Hind Ⅲ,BamH Ⅰ、Taq DNA 聚合酶,高保真酶,T4 DNA 连接酶、100 bp和DL2000 DNA ladder Marker购买于TaKaRa公司; RACE实验使用SMARTerTM RACE cDNA Amplication Kit (Clontech公司);DNA纯化回收试剂盒和质粒DNA提取试剂盒购于爱思进生物技术有限公司; Ni-Agarose His 标签蛋白纯化试剂盒(Cat. No. CW0893, 康为世纪);其它生化试剂均为国产分析纯。
3.2方法
3.2.1 ClMYB4全长cDNA的克隆
根据AtMYB4蛋白序列在杉木发育木质部转录组数据库中(本项目组保存)做本地Blast分析,查找相似性最高的contigs。根据得分最高的序列设计引物,以杉木“6421”无性系两年生茎段总RNA反转录得到的cDNA为模板做RACE反应,获取5'未知序列。经拼接后重新设计引物获得ClMYB4基因的全长cDNA,克隆至载体pMD19-T,转化大肠杆菌JM109感受态,挑选阳性克隆子测序鉴定。
引物序列:MYB4-5R: 5'-GAACGAGCAGGCGTGGCAGT-3' (RACE);MYB4-F: 5'-GCCACCCTCTTGTGAGGAAACG-3';MYB4-R: 5'-AATAACTTGTCACGCAGAAAAACTA-3'
3.2.2 ClMYB4预测蛋白的生物信息学分析
利用DNAMAN分析ClMYB4基因的cDNA序列,分析完全开放阅读框。利用ClustalX进行基于氨基酸序列的多重序列比对,BioEdit软件分析与其它物种MYB蛋白在氨基酸序列上的一致性和相似性。MEME在线分析ClMYB4的MYB结构域保守氨基酸特点。SWISS-MODEL在线构建ClMYB4的三级空间结构(Arnold et al., 2006),并与PtMYB4和AtMYB46蛋白的进空间结构分析比较。MEGA 5.0构建基于蛋白序列的基因进化树。
3.2.3 ClMYB4蛋白原核表达载体的构建
根据原核表达载体pET-30b (+)的MCS位点及翻译起始位点特征,设计带有限制性内切酶Hind Ⅲ,BamH Ⅰ酶切位点的引物,扩增ClMYB4的蛋白编码序列,不包含内含子以保证与His6-tag的正常衔接而获得His标签的重组蛋白。经双酶切连接反应后转化大肠杆菌JM109,筛选阳性克隆子测序鉴定重组质粒的正确性。
引物序列:MYB4-YF: 5'-CGGGATCCATGAGCTCCACAGATCAACC-3';MYB4-YR: 5'-CCCAAGCTTGGTGAAATACATTTGATCGA-3'
3.2.4 ClMYB4重组蛋白的原核表达与优化
提取构建正确的pET-30b-ClMYB4质粒,转化原核表达宿主菌Rosetta。将鉴定后的表达菌株接种于LB培养基(含有100 ug/ml Km)中,37℃振荡培养、过夜活化。次日,按1%的比例转接于新鲜LB培养基(含有100 ug/ml Km)中,37℃、250 rpm 振荡培养至OD600约为0.6 h,加入终浓度分别为0 mmol/L、0.01 mmol/L、0.1 mmol/L和1 mmol/L的IPTG,37℃、250 rpm 诱导4 h。8 000 rpm离心收集1 ml菌液,弃上清,加入40 u1 1×PBS振荡悬浮菌体,加入10 u1的5×SDS上样缓冲液,混匀后100℃煮沸10 min,12 000 rpm离心5 min,取其上清液进行SDS-PAGE电泳检测(李晓薇等, 2011)。
3.2.5 ClMYB4重组蛋白的纯化
将检测阳性表达的表达菌株Rosetta (pET-30b-ClMYB4)接种于1.2 L含有100 ug/ml卡那霉素的新鲜LB培养基中,37℃、250 rpm 振荡培养;至OD600约为0.6 h,加入一定量的IPTG至终浓度0.1 mmol/L,37℃、250 rpm 诱导培养4 h。离心收集诱导后菌体,用100 ml 1×PBS重悬,超声破碎,分别收集上清与沉淀,用0.5% Triton和1 mol/L、2 mol/L、4 mol/L、8 mol/L尿素(pH8.0)洗涤包涵体沉淀,SDS-PAGE电泳分别检测上清与沉淀中是否包含ClMYB4重组蛋白。
重组蛋白的纯化过程:根据优化的条件大量表达ClMYB4重组蛋白,然后根据以上电泳检测的结果将最优的尿素洗涤液,过Ni柱进行ClMYB4重组蛋白纯化。结合完毕后,分别用5, 20, 50, 100, 250, 500 mmol/L咪唑进行洗脱,收集洗脱液,进行SDS-PAGE电泳检测,详细步骤按Ni-Agarose His标签蛋白纯化试剂盒使用说明(康为世纪)。
作者贡献
吕运舟,郑佳是本研究的实验设计和实验研究的执行人并完成数据分析,论文初稿的写作;陈金慧参与实验设计,试验结果分析;施季森是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究是在国家自然基金重点项目(30930077)资助下完成;原核表达载体pET-30b (+)由中国林科院生物所馈赠,在此一并表示感谢。
参考文献
Arnold K., Bordoli L., Kopp J., and Schwede T., 2006, The SWISS-MODEL workspace: a web-based environment for protein structure homology modeling, Bioinformatics, 22(2): 195-201
http://dx.doi.org/10.1093/bioinformatics/bti770 PMid:16301204
Bedon F., Grima-Pettenati J., and Mackay J., 2007, Conifer R2R3-MYB transcription factors: sequence analyses and gene expression in wood-forming tissues of white spruce (Picea glauca), BMC plant biol., 7: 17
http://dx.doi.org/10.1186/1471-2229-7-17 PMid:17397551 PMCid:1851958
Bomal C, Bedon F., Caron S., Mansfield S.D., Levasseur C., Cooke E.K.J., Blais S., Tremblay L., Morency M-J., Pavy N., Grima-Pettenati J., Séguin A., and MacKay J., 2008, Involvement of Pinus taeda MYB1 and MYB8 in phenylpropanoid metabolism and secondary cell wall biogenesis: a comparative in planta analysis, J. Exp. Bot., 59(14): 925–939
http://dx.doi.org/10.1093/jxb/ern234 PMid:18805909 PMCid:2576632
Chang S., Puryear J., and Cairney J., 1993, A simple and efficient method for isolating RNA from pine trees, Plant Mol. Biol. Rep., 11(2): 113-116
http://dx.doi.org/10.1007/BF02670468
Dubos C., Stracke R., Grotewold E., Weisshaar B., Martin C., and Lepiniec L., 2010, MYB transcription factors in Arabidopsis, Trends Plant Sci., 15(10): 573-581
http://dx.doi.org/10.1016/j.tplants.2010.06.005 PMid:20674465
Goicoechea M., Lacombe E., Legay S., Mihaljevic S., Rech P., Jauneau A., Lapierre C., Pollet B., Verhaegen D., Chaubet-Gigot N., and Grima-Pettenati J., 2005, EgMYB2, a new transcriptional activator from Eucalyptus xylem, regulates secondary cell wall formation and lignin biosynthesis, Plant J., 43(3): 553–567
http://dx.doi.org/10.1111/j.1365-313X.2005.02480.x PMid:16098109
Hatton D, Sablowski R., Yung M-H., Simth C., Schuch W., and Bevan M., 1995, Two classes of cis sequences contribute to tissue-specific expression of a PAL2 promoter in transgenic tobacco, Plant J., 7(8): 859-876
http://dx.doi.org/10.1046/j.1365-313X.1995.07060859.x PMid:7599647
Hertzberg M., Aspeborg H., Schrader J., Andersson A., Erlandsson R., Blomqvist K., Bhalerao R., Uhlén M., Teeri T.T., Lundeberg J., Sundberg B., Nilsson P., and Sandberg G., 2001, A transcriptional roadmap to wood formation, Proc Natl. Acad. Sci. USA, 98(25): 14732-14737
http://dx.doi.org/10.1073/pnas.261293398 PMid:11724959 PMCid:64750
Lacombe E., Doorsselaere J.V., Boerjan W., Boudet A.M., and Grima-Pettenati J., 2000, Characterization of cis-elements required for vascular expression of the Cinnamoyl CoA Reductase gene and for protein-DNA complex formation, Plant J., 23(5): 663-760
http://dx.doi.org/10.1046/j.1365-313x.2000.00838.x PMid:10972892
Li X.W., Su L.T., Zhao X., Zhai Y., Zhang H.J., Zhang Q.L., Li J.W., and Wang Q.Y., 2011, High expression of two soybean MYB transcription factor in Escherichia coli, Shengwu Jishu Tongbao (Biotechnology Bulletin), 8: 99-114 (李晓薇, 苏连泰, 赵旭, 翟莹, 张海军, 张庆林, 李景文, 王庆钰, 2011, 两个大豆MYB转录因子在原核细胞中的高效表达, 生物技术通报, 8: 99-114)
Li Z., Wang H.Z., Li R.F., and Wei J.H., 2009, Lignin biosynthesis and manipulation in plants and utilization of biomass energy, Zhiwu Xubao (Chinese Bulletin of Botany), 44(3): 262-272 (李桢, 王宏芝, 李瑞芬, 魏建华, 2009, 植物木质素合成调控与生物质能源利用, 植物学报, 44(3): 262-272)
McCarthy R.L., Zhong R., Fowler S., Lyskowski D., Piyasena H., Carleton K., Spicer C., and Ye Z.H., 2010, The poplar MYB transcription factors, PtrMYB3 and PtrMYB20, are involved in the regulation of secondary wall biosynthesis, Plant cell phsiol., 51(6): 1084-1089
Ohashi-Ito K., and Fukuda H., 2010, Transcriptional regulation of vascular cell fates, Curr. Opin. Plant Biol., 13(6): 1-7
http://dx.doi.org/10.1016/j.pbi.2010.08.011 PMid:20869293
O'Neill L.P., VerMilyea M.D., and Turner B.M., 2006, Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations, Nature genetics, 38(7): 835-841
http://dx.doi.org/10.1038/ng1820 PMid:16767102
Patzlaff A., Newman L.J., Dubos C., Whetten R.W., Newman L.J., McInnis S., Bevan M.W., Sederoff R.R., and Campbell M.M., 2003a, Characterization of PtMYB1, an R2R3-MYB from pine xylem, Plant Mol. Biol., 53(4): 597–608
http://dx.doi.org/10.1023/B:PLAN.0000019066.07933.d6 PMid:15010621
Patzlaff A., McInnis S., Courtenay A., Surman C., Newman L.J., Smith C., Bevan M.W., Mansfiled S., Whetten R.W., Sederoff R.R., and Campbell M.M., 2003b, Characterization of a pine MYB that regulates lignification, Plant J., 36(6): 743–754
http://dx.doi.org/10.1046/j.1365-313X.2003.01916.x PMid:14675440
Stracke R., Mand W., and Weisshaar B., 2001, The R2R3-MYB gene family in Arabidopsis thaliana, Curr. Opin. Plant Biol., 4(5): 447-456
http://dx.doi.org/10.1016/S1369-5266(00)00199-0
Wilkins O., Nahal H., Foong J., Provart N.J., and Campbell M.M., 2009, Expansion and diversification of the populus R2R3-MYB family of transcription factors, Plant Physiol., 149(2): 981–993
http://dx.doi.org/10.1104/pp.108.132795 PMid:19091872 PMCid:2633813
Zhang X.Y., Guo A.P., He L.K., and Kong H., 2006, Advances in study of lignin biosynthesis and genetic engineering manipulation, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 4(3): 431-437 (章霄云, 郭安平, 贺立卡, 孔华, 2006, 木质素生物合成及其基因调控的研究进展, 分子植物育种, 4(3): 431-437)
Zhong R., Richardson E.A., and Ye Z.H., 2007, The MYB46 transcription factor is a direct target of SND1 and regulates secondary wall biosynthesis in Arabidopsis, Plant Cell, 19(9): 2776-2792
http://dx.doi.org/10.1105/tpc.107.053678 PMid:17890373 PMCid:2048704
Zhong R., and Ye Z.H., 2009, Transcriptional regulation of lignin biosynthesis, Plant Signal. Behav., 4(11): 1028-1034
http://dx.doi.org/10.4161/psb.4.11.9875
Zhong R., Lee C., and Ye Z.H., 2010, Evolutionary conservation of the transcriptional network regulating secondary cell wall biosynthesis, Trends Plant Sci., 15(11): 625-632
http://dx.doi.org/10.1016/j.tplants.2010.08.007 PMid:20833576
Zhou J., Lee C, Zhong R., and Ye Z.H., 2009, MYB58 and MYB63 are transcriptional activators of the lignin biosynthetic pathway during secondary cell wall formation in Arabidopsis, Plant Cell, 21(1): 248-266
http://dx.doi.org/10.1105/tpc.108.063321 PMid:19122102 PMCid:2648072