作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2010 年, 第 8 卷, 第 9 篇 doi: 10.5376/mpb.cn.2010.08.0009
收稿日期: 2010年09月21日 接受日期: 2010年10月24日 发表日期: 2010年11月03日
马振等, 2010, 水稻中花11号Tos17插入突变体库的创制, 分子植物育种 Vol.8 No.9 (doi: 10.5376/mpb.cn.2010.08.0009)
基因标签技术是植物功能基因组研究的一种重要策略。本研究利用水稻反转录转座子Tos17创建了水稻品种中花11号的突变体库,该突变体库包含15,543个独立再生株系。Southern杂交证明中花11号中含有4个Tos17原始拷贝,经过3个月的组织培养,平均每个再生植株中含有1.4个新的转座拷贝,据此推测本突变体库中含有约21 760个新的Tos17插入位点。利用接头PCR技术分离到Tos17转座位点侧翼水稻基因组序列524条,大约310个功能基因带有标签。侧翼序列的分析结果表明, Tos17显著偏爱于插入基因区,而显著不偏爱于插入基因间隔区和转座因子相关区。在基因编码区内,Tos17显著偏爱插入基因的外显子区域。对Tos17插入突变体T1代1 881个家系进行了突变表型的筛选,结果显示约97个(5.15%)家系表现出明显的突变表型,其中以植株矮化、不育的突变体占多数。本研究证明Tos17突变体库是水稻功能基因组研究的有用资源。
水稻是重要的粮食作物,由于其基因组小、精确的全基因组序列及高效的遗传转化体系,已成为作物功能基因组研究的模式植物。在功能基因的研究中,突变体对于基因的鉴定和阐释发挥着重要的作用。然而,基因自然突变的频率很低,人为创建大型突变体库已成为植物功能基因组研究的重要平台。目前构建植物突变体的方法主要可分为物理方法、化学方法和利用基因标签技术如农杆菌T-DNA插入、转座子插入等方法。以快中子为代表的物理方法和双环氧丁二烯(DEB)﹑甲基磺酸乙酯(EMS)为代表的化学诱变的方法虽然可以很快的得到突变体,但是突变体的表型和被突变的基因之间的联系很难找到,只能通过图位克隆的方法来分离基因。因此,目前构建突变体库最有效的方法是利用T-DNA或转座子等插入元件来构建突变体库。由于插入元件序列已知,鉴定插入位点基因组序列容易,因此突变表型和被突变的基因的之间的联系很容易建立,已成为植物功能基因分离鉴定的重要策略之一 (Krishnan et al., 2009)。在近十年内,各国科学家在水稻中利用T-DNA或转座子已经创建了大量的突变体(Martienssen 1998; Jeon et al., 2000; Wu et al., 2003; Hirochika et al., 2004)。虽然已经创建了大量的突变体,然而只有50%的水稻基因被“标记”,因此还需要利用各种不同的方法创建插入标签,使每一个基因都能被“标记”(Krishnan et al., 2009)。
Tos17是水稻中的一个内源的复制型反转录转座子,全长4 114 bp,在栽培稻中含有1-5个拷贝(Cheng et al., 2006),Tos17已被用于创建水稻转座子插入突变体库。Hirochika等在1996年发现了15个水稻中的反转录转座子,其中Tos10,Tos17,Tos19在组培的条件下被激活。后经过大量研究后发现Tos17具有以下优点:(1) Tos17在组培的条件下激活,分化成植株后失活,因此Tos17插入引起的突变可以稳定遗传;(2) Tos17的拷贝数随着组培的时间而增多,在5个月的组培时间下,可平均产生10个拷贝,可以通过控制组织培养时间来控制转座的拷贝数;(3) Tos17突变体的产生不涉及转基因过程,因此在一些转基因控制严格的国家也可以使用。(4) Tos17的转座是随机的并能覆盖整个基因组(Miyao et al., 2003; Piffanelli et al., 2007)。Miyao等(2007)构建了超过50,000个独立的再生植株,大约含有500,000个插入位点并对突变产生的表型进行了分类。并且分离克隆一些重要基因例如控制同源染色体配对的基因Pair1 (Nonomura et al., 2004a)和Pair2 (Nonomura et al., 2004b)。
中花11号是一个典型的粳稻品种,由于其遗传转化效率高、栽种地区适应性强,已成为我国水稻功能基因组研究的模式品种之一。本实验室利用水稻品种中花11号创建了超过120 000独立的T-DNA插入突变体(Wu et al., 2003; http://rmd.ncpgr.cn/)。农杆菌介导的T-DNA转化经历了组培过程,研究发现有Tos17 在转化体中有新的转座(Long et al., unpublished)。然而,由于不同批次的转化植株组培时间不一样,Tos17在有些批次的材料中没有转座或转座的位点较少,难以采用高通量的方法批量鉴定我室T-DNA库中Tos17的插入位点。同时为了丰富水稻的突变体资源,实现水稻全基因组饱和突变体的目标,本研究利用水稻中花11号为材料,创建了水稻Tos17突变体库。本研究共创建独立的水稻Tos17突变体15 543株,分离非重复插入位点侧翼序列524条,并对T1代1,881个家系进行了表型筛选。本研究为丰富了水稻突变体资源、发掘分离水稻重要功能基因奠定了基础。
1. 结果与分析
1.1 组织培养时间决定Tos17转座的拷贝数
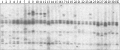
由于不同水稻品种中Tos17的拷贝数不一致,且组织培养的时间导致Tos17新的插入拷贝不一样。为了研究中花11号及不同组织培养期限后Tos17的拷贝数,我们采用Southern杂交的方法,利用Tos17的特异探针检测了不同样本中Tos17的拷贝数。如图1所示,野生型中花11号中含有4个拷贝,愈伤组织继代1个月后分化出的植株中能检测到Tos17的新拷贝,说明Tos17的转座在组织培养的初期就有可能发生转座。在愈伤组织继代培养2个月的情况下,愈伤组织分化出的植株中几乎都含有1个Tos17的新拷贝。在继代3个月的愈伤分化出的每个植株中都可以检测到Tos17新拷贝,新拷贝数为1-3个。随着愈伤组织培养时间的延长,分化出的植株的中的新拷贝数增加。考虑到要保证植株中都含有至少一个Tos17的新拷贝,而且较少的拷贝数有利于对突变体与插入标签的共分离,本研究确定3个月的愈伤组织继代的再生植株来大规模构建的Tos17插入突变体库。
|
图1 愈伤组织不同继代时间的再生植株中Tos17的拷贝数
Figure 1 The copy number of Tos17 in the regenerated rice plant underwent different calli culture time |
1.2 创建Tos17转座突变体库
将中花11号成熟种子放于N6培养基上,经过大约45天左右产生胚性愈伤组织,然后挑选结构紧实、颜色鲜黄的胚性愈伤组织转移到继代培养基上继代3个月,以激活Tos17的转座,愈伤组织诱导、继代、分化的过程如图2所示。为了避免来自同一愈伤组织分化出的植株含有相同的插入位点,愈伤组织从诱导、继代到分化,每一颗愈伤都是独立培养,每颗愈伤上分化出的植株只取一株。我们大约诱导了超过30 000粒中花11号种子,产生出大约22 000颗愈伤,最后获得独立的水稻中花11号Tos17突变体15 543株。
|
图2 Tos17突变体的创建过程
Figure 2 The process of generation Tos17 insertioanl lines |
1.3 突变体库中Tos17插入位点数分析
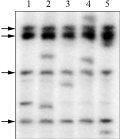
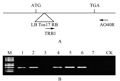
为了评估插入突变体库中Tos17转座的位点数,随机从突变体库中选取了T0代再生植株200份,采用Southern杂交的方法检测再生植株中Tos17的拷贝数。部分植株的Southern杂交结果如图3所示,箭头所示的泳带为中花11号的原始拷贝,检测的再生植株中均含有新的Tos17转座拷贝。统计检测的200份样品中总共含有Tos17 新拷贝280个,平均每个T0代再生植株中含有1.4个新的Tos17插入拷贝,据此推测本突变体库中含有的新的Tos17拷贝数约为21 760个。
|
图3 突变体库中Tos17插入拷贝数检测
Figure 3 The examination of Tos17 copy number in the regeneration plants |
1.4侧翼序列的分离
接头PCR(Adapter-PCR)的一种非常有效的分离侧翼序列的方法(Piffanelli et al., 2007),通过设计合适的限制性内切酶,连上特异接头,通过两步巢式PCR反应,可以获得60%以上植株的Tos17插入位点的侧翼序列(图4)。由于我们所用的Tos17左右端特异引物据左右边界都超过200 bp,因此Adapter-PCR产物经1%TBE凝胶电泳后,回收片段大小在200 bp以上的片段送往上海国家基因研究中心测序,小于200 bp的片段由于序列太短没有被回收。
|
图4 Tos17侧翼基因组序列的扩增
Figure 4 Amplification of the flanking sequence of Tos17 in regenerated plants |
1.5 侧翼序列分析
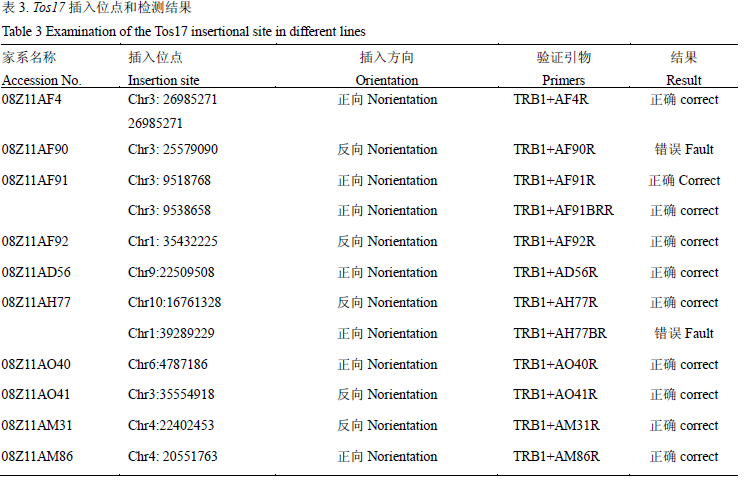
根据TIGR的数据我们将水稻基因组简单的划分为三个区域:基因区(Genic region),基因间隔区(Intergenic region),转座因子相关区(TE-related region)。依据对基因功能产生影响的直接区域,将基因区界定为基因编码区,ATG上游1 kb和翻译终止密码子下游500 bp(Zhang et al., 2007)。卡方检测结果证明Tos17在基因区、基因间隔区和转座子相关区的分布是不均匀的(c2=210, P<0.005)。从SR值的计算结果来看,Tos17在基因区内的插入数目要显著高于期望值(SR=10.85),而在转座因子相关区(-3.68)和基因间隔区(-8.88)要显著少于期望值(表3)。我们初步认为Tos17极度偏向插入基因的编码区而不偏向于插入基因的间隔区和转座因子相关区。
我们将基因区初步划分为编码区,ATG上游1 kb区和翻译终止密码子下游500 bp。同样通过卡方检测证明Tos17的插入具有显著的偏爱性(c2=52.58, P<0.005)。SR值证明Tos17在基因编码区的插入要显著的高于期望值,而在ATG上游1 kb区和翻译终止密码子下游500 bp这两个区内要显著少于期望值,初步证明Tos17偏爱插入基因区(SR=4.55)而不偏向插入ATG上游1kb区(SR=-4.62)和翻译终止密码子下游500 bp (SR=-3.25)这两个区。而基因的编码区又可以细分为外显子(Exon)和内含子(Intron)这两个区域。卡方测验(c2=11.4, P<0.005)证明Tos17插入数目在编码区内的分布也不是一个平均的状态,而且存在显著差异。通过SR分析发现Tos17在外显子内的插入数目要显著高于期望值(SR=2.48),而在内含子内的插入虽然少于期望值但是没有达到显著水平(SR=-2.29) (表1)。因此我们初步认为Tos17在基因编码区内显著偏爱插入基因的外显子区域。
.png) 表1 524条Tos17标签在水稻基因组中各区间内的分布(以SR值>2.33为显著水平) Table 1 Distribution of the 524 Tos17 insertions in different regions of the rice genome. |
1.6 Tos17插入突变体的筛选
为了检测Tos17插入导致植株突变的效率,我们对1,881个T1代家系进行了突变表型的观察,每一个家系至少种植20株,理论上二倍体物种中如果一个染色体上含有一个Tos17插入拷贝,根据孟德尔遗传分离比后代应该出现1:2:1的基因型分离,纯合突变体应该占到观察植株数的1/4。在水稻正常栽培条件下,我们在苗期、分蘖期、抽穗期、成熟期考察了植株产生的变异,其中对育性、分蘖数、抽穗期和穗型进行了重点观察。
在苗期出现的最多表型为白化和黄化,共观察到白化突变体家系12个,占突变家系的11.0%,突变频率为0.64%。出现黄化表型家系9个,占突变家系的8.26%,突变频率为0.48%。而移栽后,共计88个家系出现突变表型,突变频率为4.67%,其中最主要的突变表型为矮化,共观察到42个家系,占突变家系的38.53%,突变频率为2.23%。以晚抽穗15天为界限,共计观察到晚抽穗家系9个,占总突变家系的8.26%,突变频率为0.48%,其中一个家系“08Z11AD56”晚抽穗20天。苗期和移栽后共计观察到突变家系109个,突变频率为5.79%。所有突变类型及突变频率如表2所示,图5为部分典型突变体照片。
|
表2 Tos17突变表型和突变频率
Table 2 Summary of Tos17 insertional mutant phenotypes in T1 families |
|
图5 部分突变体表型
Figure 5 Examples of mutant phenotypes |
1.7 Tos17插入位点的验证
插入标签法分离克隆功能基因首先要证实插入标签与突变体表型共分离。由于Tos17插入位点的侧翼基因组序列是通过PCR扩增得到的,在批量化收集样品、回收扩增片段及引物特异性方面往往导致侧翼序列与水稻基因组序列匹配性地,少数序列与植株样品不对应。为了验证本研究获得的侧翼序列的正确性,我们通过设计一条和插入位点附近的基因组序列特异匹配的引物与Tos17边界的特异的引物匹配对样品进行PCR扩增,如果目的位点确实存在Tos17的插入,那么就会扩增出预期大小的目的条带(图6A),从而初步证实Tos17在该植株中的插入位点。
选取已有Tos17的侧翼序列的10个家系共12个插入位点进行了检测,其中有10的插入位点证实是正确的,Tos17在不同家系中的插入位点如表3所示。图6B为其中一个家系08Z11AO40 PCR扩增片段的电泳胶图。在08Z11AO40中,Tos17插在第6染色体的一个基因LOC_Os06g09430内,在插入位点Chr6: 4787186上下游各选取1 Kb,设计基因特异引物AO40R (5' -AGGAGGGCAGGAA GTGA -3'),和Tos17特异引物TRB1配对进行PCR扩增。在08Z11AO 40家系的T1代7个单株中,有5个单株扩增出了500 bp左右的预期条带,说明这5个单株在该位点含有Tos17标签;另外2株没有扩增出预期条带,说明它们是在T1代中分离出的该位点野生型基因型。
|
表3 Tos17插入位点和检测结果
Table 3 Examination of the Tos17 insertional site in different lines |
|
图6 Tos17在基因LOC_Os06g09430中的插入位点以及PCR验证结果
Figure 6 Verification of the insertion of Tos17 on the gene LOC_Os06g09430 |
2讨论
接头PCR由于涉及酶切连接的过程,因此对于模板DNA的质量有很高的要求,同时对于限制性内切酶的选择很重要。由于在水稻基因组中存在Tos17的原始拷贝,因此接头PCR的体系和设计需要满足不能将原始拷贝附近的基因组序列扩出的要求。对于水稻 品种“日本晴”,两个原始拷贝附近的基因组序列酶切位点分析表明,DraІ更适合分离Tos17侧翼序列,因为“日本晴”的中的两个Tos17原始拷贝的右端的DraІ酶切位点据这两个Tos17原始拷贝的右端边界的距离都在1.8 kb以上,而所用的接头PCR中进行巢式PCR扩增的反应程序很难将2 kb以上的片段扩出,这样分离出的Tos17侧翼序列即为新插入位点的侧翼序列。对于中花11号,经过摸索我们认为XbaІ和DraІ这两种酶比较适合用于进行接头PCR反应。同样我们还在据Tos17左边界200 bp左右设计了从Tos17左端进行巢式PCR扩增的Tos17特异引物,从而比较从不同方向分离Tos17侧翼序列的效率。实验证明,从Tos17右端分离侧翼序列PCR扩增效率可以达到60%以上,测序效率更是可以达到90%,而从左端分离Tos17侧翼序列往往存在原始拷贝的公共带,影响侧翼序列的分离效率。
我们总共获得到非重复的Tos17侧翼序列524条,通过分析发现Tos17的插入位点覆盖在水稻整个基因组上。就Tos17在基因区间的分布而言,Tos17倾向于插入基因区间内,不倾向于插入到基因间隔区和转座子区域,在基因内则比较倾向于插入基因的编码区,而在基因的编码区内则显著的偏爱于插入基因的外显子区域。这些特征和Tos17在“日本晴”中的报道相一致(Miyao et al., 2003; Piffanelli et al., 2007)。同时,这些这些特征在水稻T-DNA插入突变体库中也是类似的(Zhang et al., 2007)。
在2009年夏季对1 881个T1代家系进行了正向遗传学的筛选,希望从正向遗传学的途径筛选到与Tos17共分离的突变家系。从筛选的结果来看,田间最主要的突变表型是矮化,突变频率达到2.23%,占总突变家系的38.53%,其次是育性,不育突变频率达到1.17%,占总突变家系的20.56%。这和日本NIAS研究所的筛选结果基本一致,其筛选的M2代家系中主要的表型也是矮化和不育(Miyao et al.,2007)。 而且从田间的表型来看,突变往往不是只有一个性状,存在两个或三个突变性状同时发生的现象,例如矮化往往伴随不育或抽穗期的延迟,叶色变化与株高和穗部突变往往联系在一起,这说明被插入的基因可能具有一因多效或是有多个基因被同时敲除掉。从筛选的整个结果来看,1 881个家系中表型突变频率为5.69%。而NIAS研究所对其Tos17突变体进行了表型筛选,发现后代几乎一半的家系出现了至少一个突变表型。其突变频率要高于我们的观察结果。其原因可能是:NIAS研究所构建的Tos17突变体每株中Tos17的平均拷贝数达到10个(Miyao et al.,2007),远远高于本室Tos17突变体中的平均拷贝数。
Krysan等于1999年提出了的公式:P= 1- (1-x/g)n,已被广泛用于计算构建饱和突变体库所需的标签数目。公式中X代表目标作物基因的平均大小,G代表目标作物基因组的大小,n代表以一定概率P饱和基因组所需的标签数目。以水稻基因组大小为372,000 Kb,基因的平均长度为2.8 Kb来计算,预计是水稻每一个基因含有标签的概率99%需要得到561,677 条标签序列。目前在水稻中的突变体资源已经超过2 000 000,侧翼序列的数目也超过了200,000。那么由此产生了一个问题,到底要需要多少标签能够饱和掉水稻的基因组,即任何一个基因有99%的概率能至少找到一个标签。目前水稻中的突变体库资源中的插入位点已经超过2 000 000已经远远满足饱和水稻基因组的要求,但是水稻中侧翼序列的数目仅有200 000条左右,远远低于饱和水稻基因组的要求,因此目前比较迫切的任务是采用更有效的方法分离更多的侧翼序列。新一代测序技术的出现和发展可能会是给这一难题提供解决方法( Mardis, 2008)。随着测序成本的降低和测序效率的提升,未来希望能够通过深度测序或其他方法高效地鉴定标签的插入位点,实现高效剖析水稻全基因组中功能基因的目的。
3.材料和方法
3.1 Tos17突变体的产生
将脱壳的野生型中花11号种子,于75%酒精中消毒15min,10% HgCl2消毒30 min,转移到诱导培养基中。待30天后愈伤长出,为防止产生的后代Tos17突变体之间有相同的突变位点,所以每一颗种子诱导出的愈伤仅取一颗转移到继代N6培养基中(Wu et al., 2003),每30天更换培养基一次,继代培养3个月。将经过3个月继代培养的愈伤转移到分化培养基中,分化产生T0代突变植株。每一个愈伤上诱导出的植株只取一株,夏季种于武汉华中农业大学试验田。
3.2 T1代突变体的筛选
2009年4月在华中农业大学实验田播种Tos17突变体T1代家系1,883个,5月20日将每个家系随机取20株以16.5cm×26.4cm的密度移栽大田,于分苗期、分蘖期、抽穗期和成熟期四个时期观察表型。对家系内突变体表型一致的家系进行记载、拍照。
3.3 DNA抽提和接头PCR分离侧翼序列
取水稻新鲜幼嫩片,在-20℃预冷的研钵中磨至粉末状转入预冷的2 ml离心管中,加入约半管。每管加入600 μl 2% CTAB混合液(2% CTAB、1.4 mM NaCl、20 mM EDTA、100 mM Tris-HCl、15 μl RNA酶、0.2% β-巯基乙醇),上下颠倒摇匀,于65℃水浴锅中水浴15 min,11,000 rpm 离心5 min,吸取上清约500 μL。加入等体积的氯仿:异戊醇(24:1),上下颠倒轻摇5 min。11,000 rpm离心5 min,吸上清400 μL。加入异丙醇400 μL,上下颠倒轻摇沉淀DNA。离心5000 rpm,30 sec沉淀DNA,丢弃上清。加入500 μL75%乙醇,洗沉淀,离心10 sec,弃上清,自然风干,DNA沉淀溶于50 μL 无菌水中。
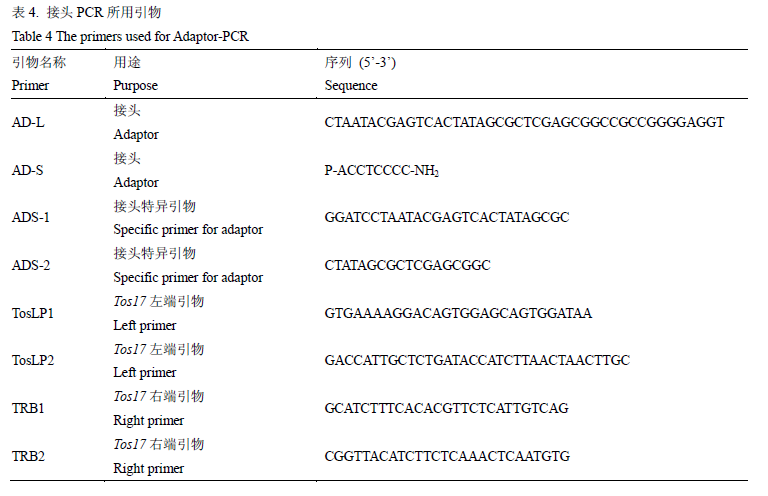
本研究采用接头PCR的方法分离侧翼序列,所用引物见表4。接头(AD)制备方法:等体积混合引物AD-L (100 mM)和引物AD-S (100 mM),最终反应浓度为50 mM,在95%水浴锅中水浴10 min,自然冷却至室温。具体的反应步骤和反应程序如下:
第一步:模板DNA的酶切和连接。体系:取用分离侧翼序列用CTAB法分离的模板DNA样品1 μL,接头(50 mM) 0.2 μL,10×M buffer 1 μL,10×T4 DNA连接酶buffer 1 μL,DraІ (15 U/μl) 0.15 μL, T4 DNA 连接酶(10 U/μL) 0.2 μL,补ddH2O水到10 μL。反应置于25℃(室温)反应约16-20h。
第二步:PCR反应第一步。第一步反应产物1 μL,10×PCR buffer 2 μL,2 mM dNTPs 1.8μL, 25 mM MgCl2 1.3 μL,ADS-1 (50 mM) 0.5 μL,3’端或5’端特异引物(TRB1或TosLP1),rTaq DNA polymerase(Takara 公司) 0.2 μL,补充ddH2O到20 μL。反应程序:94℃ 4 min;94℃ 30 sec,72℃ 3 min,7 cycles;94℃ 30sec,67℃ 3min,32cycles;67℃ 7min;25℃ 10sec。
第三步:PCR反应第二步。第一步PCR反应产物1 μL,10×PCR buffer 2 μL,2 mM dNTPs 1.8 μL,25 mM MgCl2 1.2 μL,ADS-2 (50 mM) 0.5 μL,3’端或5’端特异引物(TRB2或TosLP2),rTaq DNA polymerase(Takara 公司) 0.2 μL,补充ddH2O到20 μL。反应程序:94℃ 4 min; 94℃ 30 sec, 72℃ 3 min, 5 cycles; 94℃ 30 sec, 67℃ 3 min, 35 cycles; 67℃ 7 min; 25℃ 10 sec。
|
表4 接头PCR所用引物
Table 4 The primers used for Adaptor-PCR |
3.4 Southern杂交
抽提的水稻样品DNA 5 μg,采用限制性内切酶XbaІ完全酶切,用1%TAE凝胶电泳,然后转移到尼龙膜上(Zhang et al., 2007)。PCR反应扩增Tos17探针的引物为:TosL:5'-GCT ACCCGTTCTTG GACTAT-3';TosR:5'-CTGAAATCGGAGCACTGACA-3';探针合成PCR反应体系:野生型中花11号样品DNA 5 ng, 10×PCR Buffer 2 μL, 2 mmol/L dNTP 1.5 μL, 10 μmol/L引物0.3 μL, 10 U/μLTaq酶0.2 μL。PCR反应程序:94℃ 5 min; 94℃ 45 sec, 55℃ 45 sec, 72℃, 1 min, 30 cycles; 72℃, 7 min; 25℃ 30 sec。探针长度为600 bp。
3.5 T1代共分离检测
根据侧翼序列的信息在水稻生物信息网站(http://rice.plantbiology.msu.edu/)上检索,确定Tos17插入的位点,根据插入位点附近的侧翼序列信息,利用Premier 5设计引物,通过PCR反应验证插入位点是否正确。在插入位点两端设计特异引物,通过一条能和插入位点附近基因组特异匹配的引物以及一条能和Tos17特异匹配的引物配对扩增来确定插入位点是否正确。如果能配对扩增出特异性条带,则证明插入位点正确。PCR反应在ABI9700PCR仪(Applied Biosystem)上进行,反应体系如下:模板DNA 5 ng,10×PCR Buffer 2 μL, 2 mmol/L dNTP 1.5 μL, 10 μmol/L引物0.3 μL, 10 U/μl Taq酶0.2 μL。PCR反应程序:94℃ 5 min; 94℃ 45 sec, 55℃ 45 sec, 72℃1 min, 30 cycles; 72℃, 7 min; 25℃ 30 sec;Tos17插入位点两侧设计的PCR引物如表5所示。
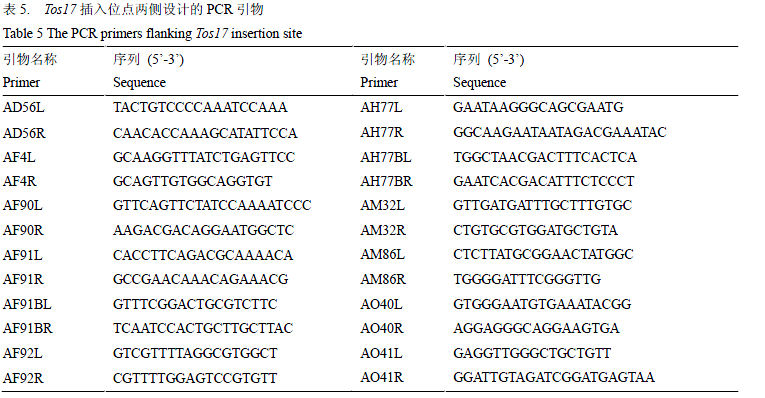 表5 Tos17插入位点两侧设计的PCR引物 Table 5 The PCR primers flanking Tos17 insertion site |
作者贡献
本项研究是在吴昌银的指导下完成的。马振参与了突变体的创建过程,完成数据分析,论文初稿的写作;突变的田间种植、收种主要由陈国兴完成;吴昌银设计并组织完成了该项研究,论文修改。全体作者都阅读并同意论文的文本投稿。
致谢
本研究由国家863计划项目(2006AA10A103)资助及国家转基因重大专项课题(2008ZX08010-002)资助。作者感谢李才顺、章成君、曹娟、陈玲、郜新强等人的帮助。
参考文献
Cheng C., Daigen M., and Hirochika H., 2006, Epigenetic regulation of the rice retrotransposon Tos17. Mol Genet Genomics, 276: 378-390 doi:10.1007/s00438-006-0141-9
Hirochika H., Guiderdoni E., An G., Hsing Y I., Eun M Y., Han C D., Upadhyaya N., Ramachandran S., Zhang Q., Pereira A., Sundaresan V., and Leung H., 2004, Rice mutant resources for gene discovery. Plant Mol Biol, 54:325-334 doi:10.1023/B:PLAN.0000036368.74758.66
Hirochika H., Sugimoto K., Otsuki Y., Tsugawa H., and Kanda M.,1996, Retrotransposons of rice involved in mutations induced by tissue culture. Proc Natl Acad Sci USA, 93: 7783-7788 doi:10.1073/pnas.93.15.7783
Jeon J S., Lee S., Jung K H., Jun S H., Jeong D H., Lee J., Kim C., Jang S., Yang K., Nam J., An K., Han M J., Sung R J., Choi H S., Yu J H., Choi J H., Cho S Y., Cha S S., Kim S I., and An G., 2000, T-DNA insertional mutagenesis for functional genomics in rice. Plant J, 22: 561-570 doi:10.1046/j.1365-313x.2000.00767.x
Krishnan A., Guiderdoni E., An G., Hsing Y I., Han C D., Lee M C., Yu S M., Upadhyaya N., Ramachandran S., Zhang Q., Sundaresan V., Hirochika H., Leung H., and Pereira A., 2009, Mutant resources in rice for functional genomics of the grasses. Plant Physiol, 149: 165-170 doi:10.1104/pp.108.128918
Krysan P J., Young J C., and Sussman M R., 1999, T-DNA as an insertional mutagen in Arabidopsis. Plant Cell, 11: 2283-2290
Mardis E R., 2008, The impact of next-generation sequencing technology on genetics. Trends Genet, 24: 133-141
Martienssen RA., 1998, Functional genomics: probing plant gene function and expression with transposons. Proc Natl Acad Sci USA, 95: 2021-2026 doi:10.1073/pnas.95.5.2021
Miyao A., Iwasaki Y., Kitano H., Itoh J., Maekawa M., Murata K., Yatou O., Nagato Y., and Hirochika H., 2007, A large-scale collection of phenotypic data describing an insertional mutant population to facilitate functional analysis of rice genes. Plant Mol Biol, 63: 625-635 doi:10.1007/s11103-006-9118-7
Miyao A., Tanaka K., Murata K., Sawaki H., Takeda S., Abe K., Shinozuka Y., Onosato K., and Hirochika H., 2003, Target site specificity of the Tos17 retrotransposon shows a preference for insertion within genes and against insertion in retrotransposon-rich regions of the genome. Plant Cell, 15: 1771-1780 doi:10.1105/tpc.012559
Nonomura K., Nakano M., Fukuda T., Eiguchi M., Miyao A., Hirochika H., and Kurata N., 2004a, The novel gene HOMOLOGOUS PAIRING ABERRATION IN RICE MEIOSIS1 of rice encodes a putative coiled-coil protein required for homologous chromosome pairing in meiosis. Plant Cell, 16: 1008-1020 doi:10.1105/tpc.020701
Nonomura K I., Nakano M., Murata K., Miyoshi K., Eiguchi M., Miyao A., Hirochika H., and Kurata N., 2004b, An insertional mutation in the rice PAIR2 gene, the ortholog of Arabidopsis ASY1, results in a defect in homologous chromosome pairing during meiosis. Mol Genet Genomics, 271: 121-129 doi:10.1007/s00438-003-0934-z
Piffanelli P., Droc G., Mieulet D., Lanau N., Bes M., Bourgeois E., Rouviere C., Gavory F., Cruaud C., Ghesquiere A., and Guiderdoni E., 2007, Large-scale characterization of Tos17 insertion sites in a rice T-DNA mutant library. Plant Mol Biol, 65: 587-601 doi:10.1007/s11103-007-9222-3
Wu C., Li X., Yuan W., Chen G., Kilian A., Li J., Xu C., Li X., Zhou D X., Wang S., and Zhang Q., 2003, Development of enhancer trap lines for functional analysis of the rice genome. Plant J, 35: 418-427 doi:10.1046/j.1365-313X.2003.01808.x
Zhang J., Guo D., Chang Y., You C., Li X., Dai X., Weng Q., Zhang J., Chen G., Li X., Liu H., Han B., Zhang Q., and Wu C., 2007, Non-random distribution of T-DNA insertions at various levels of the genome hierarchy as revealed by analyzing 13804 T-DNA flanking sequences from an enhancer-trap mutant library. Plant J, 49: 947-959 doi:10.1111/j.1365-313X.2006.03001.x