 作者
作者  通讯作者
通讯作者
豆科基因组学与遗传学, 2011 年, 第 2 卷, 第 2 篇 doi: 10.5376/lgg.cn.2011.02.0002
收稿日期: 2011年02月20日 接受日期: 2011年03月01日 发表日期: 2011年03月14日
Chen et al., 2011, Construction and evaluation of a High-qulity peanut inflorescence full-length cDNA liabray, Douke Jiyinzuxue Yu Yichuanxue(online) (Legume Genomics and Genetics) Vol.2 No.2 pp.14-19 (doi: 10.5376/lgg.cn.2011.02.0002) (陈华等, 2011, 高质量花生花序全长cDNA文库的构建和鉴定, 豆科基因组学与遗传学(online) Vol.2 No.2 pp.14-19 (doi: 10.5376/lgg.cn.2011.02.0002))
为构建花生花序全长cDNA文库,为花生花序功能基因的筛选与克隆,花序发育分子调控网络研究奠定基础。本研究采用SMART (switching mechanism at 5' end of RNA transcript)技术构建花生花序全长cDNA文库,经涂平板测定文库的克隆数,PCR测定重组率,酶切反应快速鉴定插入片断的大小,随机测序并进行生物信息学分析检测文库质量。结果表明,原始文库库容为1.2×106 cfu,插入片段大小多在1.0~2.0 kb之间,平均大小在1.3 kb左右,重组率达100%。生物信息学分析显示本文库信息量丰富,并包含了大量未知功能的新基因。所构建文库的代表性和重组片段的序列完整性达到了用于目的基因的分离筛选和克隆表达的建库要求。
花序发育研究一直是植物学家研究的重点。近年来,随着对拟南芥,金鱼草等材料的研究,克隆了不少调控花发育的同源异形基因,参与花序分生组织诱导,花分生组织转变和花器官形成的调控(Weigel and Meyerowits, 1994)。其中大部分属于MADS-box基因(Schwar et al 1990; Shore and Ssarrocks, 1995; Ma et al., 1991)。研究发现花序有许多重要生物学及医学功能。郑亚军等研究发现椰子花序汁液中的多糖具有抗氧化活性(郑亚军等, 2009)。梁淑红等发现金鸡菊提取物具有明显降血脂的作用(梁淑红等, 2010)。王晓等人发现加杨雄花序提取物具有抗氧化功能,对DNA损伤具有保护功能(王晓等, 2005)。
花序的形成及发育对花生的产量具有重要的作用。目前对花生花序形态发育、物质结构已有详尽的研究。关于花生花序在分子生物学方面研究已有报道,袁美等(2005)人在多序列比对的基础上设计兼并引物,成功地从花生中克隆花生MADS-box片段。但是相对其他植物花序,花生花序在分子方面研究尚属起步阶段,关于花生花序生长发育及物质代谢相关基因研究报道甚少。为了探讨花生花序发育机制,揭秘花生产量影响因素,发现花生花序重要基因,为基础生物学研究以及医学应用花生花序提供实验依据,我们成功构建了一个高质量的花生花序全长cDNA文库。该文库的构建对分离、克隆和筛选花序特异基因以及功能基因组学研究具有重要作用。
1结果与分析
1.1花生花序总RNA的提取
用琼脂糖凝胶电泳检测总RNA的完整性。从电泳结果来看,本实验所提取的RNA 28S rRNA和18S rRNA条带清晰,亮度比接近2:1。RNA经紫外分光光度计检测,其紫外吸收值OD260/OD280为2.05。结果表明获得的RNA纯度较高,没有基因组DNA污染,并且总RNA没有降解,比较完整,可用于cDNA文库的构建(图1)。
 图1 总RNA电泳图 Figure 1 Results of RNA Electrophoresis |
1.2双链cDNA的合成
取3 μg总RNA进行反转录反应合成第一链cDNA后采用LD-PCR法合成双链cDNA。取2 μL合成产物经1%琼脂糖凝胶电泳检测,呈现从小到大均匀分布的弥散状条带,片段分布在0.2~3.0 kb之间,而且条带清晰、正常。结果表明合成的双链cDNA质量高,可以满足全长cDNA文库构建的需要(图2)。
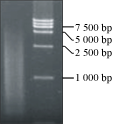 图2 花生花序dsDNA电泳图 Figure 2 Results of dsDNA Electrophoresis |
1.3文库质量的检测
双链经酶切、连接载体、电击转化、涂板过夜培养后计数培养板克隆数,计算获得原始文库含量。经计算,原始文库得到1.2×106个重组子。扩增文库经滴度测定结果为3.51×109 pfu/cm。
随机挑取的15个单菌落摇菌,菌液PCR扩增后电泳,结果显示其中含有一个双插入,其它均为但插入重组率达100% (图3)。随机挑取10个单菌落摇菌提取质粒酶切后电泳(图4)显示,插入片段大小主要集中在750~2 000 bp之间,其中750~1 000 bp之间有一个,1000~2 000 bp之间有8个,有一个插入片段小于750 bp。所构建文库插入片段平均长度约1.3 kb。
图3 菌液PCR电泳图 Figure 3 Identification of some recombinants by PCR |
图4 质粒酶切电泳图 Figure 4 Identification of the inserted fragments by enzyme digestion |
结果表明本文库达到了文库质量的要求,基本包含了花生花序全长功能基因,文库质量较高。
1.4文库随机测序和生物信息学分析结果
随机挑选文库30个单克隆进行双向测序,利用DNA序列常规分析软件BioXM 2.6分析测序结果,共有25个基因测通,且均具有开放阅读框结构。网上Blastn比对分析,其中有19个为全长序列,全长率为76%。Blastx分析显示:有1个序列未发现同源序列;其余24个均与已知的植物氨基酸序列具有同源性,其中16个为功能未知蛋白或假定蛋白序列,占66.7%。其中有7个序列与功能已知植物氨基酸序列具有同源性,所占比例为29%,包括水通道蛋白、真核生物翻译抑制因子、受体类蛋白激酶、光系统Ⅰ活性中心亚单位Ⅺ、磷脂酶 Dα2、发病机制诱导蛋白、乙烯利反应相关转录因子12及40S核糖体蛋白S2等,具体结果见表1。
表1 已知功能基因与GeneBank Blastx同源性比对结果 Table 1 Result of sequences homology comparison of function-known genes by Blastx on GeneBank |
7个基因编码序列与大豆已报道氨基酸序列同源性较高,6个与葡萄已报道氨基酸序列具有较高同源性,与花生氨基酸序列具有较高同源性的仅有3个。这可能是由于花生与大豆、葡萄同属双子叶植物,而大豆和葡萄在功能基因组学方面研究相对较深入。
生物信息学结果表明,本实验所构建的花生花序文库包含了大量的信息,同时还发现了许多未知基因,说明本文库质量较高,可用于目的基因的筛选和克隆。同时,该结果也表明花生功能基因组学研究相对薄弱,亟待加强。
2讨论
作为植物的精华花序不仅具有观赏价值,而且还具有独特的美食、保健、药用作用(徐怀德, 2000)。花序是一个着生花的变态枝,花生的花序为总状花序,在花序轴每一节上着生1片苞叶,其叶腋内着生一朵花。花生花器由苞叶、花萼、花冠、雄蕊和雌蕊组成。花生是喜温植物,温度、水分及光照强度均可影响花生开花,花生开花量直接影响花生产量。
全长cDNA文库是高效、大规模获得基因序列信息的一条有效途径,尤其是对基因组庞大,近期内尚不能进行全基因组测序的生物来说更是进行功能基因组研究的一条重要途径。目前,许多作物如小麦 (秘彩莉等, 2006)、大豆(王敏等, 2005; 董志敏等, 2006)、油菜(董海滨和管荣展, 2005)等均有以某一特定器官、特定时期或特定处理条件等为基础的全长cDNA文库构建,本实验室蔡宁波等人以SMART方法构建了花生种子全长 cDNA文库(蔡宁波等, 2007),山东省花生研究所禹山林等构建了花生幼苗全长cDNA文库(禹山林等, 2010),有关花生花序的文库未见报道。因此构建该文库对进一步克隆和研究相关基因,研究花生花序发育的分子生物学机制有着重要的意义。
SMART技术是近年发展起来的一项利用较少mRNA甚至总RNA建立cDNA文库的方法(Wellenreuther et al., 2004; Chenchik et al., 1996; Sehuler, 1997)。其主要原理是应用5′ SMART引物得到全长cDNA,并利用LD-PCR法特异扩增全长cDNA,排除了不完整的cDNA片断,并通过基因组内极其稀有的酶切位点SfiⅠ切割cDNA,得到接头并定向连接到载体中,大大提高了效率。
为了达到一个合理的概率筛选出单拷贝的目的基因,根据Clarke-Carbon的公式:N=ln(1-p)/ln(1-f),N代表所需克隆数,p即代表要求的概率,f为1/n,n为一种稀有的mRNA在总mRNA中所占有的相对比例(侯占铭, 2000)。一般情况下,cDNA文库库容达到1.0×106,就能够从库中筛选出低丰度的基因(王玉荣等, 2009),本实验所获得的cDNA文库重组子数为1.2×106 cfu,可以满足构建cDNA文库所需的数量要求。同时该cDNA文库重组率达到100%,插入片段也较长,主要集中于750~2 000 bp之间,平均插入片段长约1.3 kb,可以满足全长基因筛选的要求,对于研究花生花序发育基因、不同生物过程基因、抗耐胁迫基因、花序特异表达基因等具有重要作用。
3材料与方法
3.1材料
3.1.1实验材料
以福建农林大学油料作物所选育的优良花生品种闽花6号未开放花序为材料,-70℃冰箱保存备用。
3.1.2试剂
文库构建试剂盒CreatorTM SMARTTM cDNA Library Construction Kit购自Clontech公司。PrimerScript Reverse Transcriptase、EX-Taq,dNTP,DL2000及DL15000分子标准、电转化感受态细胞DH5α均购自大连宝生物公司(TAKARA)。胶回收试剂盒购自杭州博日科技有限公司(BIOER)。其它试剂采用国产分析纯。
所有引物均由上海生工生物技术服务有限公司合成,名称及序列如下:SMART IV Oligonucleotide:5′-AAGCAGTGGTATCAACGCAGAGTGGCCATTACGGCCGGG-3′:3′ PCR Primer:5′-AAGCAGTGGTATCAACGCAGAGTGGCCGAGGCGGCCGA-d(T) 30VN-3′ (N=A, G, C, or T; V=A, G, or C);5′ PCR Primer:5′-AAGCAGTGGTATCAACGCAGAGT-3′;M13 Forward Primer:5′-GTAAAACGACGGCCAG-3′;M13 Reverse Primer:5′-CAGGAAACAGCTATGAC-3′。
3.2方法
3.2.1花生花序总RNA的提取
取5 g花序材料放入液氮预冷的研钵中,充分研磨。采用本实验室改良的CTAB法提取总RNA。总RNA经紫外分光光度计检测浓度及纯度,经1.0%琼脂糖凝胶电泳检测其完整性。
3.2.2 cDNA单链和双链的合成
cDNA第一链的合成:在一个DEPC处理的200 μL离心管中加入以下试剂:3 μg总RNA,1 μL SMART IV Oligonucleotide,1 μL 3′ PCR Primer,72℃温浴2 min后冰浴2 min。稍加离心后依次加入下列试剂:1.0 μL RNase Inhibitor,3.0 μL MMLV Buffer,3 μL MgCl2,1.0 μL dNTP Mix (10 mmol /L),1.0 μL Primer Script Reverse Transcriptase,1.0 μL SMART IV 42℃温浴3 h后置于冰上终止第一链合成。
cDNA第二链的合成:在反应管中加入下列试剂: 2.0 μL的第一链cDNA、2.5 μL 10×Ex Buffer、0.5 μL 10 mmol/L dNTP、1.0 μL 5′ PCR Primer、0.25 μL ExTaq,双蒸水补足至25 μL,混匀各组分,稍加离心后放到已预热(95℃)的PCR仪中。按下面程序开始PCR:95℃ 2 min;8 cycles:95℃ 15s,68℃ 30s,72℃ 5 min;72℃ 10 min。循环结束后取2 μL产物电泳检测。
3.2.3蛋白酶K消化与SfiI限制性酶切
取50 μL双链cDNA的PCR产物,加入2 μL蛋白酶K (20 g/L),45℃温育20 min灭活DNA聚合酶活性,用酚/氯仿/异戊醇和氯仿/异戊醇各抽提一次,然后用80%乙醇、醋酸钠、糖原室温沉淀。沉淀用85%乙醇洗涤以后用75 μL去离子水重悬。
重悬后的产物中加入15 μL SfiI酶,于50℃酶切2 h,再以1.5%琼脂糖凝胶电泳酶切产物,回收750 bp以上片段。回收产物经乙醇、醋酸钠、糖原-20℃共沉淀后,以7 μL去离子水重悬,保存于-70℃备用。
3.2.4双链与载体的链接和转化
酶切产物与质粒载体pDNR-LIB (经SfiⅠ酶处理过)在16℃下连接后经电击转化入45 μL感受态细胞(大肠杆菌DH5a)。转化后迅速加入950 μL LB培养基,置于37℃,225 r/min振荡培养1 h即获得1 mL原始文库菌液。
3.2.5文库质量的检测
吸取1 μL原始文库菌液均匀地涂在含30 mg/L氯霉素的LB培养板上,过夜培养。计数过夜培养LB板单克隆数,计算原始文库量,原始文库量(cfu)=克隆数×103。
随机挑取22个单克隆摇菌,过夜后做菌液PCR鉴定文库重组率。PCR引物为M13引物。随机挑取10个单菌落摇菌提取质粒经SfiⅠ酶切后电泳检测文库cDNA插入片段的大小。随机挑取30个单菌落做穿刺stock并送上海国家人类基因组南方研究中心进行测序并对测序结果进行生物信息学分析。
作者贡献
陈华是本研究的实验设计和实验研究的执行人,完成数据分析,论文的写作与修改;姜宝杰参与实验设计、实验研究与试验结果分析;邓烨、曾建斌、张冲参与实验研究;庄伟建是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改,是本文的责任作者(通信作者)。全体作者都阅读并同意最终的文本。
致谢
本研究是在福建省科技厅项目基因工程改良花生油亚比(O/L)的研究(2008I0002)和科技部国际科技合作项目花生抗黄曲霉基因工程及育种研究(2008DFA31150)资助下完成。
参考文献
Bei C.L., Guo G.Y., Qi Z.G., and Shen Y.Z., 2006, Construction and detection of a cDNA library of wheat seedling salt stressed for 72 hours, Journal of Hebei Normal University (Natural Science Edition), 30(5): 585-588 (秘彩莉, 郭光艳, 齐志广, 沈银柱, 2006, 小麦苗期盐胁迫72 h cDNA文库的构建及质量检测, 河北师范大学学报(自然科学版), 30(5): 585-588)
Cai N.B., Huang X.W., and Zhuang W.J., 2007, Construction and identification of a full-length cDNA library from peanut seeds, Journal of Peanut Science, 36(2): 1-5 (蔡宁波, 黄湘文, 庄伟建, 2007, 花生种子全长cDNA文库的构建和鉴定, 花生学报, 36(2): 1-5)
Chenchik A., Moqadam F., and Siebert P., ed, 1996, A new method for full-length cDNA cloning by PCR, Krieg P A.A laboratory guide to RNA isolation, analysis and synthesis, Wiley-Liss, Inc, New York, 273-32l
Dong H.B., and Guan R.Z., 2005, Construction of seedling cDNA library of canola variety Huashuang 3 in Brassica napus, Journal of Nanjing Agricultural University, 28(3): 123-125 (董海滨, 管荣展, 2005, 双低油菜华双3号幼苗全长cDNA文库的构建.南京农业大学学报, 28(3): 123-125)
Dong Z.M., Li Y.H., Zhang B.S., Guan R.X., Chang R.Z., and Qiu L.J.,2006, Construction and identification of soybean (Glycine max) leaf, Crops, (5): 1-4 (董志敏, 李英慧, 张宝石, 关荣霞, 常汝镇, 邱丽娟, 2006, 大豆叶片全长cDNA文库的构建与鉴定, 作物杂志, (5): 1-4)
Hou Z.M., 2000, A Deduction for the Clarke-Carbon’s Equation, Hereditas (Beijing), 22(2): 101-102 (侯占铭, 2000, Clarke-Carbon公式的推导, 遗传, 22(2): 101-102)
Liang S.H., Pang S.B., Liu X.Y., Xu L., Ha M.L.T., and Su Y.H., 2010, The coreopsis extraction’s hypolipidemic effect research, Chinese Journal of Experimental Traditional Medical Formulae, 16(8): 234-235 (梁淑红, 庞市宾, 刘晓燕, 徐磊, 哈木拉提, 孙玉华, 2010, 金鸡菊提取物降血脂作用的研究, 中国实验方剂学杂志, 16(8): 234-235)
Ma H., Yanofsky M.F., and Meyerowitz E.M., 1991, AGL1~AGL6, an Arabidopsis gene family with similarity of floral homeotic and transcription factor genes, Genes Dev., 5: 484-495 doi:10.1101/gad.5.3.484
Schwar Z.S., Huijser P., Nacken W., Saedler H., and Sommer H., 1990, Genetic control of flower development: homeotic genes in Antirrhinum majus, Science, 250: 931-936 doi:10.1126/science.250.4983.931
Sehuler G.D., 1997, Pieces of the puzzle: expressed sequence tags and the catalog of human genes, J. Mol. Med., 75: 694-698
Shore P., and Ssarrocks A.D., 1995, The MADS-box family of transcription factors, Eur. J. Bio. Chem., 229: 1-13
Wang M., Liu P., Shi M.W., and Wang Q.L., Construction and characterization of cDNA library from seeds of wild soybean (Glycine soja), Letters in Biotechnology, 16(5): 509-511 (王敏, 刘萍, 石明旺, 王清连, 2005, 野生大豆种子cDNA文库的构建与分析, 生物技术通讯, 16(5): 509-511)
Wang X., Zhang H.X., Deng Y.G., Shi X.G., and Liu J.H., 2005, Effects of the extract of Populus Canadensis Moench. male anthotaxy on antioxidation and prevention of DNA damage caused by hydroxyl radical in vitro, Food Science, 26(4): 216-219 (王晓, 张红侠, 邓煜光, 时新刚, 刘建华, 2005, 加杨雄花序提取物体外抗氧化及对DNA损伤的保护作用研究, 食品科学, 26(4): 216-219)
Wang Y.R., Zhang X.Y., Liu C.L., Chen Y.J., Li F.G., 2009, Construction and characterization of normalized full-Length cDNA library of Asian cotton (Gossypium arboreum L.) in the whole-Life cycle, Scientia Agricultura Sinica, 42(4): 1158-1164 (王玉荣, 张雪妍, 刘传亮, 陈亚娟, 李付广, 2009, 亚洲棉(Gossypium arboreum L.)全生育期均一化全长cDNA文库的构建和鉴定, 中国农业科学, 42(4): 1158-1164)
Weigel D., and Meyerowits E.M., 1994, The ABCs of floal homeotic genes, Cell, 78: 203-209
Wellenreuther R., Schupp I., Poustka A., Wiemann S., German cDNA Consortium., 2004, SMART amplification combined with cDNA size fractionation in order to obtain large full-length clone, BMC Genomics, 36(5):1-8
Xu H.D., ed, 2000, Flowers foods, China Light Industry Press, Beijing, China, pp.116-228 (徐怀德, 主编, 2000, 花卉食品, 中国轻工业出版社, 中国, 北京, pp.116-228)
Yu S.L., Chi X.Y., Yang Q.L., Pan L.J., He Y.N., Chen M.N., and Yang Z., 2010, Construction of a full-length cDNA library from peanut seedlings, Journal of Peanut Science, 39(2): 11-15 (禹山林, 迟晓元, 杨庆利, 潘丽娟, 和亚男, 陈明娜, 杨珍, 2010, 花生幼苗全长cDNA文库的构建, 花生学报, 39(2): 11-15)
Yuan M., Sharma K.K., Li S.L., Tao H.T., Ren Y., and Yu S.L., 2005, Cloning of a partial MADS-box gene in groundnut (Arachis hypogaea L.), Journal of Peanut Science, 34(4): 25-27 (袁美, Sharma K.K., 李双铃, 陶海腾, 任艳, 禹山林, 2005, 花生MADS-box基因片段的克隆, 花生学报, 34(4): 25-27)
Zheng Y.J., Chen W.J., and Xin B.,2009, Antioxidant activities of polysaccharides from coconut inflorescence sap, Chinese Journal of Tropical Crops, 30(3): 392-395 (郑亚军, 陈卫军, 辛波, 2009, 椰子花序汁液中多糖的抗氧化活性, 热带作物学报, 30(3): 392-395)