2. 新疆农业大学农学院, 乌鲁木齐, 830052
1 亚热带森林培育国家重点实验室培育基地,浙江农林大学,杭州临安 311300
 作者
作者  通讯作者
通讯作者
基因组学与应用生物学, 2011 年, 第 30 卷, 第 16 篇 doi: 10.5376/gab.cn.2011.30.0016
收稿日期: 2011年03月17日 接受日期: 2011年05月06日 发表日期: 2011年05月10日
引用格式(中文):
何广海等,2011,姜黄EST-SSR信息分析与标记开发,基因组学与应用生物学(online),Vol.30 No.16 pp.1098-1104 (doi: 10.5376/gab.cn.2011.30.0016)
引用格式(英文):
He et al.,2011, Information analysis and molecular marker exploitation of EST-SSR in Curcuma longa L, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), Vol.30 No.16 pp.1098-1104 (doi: 10.5376/gab.cn.2011.30.0016)
为从现有的姜黄EST数据中获取有效的微卫星标记,我们从NCBI网站上下载了12 593条姜黄EST序列,然后利用在线程序Websat对所有下载的EST序列进行搜寻SSR位点并设计引物。对于已设计出引物的EST序列,通过重复类型、Tm值和引物序列比对去除冗余EST,得到12 513条无冗余EST序列,全长为8 441.73 kb。结果共搜索到565个SSR位点,出现频率为4.52%,平均分布距离为14.94 kb,其中有452个位点能设计出引物(占80%)。在姜黄EST-SSR中,三、六核苷酸重复基元是主导类型,分别占总SSR的42.30%和34.34%;TA/AT是二核苷酸优势重复基元,占二核苷酸总数的42.42% (28/66)。根据重复类型所占比例随机选取30对引物,以生姜基因组DNA为模板,对引物进行筛选,20对引物检测到多态性,有效扩增率为66.67%。结果证明,基于姜黄EST信息建立SSR标记是可行的且在姜科植物中具有通用性。
EST-SSR(表达序列标签微卫星)技术就是利用已有的EST序列通过电子筛选鉴别SSR,然后根据SSR两端保守的侧翼序列设计引物,通过PCR扩增基因组编码区的微卫星序列,将扩增片段进行聚丙烯酰胺凝胶电泳,再经银染后分析微卫星中核心序列重复单位数目的多态性(李卫国等, 2008)。EST-SSR标记源自于比较保守的转录区,其侧翼序列往往在物种之间高度保守,因此EST-SSR在相关物种之间具有很高的可转移性和通用性。对于尚未进行EST测序的物种,利用其近缘物种已有的EST-SSR标记或EST序列来开发目标物种的SSR标记不失为一种有效的替代方法,这加快了物种间的基因组信息转换,为基因组研究提供了新的思路和途径。利用EST开发SSR避免了SSR引物开发过程中的克隆和测序步骤,充分利用了现有数据,降低了开发成本,在生物上成为应用最广、发展最快、最为重要的分子标记(黄心尧和卢茵, 2010, 禽畜业, 256(8): 40-43)。
姜黄为姜科姜黄属植物姜黄(Curcuma longa L.)的干燥根茎,其主要成分为挥发油和油姜黄素类化合物(姜黄素, 去甲氧基姜黄素和去二甲氧基姜黄素)。姜黄挥发油除具有广谱抗菌消炎作用,还有抗癌、抗生育作用(陈晋红等, 2009)。而姜黄素是国内外食品行业允许使用的7大天然色素之一。现代药理研究证明,姜黄素类物质具有多种药理作用,包括抗炎、抗氧化、抗凝、降血脂及抗动脉粥样硬化、抗肿瘤作用。其抗癌谱广,毒副作用小,是一种具有广泛应用前景的抗癌新药,有的已进入临床前毒理试验阶段(刘硕谦等, 2005)。
由于姜黄具有较高药用价值,国内外对姜黄的研究方兴未艾,NCBI数据库中积累了大量姜黄EST资源,极大增加了姜黄EST-SSR标记的开发能力。但目前关于利用姜黄EST序列建立其多态性的标记还未见报道。本研究利用现有的姜黄EST资源,对其进行搜索SSR并设计引物,建立了EST-SSR标记,以便于对该植物更好地研究、开发和利用。
1结果与分析
1.1姜黄EST-SRR的分布和频率
姜黄EST数据库共有12 593条序列,其中冗余序列80条,因而非冗余序列有12 513条,序列全长8 441.73 kb。此次研究发现24条序列具有2个位点,5条具有3个位点,其中6条具有重叠位点,共搜索到565个SSR位点,其中有452个位点能设计出引物(占80%)。
姜黄EST-SRR种类较为丰富,二至六核苷酸重复类型都能被检出,但出现的频率迥异。属于二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复的位点分别占总SSR 11.68% (66/565)、42.30% (239/565)、6.73% (38/565)、4.96% (28/565)和34.34% (194/565) (表1)。由此可见,在姜黄EST-SRR中,三、六核苷酸重复类型占主导地位,两者共占了总SRR的76.64%。从SRR的分布情况看,姜黄EST中平均每14.94 kb就出现1个SRR,但不同重复类型出现的平均距离各不一致, EST-SRR出现的频率越高,其平均距离越小(表1)。
 表1 姜黄EST-SSR的分布特征 |
优势重复基元的比较分析表明,TA/AT是二核苷酸优势重复基元,占二核苷酸总数的42.42% (28/66);三核苷酸中TCT/AGA,GAG/CTC,GAA/CTT均是出现22次,每个占三核苷酸总数的9.21%,共占27.62%;四核苷酸中AATA,TAGA和五核苷酸中TCCTT/AGGAA,AGAAG/TCTTC比例相当,分别共占各苷酸类型总数的21.05% (4*2/38),21.43% (3*2/28);六核苷酸优势重复基元不明显,GGCGAC,CATTCC,GCGGCA/CGCCGT,GAGGCG/CTCCGC每个仅占六核苷酸总数的2.58% (5/194),共占10.31%。
1.2姜黄EST-SRR重复单位的特点
1.2.1重复次数
重复单元的重复次数是SSR的重要特征。就姜黄EST-SRR重复单元的重复次数而言,在3~11次的重复范围内都有连续分布,其中最多的是3次重复138个,占总SSR的24.42%;其次为6次重复124个,占21.95%;然后是7次重复70个,占12.39%;其它重复次数的则少得多,均未超过10.00% (图1)。姜黄EST-SSR重复单元的重复次数最多的达到45次,但仅有一例,即AG重复类型。
 图1 姜黄EST-SSR主要重复单元的重复次数分布 |
1.2.2基序长度
从图2可以看出,基序长度为18 bp处绝对优势,占总SSR的46.02%;长度为24 bp、21 bp次之,分别占14.34%、12.04%;而长度为20 bp、30 bp、22 bp及其它的则比较少,均未超过10.00%。Temnykh等(2001)报道,当SSR长度在20 bp以上(含20 bp)时,在不同品种间显示出较高的多态性;当长度在12~20bp之间时,SSR的多态性较低;当长度小于12 bp时,SSR的多态性很低。在姜黄EST-SSR搜寻过程中设置的最小长度标准为18 bp,已完全过滤多态潜能很低的小于12 bp的SSR。在本研究所发掘的565个SSR中,长度20 bp以上的SSR有299个,18~20 bp的SSR有266个,即多态潜能高的SSR占52.92%,多态潜能次高的SSR占47.08%。
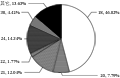 图2 姜黄EST-SSR主要基序长度分布 |
1.3 EST-SSR通用性检测
根据统计的SSR位点的EST序列信息,共设计了452对引物。为了验证姜黄EST-SSR标记的有效性,按二、三、四、五和六核苷酸在总SSR中所占比例,随机选取30对引物,其中包含两条Over-laped SSR所对应的引物,对生姜的基因组DNA进行PCR扩增,共筛选出20对扩增出清晰条带,有效扩增率为66.67%,6%非变性聚丙烯酰胺凝胶电泳结果见图3。在30对EST-SSR引物中,20对有扩增产物,有3对引物扩增出单一带谱,其余17对引物检测的等位基因较丰富在2~6个之间,平均3.06个/对(表2)。该实验说明利用姜黄EST序列开发EST-SSR标记是有效且可行的。
 表2 20对姜黄EST-SSR引物信息 |
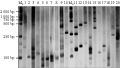 图3 6%变性聚丙烯酰胺凝胶电泳图 |
1.4 EST-SSR靶向基因的功能分析
值得关注的是,EST-SSR可能与基因功能表达具有直接或间接关系,从而强化了SSR标记在遗传研究中的应用。筛选出姜黄20对引物所对应的EST,预测ORF并概念性翻译序列,随后采用BLASTp搜索蛋白质数据库,发现获得的位点中5个(5/20, 25%)为编码假定蛋白,1个(1/20, 5%)为未知蛋白,4个(4/20, 20%)为未见报道,而9个(9/20, 45%)的SSR位点可被成功进行功能注释,其中有尿苷二磷酸葡萄糖脱氢酶,墨西哥类蜀黍硬壳构造Tga1,氨基酸转运蛋白,叶绿素a/b结合蛋白,EF手臂钙离子结合蛋白CCD1,HemY蛋白,转录因子,RNA结合蛋白Nova-1。与此同时,我们发现8个(8/20, 40%)具有明显结构域。详细结果见表2。
2讨论
对NCBI数据库12 593条姜黄EST序列去除冗余后,共得到非冗余序列12 513条,序列全长8 441.73 kb,从中共搜索到565个SSR位点,平均距离为14.94 kb。本研究中得到的姜黄EST-SRR平均分布距离低于很多植物,如黄瓜(3.80 kb) (胡建斌和李建吾, 2008)、杨树(3.88 kb) (张新叶等, 2009)、大豆(6.62 kb) (陈相艳等, 2009),但与烟草(14.21 kb) (胡重怡等, 2009)相近,这种差异可能是物种间的真实SSR信息差异或搜寻SSR时所用标准(如最低长度、是否统计单核苷酸)不同造成的。
从目前的报道来看,大多数植物的EST-SSR以三核苷酸和二核苷酸重复类型为主(李永强等, 2004)。分析姜黄EST-SSR的重复类型,发现三、六核苷酸重复类型占主导地位,两者共占了总SRR的76.64%,其中三核苷酸42.30% (239/565),六核苷酸34.34% (194/565)。这与前人研究其他植物结果存在一定差异。优势重复基元的比较分析表明,TA/AT是二核苷酸优势重复基元,占二核苷酸总数的42.42% (28/66),这与Powell等认为植物二核苷酸重复中主要是(AT)n的看法一致(姜春芽等, 2009)。GC重复基元在多数植物中很难见到,如拟南芥、杏树、桃树、水稻和玉米(Gao et al., 2003)等植物中都没发现。姜黄EST-SSR二核苷酸中同样未发现GC基元。
研究结果表明,利用姜黄EST信息建立SSR标记是可行的且在生姜中具有通用性,为其它姜科植物的进一步研究奠定了基础。
3材料与方法
3.1姜黄EST序列的获得
登陆NCBI数据库(http://www.ncbi.nlm.nih. gov/guide/),以Curcuma longa为关键词搜索EST序列,共获得EST序列12 593条(截至2011年2月22日)。
3.2姜黄EST-SSR的发掘及引物设计
利用在线程序Websat (http://wsmartins.net/websat/)对所有下载的EST序列进行SSR位点搜寻。搜寻标准为:二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸,最少重复次数分别为9、6、5、4和3。采用Websat内置Primer Premier 3.0程序,使用默认参数对含有SSR的EST设计PCR引物。
3.3姜黄EST-SSR的统计分析
对于已设计出引物的EST序列,通过重复类型、Tm值和引物序列比对进一步去除冗余EST。姜黄EST-SSR类型、数目及出现频率等采用Excel软件进行统计分析。根据重复类型所占比例随机选取30对引物,由上海生工生物工程技术服务有限公司合成。
3.4姜黄EST-SSR引物性能检测
采集新鲜生姜块茎于-20℃保存,采用改良CTAB法(张盈娇, 2006, 荨麻部分药理作用及有效成分的研究和干姜遗传多样性研究, 硕士学位论文, 成都中医药大学, 导师: 卫莹芳, pp.37-41)提取生姜基因组DNA。PCR扩增体系为(25 μL):10×buffer 2.5 μL, Mg2+ 25 mmol/L 1.5 μL, dNTP 10 mmol/L 0.5 μL,上下游引物100 μmol/L各1 μL,Taq酶5.0 U 0.2 μL,模板DNA 50 ng/μL 1 μL。PCR反应程序为:94℃预变性5 min;94℃变性30 s,51℃退火40 s,72℃延伸50 s,38个循环;72℃终延伸7 min,8℃保存。PCR产物用1.5%琼脂糖凝胶电泳分离,以检测扩增产物的可用性,对有清晰条带的引物用6%变性聚丙烯酰胺凝胶电泳结合银染检测其多态性。
作者贡献
在整个实验过程以及最终成稿期间,何广海在张智俊老师、罗淑萍老师的指导和帮助下完成选题、文章初稿的写作以及文章的整体构思、修改和完善。
致谢
本研究由国家“863”项目(2007AA021403)、浙江省自然科学基金资助项目(Y307469)、浙江农林大学人才启动基金共同资助。特别感谢杨丽学姐、管雨学姐在实验过程中提供的帮助。感谢两位匿名的同行评审人的评审意见和修改建议。
参考文献
Chen J.H., Li W.R., Liu D.W., Huang T.L., Mi S.Q., and Wang N.S., 2009, Determination of effective constituents in Rhizoma Curcumae longa, Zhongyao Xinyao Yu Linchuang Yaoli (Traditional Chinese Drug Research and Clinical Pharmacology), 20(3): 253-256 (陈晋红, 李伟荣, 刘大伟, 黄天来, 宓穗卿, 王宁生, 2009, 姜黄药材中有效成分含量测定, 中药新药与临床药理, 20(3): 253-256)
Chen X.Y., Li W., Dai H.Y., and Zhang L.F., 2009, Analysis of SSR information in EST resource of soybean (Glycine max), Dadou Kexue (Soybean Science), 28(3): 394-399 (陈相艳, 李伟, 戴海英, 张礼凤, 2009, 大豆EST资源的SSR信息分析, 大豆科学, 28(3): 394-399)
Gao L.F., Tang L.F., Li H.W., and Jia J.Z., 2003, Analysis of microsatellites in major crops assessed by computational and experimental approaches, Molecular Breeding, 12(3): 245-261
Hu J.B., and Li J.W., 2008, Distribution of EST-SSRs in cucumber genome and development of EST-SSR marker, Xibei Zhiwu Xuebao (Acta Botanica Boreali-Occidentalia Sinica), 28(12): 2429-2435 (胡建斌, 李建吾, 2008, 黄瓜基因组EST-SSRs的分布规律及EST-SSR标记开发, 西北植物学报, 28(12): 2429-2435)
Hu Z.Y., Cai L.T., and Chen X.J., 2009, Analysis of SSR information in ESTs resource of tobacco (Nicotiana tabacum), Shengwu Jishu Tongbao (Biotechnology Bulletin), (7): 82-85 (胡重怡, 蔡刘体, 陈兴江, 2009, 烟草ESTs资源的SSR信息分析, 生物技术通报, (7): 82-85)
Jiang C.Y., Liao J., Xu X.B., Gu Q.Q., Liu S.J., and Chen J.Y., 2009, Plant EST-SSR technology and its application, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 7(1): 125-129 (姜春芽, 廖娇, 徐小彪, 辜青青, 刘善军, 陈金印, 2009, 植物EST-SSR技术及其应用, 分子植物育种, 7(1): 125-129)
Li W.G., Chang T.J., and Gong H.M., 2008, Simple sequence repeats derived from expression sequence tags (EST-SSRs) and its application to plant genomics, Shengwu Jishu (Biotechnology), 18(4): 90-93. (李卫国, 常天俊, 龚红梅, 2008, EST-SSR及其在植物基因组学研究中的应用, 生物技术, 18(4): 91-93)
Li Y.Q., Li H.W., Gao L.F., and He B.R., 2004, Prograss of simple sequence repeats derived from expressed sequence tags, Zhiwu Yichuan Ziyuan Xuebao (Journal of Plant Genetic Resources), 5(1): 91-95 (李永强, 李宏伟, 高丽锋, 何蓓如, 2004, 基于表达序列标签的微卫星标记(EST-SSRs)研究进展, 植物遗传资源学报, 5(1): 91-95)
Liu S.Q., Liu Z.H., Huang J.A., Tian N., Lu Y., and Luo G.A., 2005, Simultaneous determination of three main components in Curcuma longa L. by reversed-phase high performance liquid chromatography, Fenxi Huaxue (Chinese Journal of Analytical Chemistry), 33(3): 309-312 (刘硕谦, 刘仲华, 黄建安, 田娜, 陆英, 罗国安, 2005, 反相高效液相色谱法同时测定姜黄药材中3个组分的含量, 分析化学, 33(3): 309-312)
Temnykh S., DeClerck G., Lukashova A., Lipovich L., Cartinhour S., and Mccouch S., 2001, Computational and experimental analysis of microsatellites in rice (Oryza sativa L.): Frequency, length variation, transposon associations, and genetic marker potential, Genome Res., 11(8): 1441-1452
Zhang X.Y., Song C.W., Zhang Y.D., Yang Y.L., and Huang M.R., 2009, Development of EST-SSR in Populus deltoides and P. Euramericana, Linye Kexue (Scientia Silvae Sinicae), 45(9): 53-59 (张新叶, 宋丛文, 张亚东, 杨彦伶, 黄敏仁, 2009, 杨树EST-SSR标记的开发, 林业科学, 45(9): 53-59)