2吉林省农业科学院大豆研究所, 长春, 130033
 作者
作者  通讯作者
通讯作者
豆科基因组学与遗传学, 2014 年, 第 5 卷, 第 3 篇
收稿日期: 2014年06月01日 接受日期: 2014年06月01日 发表日期: 2014年06月01日
引用格式(中文):
刘海军, 赵丽梅, 董英山, 石瑛, 张春宝, 2014, 大豆细胞质雄性不育系及其保持系线粒体基因的RNA编辑位点研究, 分子植物育种, 12(4): 694-700 (doi: 10.13271/j.mpb.012.000694)
引用格式(英文):
Liu H.J., Zhao L.M., Dong Y.S., Shi Y., and Zhang C.B., 2014, RNA Editing Analysis of Mitochondrial Gene in Cytoplasmic Male Sterile Line and Maintainer Line in Soybean, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 12(4): 694-700 (doi: 10.13271/j.mpb.012.000694)
RNA 编辑可引起线粒体相关基因的碱基发生插入、缺失或替换,进而影响初始转录物的剪接和加工,形成细胞质雄性不育(CMS)。本研究以大豆 RN 型 CMS 不育JLCMS9A 及其对应的保持系 JLCMS9B 为材料,比较分析了大豆线粒体基因 atp1、atp6、atp9、coxⅡ、coxⅢ和 cob 的编辑位点与 CMS 的关系。结果表明,在 JLCMS9A 和 JLCMS9B 中 coxⅡ未发生 RNA 编辑;atp1、atp9、coxⅢ和 cob 基因发生的 RNA 编辑与 CMS 无关;而 atp6 基因在 JLCMS9A 中比 JLCMS9B 多 3 个编辑位点,其中 2 个位点导致了氨基酸变化,并使得预测的蛋白结构发生变化。因此,推测这种改变很可能引起 JLCMS9A 中 atp6 基因的正常功能受到影响,而形成 CMS。
RNA 编辑(RNA editing)是一种通过核苷酸的插入、缺失或碱基修饰,使得转录表达的氨基酸编码序列不同于基因组序列现象,它可以在几乎所有的植物线粒体中被检测出来,是RNA 成熟化的表现以及线粒体基因产生功能蛋白的必备环节,同时还是细胞核调控线粒体基因表达的关键方式(Gray et al., 1992;Malek et al., 1996; Brennicke et al., 1999)。细胞质雄性不育(cytoplasmic male sterile, CMS)是由细胞质基因和核基因互作控制的不育,是生产杂交种的重要途径之一。研究表明,CMS 与高等植物中线粒体的RNA 编辑有重要关系,主要是由于RNA 编辑,导致线粒体相关基因的碱基发生插入、缺失或替换,进而影响初始转录物的剪接和加工,导致了基因结构或表达调控模式发生改变,或者形成嵌合基因等,这些非正常或不充分的编辑产物可能会导致线粒体功能的非正常发挥而形成CMS (Stahl et al., 1994)。
Howad 和Kempken (1997)对高粱线粒体的atp6、atp9、nad3、nad4 和rps12 基因的研究中发现,其雄性不育产生的原因可能是atp6 基因发生了RNA 编辑。Rurek 等(2001)对胡萝卜不育系和保持系的nad3 基因进行了RNA 编辑研究,发现不育系与保持系相比,有3 处碱基发生了C-U 转换,可能引起CMS。孔进等(2006)以红莲型水稻细胞质雄性不育系粤泰A和保持系粤泰B 及杂种红莲优6 为材料,研究了atp6、coxⅡ及嵌合基因aorfH79 的RNA 编辑位点,结果表明,atp6 基因的RNA 编辑能力受恢复基因的影响。coxⅡ基因在3 个材料中RNA 编辑状态没有变化,而嵌合基因aorfH79 在各个材料中均不被编辑,推测红莲型水稻细胞质雄性不育与atp6 基因RNA 编辑能力的丧失紧密相关。Yang 等(2007)对芥菜atp9 基因的RNA 编辑进行了研究,发现不育系atp9 基因与正常可育材料间发生了4 处碱基变化,其中3 处为C-U 的变化,一处为U-C 改变,推测其可能导致atp9 蛋白的疏水性降低,还可进一步推测RNA 编辑所产生的特定的线粒体基因的功能障碍,很可能是导致CMS 的一个因素。Wei 等(2008)对紫稻细胞质雄性不育系樱香A 及其保持系樱香B 的atp6 基因转录本cDNA 序列进行分析发现,樱香A中没有发生RNA 编辑,而樱香B 的atp6 基因cDNA序列中有16 个编辑位点,位于15 个密码子中,所编码的氨基酸均发生改变,尤其是在1003 位点由C 替换为T,导致原来编码谷氨酰胺密码子(CAA)成为终止密码子(TAA),保证了atp6 mRNA 编码一个正常的多肽。然而,在不育系atp6 基因由于没有发生RNA 编辑,导致其缺乏终止密码子而不能形成正常的多肽,因此认为其可能与CMS 相关。Wang 等(2009)对同核异质和同质异核玉米材料的atp6 和coxⅡ基因的RNA 编辑进行了研究,发现两个基因均存在大量RNA 编辑位点,并认为这两个基因的RNA 编辑可能与雄性不育有关。
中国是世界上最早利用细胞质雄性不育生产大豆杂交种的国家(Sun et al., 1994; 赵丽梅等, 2004)。目前已利用RN 型CMS“三系”选育春大豆杂交种8个;利用ZD 型CMS“三系”选育了夏大豆杂交种3个,在大豆杂种优势利用方面处于世界领先地位,然而我们在CMS 机理研究方面的报道较少。由于有大量研究表明CMS 与RNA 编辑有关,韩利涛等(2010)对大豆N8855 型CMS 不育系NJCMS1A 与其保持系NJCMS1B 的atp6 基因的RNA 编辑进行了分析,发现在不育系与保持系的atp6-3 基因保守区中均发现2 个互不相同且编码不同氨基酸的编辑位点。Jiang等(2013)采用同样方法对NJCMS2A 与NJCMS2B 的atp9 基因进行了比较分析,发现在cDNA 水平上不育系和保持系之间发生了两次C-U 转换,导致二者编码蛋白不同,但是否其是导致所用不育系产生CMS 的原因,并未加以证实。
本研究以吉林省农业科学院选育的大豆RN 型CMS 不育系JLCMS9A 和保持系JLCMS9B 为材料,对已在其它植物中发现的与CMS 有关的6 个线粒体基因atp1、atp6、atp9、coxⅡ、coxⅢ和cob 进行RNA编辑位点研究。希望通过分析大豆CMS 不育系和保持系中这些基因在转录水平和基因组水平的差异,探讨它们是否与RN型细胞质雄性不育有关。
1结果与分析
1.1目的基因的扩增
首先以大豆内参基因Actin11 片段引物(F: 5'-GGGCATTTGGATTTGTTTCG-3'; R: 5'-GAAGTTATTTGAAGGACCG-3'),对JLCMS9A 和JLCMS9B 的cDNA 第一链进行检测,确定其中是否含有DNA 污染。该片段含有330 bp 内含子序列的,通过电泳检测发现,cDNA 扩增片段长度为390 bp 左右,DNA扩增片段长度为720 bp 左右,与预计片段长度相符,证实JLCMS9A 和JLCMS9B 的cDNA 中无DNA 污染(图1)。
.png) 图 1 不育系和保持系cDNA 第一链纯度检测图 注: M: 1 kb DNA ladder marker; 1: JLCMS9A cDNA; 2: JLCMS9B cDNA; 3: JLCMS9A DNA; 4: JLCMS9B DNA Figure 1 Purity test of cDNA first strand in CMS and maintainer line Note: M: 1 kb DNA ladder marker; 1: JLCMS9A cDNA; 2: JLCMS9B cDNA; 3: JLCMS9A DNA; 4: JLCMS9B DNA |
atp6、atp9、coxⅡ、coxⅢ和cob 基因进行PCR扩增,扩增产物电泳结果如图2 所示,所有目的基因中均能扩增出与预计片段大小一致的条带,而且基因组水平和转录水平扩增出的片段长度基本相同。另外,本研究还进行了2 次重复PCR 扩增,3 次PCR产物测序结果完全一致,确保了扩增序列的准确性。进一步比对基因组水平和转录水平扩增序列的测序结果证实,这6 个基因均无内含子。
.png) 图 2 不育系和保持系的 atp1, atp6, atp9, coxⅡ, coxⅢ和 cob 基因扩增结果 注: 1: JLCMS9A 的 mtDNA 扩增结果; 2: JLCMS9B 的 mtDNA 扩增结果; 3: JLCMS9A 的 cDNA 扩增结果; 4: JLCMS9B 的 cD- NA 扩增结果 Figure 2 Amplification results of atp1, atp6, atp9, coxⅡ, coxⅢ and cob genes in CMS and maintainer line Note: 1: The amplification results of mtDNA of JLCMS9A; 2: The amplification results of mtDNA of JLCMS9B; 3: The amplification results of cDNA of JLCMS9A; 4: The amplification results of cDNA of JLCMS9B |
1.2目的基因序列的RNA 编辑分析
用Vector NTI Suite 8.0 软件的Align X 功能对所有基因的基因组序列和cDNA 序列进行分析发现,所有基因序列在基因组水平上都无差异;在转录水平上,atp1 仅在JLCMS9B 中发生了RNA 编辑,而atp6、atp9、cox Ⅲ 和cob 这4 个基因在JLCMS9A 和JLCMS9B的cDNA 序列中均发生了RNA 编辑,所有编辑均发生在密码子的第一或第二位点上,且都是由C-U 的转变(图3),而coxⅡ基因在JLCMS9A 和JLCMS9B中均未发生RNA 编辑。
.png) 图 3 大豆 atp1, atp6, atp9, coxⅢ和 cob 基因的核苷酸序列及 RNA 编辑位点 注: A 和 B 为不育系和保持系线粒体 DNA 序列; CA 和 CB 为不育系和保持系 cDNA 序列; 灰色和深灰色背景为一致性序列, 白色背景为 RNA 编辑位点 Figure 3 The nucleotide sequence and RNA editing sites of atp1, atp6, atp9, coxⅢ and cob in soybean Note: A and B are mtDNA sequences of CMS and maintainer line; CA and CB are cDNA sequences of CMS and maintainer line; Con- sensus sequences are in gray and dark gray background, RNA editing sites are in white background |
其中atp1 基因虽然在JLCMS9B 中特异性存在RNA 编辑位点,但其翻译的氨基酸序列与JLCMS9A翻译的氨基酸序列完全相同,不存在蛋白功能变化,因此可推断其与CMS 无关。atp9、coxⅢ和cob 这3 个基因在不育系和保持系的cDNA 序列中均发生了C-U 的转变,这其中coxⅢ基因发生RNA编辑的比率最高,达到了1.38%,而cob 为1.19%,atp9为0.89% (表1)。虽然上述3 个基因均在JLCMS9A 和JLCMS9B 中发生了不同比率的RNA 编辑,但这些基因在两份材料中发生编辑的位点一致,最终编码氨基酸也完全相同,因此与atp1 基因一样,它们在转录水平发生的C-U 转换与CMS 不存在直接关系。
.png) 表 1 不育系和保持系中各基因RNA编辑发生比率 Table 1 RNA editing occurrence ratio of each gene between CMS and maintainer line |
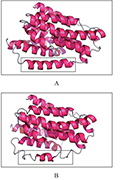
然而atp6 基因存在特殊情况,其在JLCMS9A的cDNA 序列中存在15 个RNA 编辑位点,而在JLCMS9B 中只有12 个。二者的RNA 编辑位点均为C-U 的转换,JLCMS9B 的12 个RNA 编辑位点均在JLCMS9A 中存在,但JLCMS9A 却独有另外3 个RNA 编辑位点。这3个特异位点分别发生在JLCMS9A的atp6 cDNA 序列的第213 bp、614 bp 和623 bp 处,其中第213 bp 处的C-U 转换为亮氨酸(L)同义转换,而第614 bp 和623 bp 处分别发生丝氨酸(S)- 苯丙氨酸(F)和丝氨酸(S)- 亮氨酸(L)转换(图4),这样就引起氨基酸类型的变化,使得JLCMS9A和JLCMS9B 二者编码蛋白完全不同,导致二者的atp6 基因功能有可能发生改变。进一步利用RaptorX在线服务器进行蛋白质结构预测发现,JLCMS9A的atp6 蛋白在图5 标注区域有5 个α螺旋,而JLCMS9B 却只有2 个,可见这2 个氨基酸的差异引起atp6 蛋白结构的变化。由于JLCMS9B 是可育的保持系,而JLCMS9A 是细胞质雄性不育系,因此可推断JLCMS9A 中atp6 基因编码蛋白的改变,导致了蛋白结构的变化,这可能会引起atp6 基因不能正常行使功能。
.png) 图 4 不育系和保持系 atp6 编码氨基酸序列比较 注: PA: JLCMS9A; PB: JLCMS9B; 黑色方框是一致氨基酸, 虚线方框是差异氨基酸 Figure 4 Aligment of amino acids sequences of atp6 between CMS and maintainer line Note: CA and CB are JLCMS9A and JLCMS9B; Black box is the same amino acid, dotted box are the difference amino acid |
|
图 5 不育系和保持系atp6蛋白结构预测图 注: A: JLCMS9A; B: JLCMS9B; 黑色方框标注为二者差异结构 Figure 5 The protein structure prediction of atp6 between CMS and maintainer line Note: A: JLCMS9A; B: JLCMS9B; Black box marked differ- ences structure between A and B |
2讨论
线粒体中与能量代谢相关的功能基因的RNA编辑,是其产生功能蛋白的重要步骤。但一些非正常的编辑将导致了基因结构或表达调控模式发生改变,进而影响相应功能,造成线粒体能量代谢紊乱。通过本研究发现在大豆RN 型CMS 不育系和保持系中,atp1、atp9、coxⅡ、coxⅢ和cob 这些控制线粒体能量代谢的基因的编码蛋白之间没有任何差别,可见这5 个基因的RNA 编辑均不是引起RN 型细胞质雄性不育的相关因子。但由于RNA 编辑仅仅是产生CMS 的一个因素,已知报导的大多数CMS 都是线粒体相关基因起始密码子前端或终止子后端出现插入、缺失或突变而产生新的ORF,引起相关基因功能紊乱造成的,因此我们也不能排除大豆RN 型CMS 存在这种可能。本研究中,atp6 基因在JLCMS9A 和JLCMS9B中发生RNA 编辑的翻译的氨基酸序列并不相同,并且其与其他几个基因相比无论是JLCMS9A 和JLCMS9B,发生RNA 编辑的比率是最高的,其中JLCMS9A达到了2.23%,而JLCMS9B 也有1.79%(表2)。JLCMS9A 独有的3 个编辑位点,其中2 个引起氨基酸的改变,分别是由丝氨酸- 苯丙氨酸和丝氨酸- 亮氨酸的转变。两个经过RNA 编辑的位点均由丝氨酸转换,而丝氨酸为亲水性氨基酸,而转换成的苯丙氨酸和亮氨酸均为疏水性氨基酸,这在一定程度上增加了atp6 编码蛋白质的疏水性。通过蛋白质结构预测还发现二者在结构上也存在明显差异,这种结构差异有可能会造不育系atp6 蛋白不能正常行使功能。
.png) 表 2 本研究所用基因及其引物序列 Table 2 Genes and primer sequences in this study |
韩利涛等(2010)证实在N8855 型CMS 不育系与保持系的atp6 基因保守区有2 个能引起氨基酸编码变化的RNA 编辑位点,并认为可能与CMS 有关。而本研究发现atp6 基因在RN 型CMS 不育系中同样存在特异性RNA 编辑,与保持系相比,不育系多了3 个RNA 编辑位点,这其中2 个位点导致其与保持系编码氨基酸存在差异,这与N8855 细胞质得到的差异氨基酸数目相同,但二者发生变异的氨基酸位点位置却完全不同,而且N8855 和RN 细胞质atp6基因RNA 编辑比率存在显著差异,RN 细胞质显著高于N8855 细胞质。
以上分析表明,不同类型的细胞质的线粒体内某些未知因素可能参与了atp6 基因的RNA 编辑,而atp6 是线粒体呼吸代谢过程中重要的复合酶亚基基因,其参与线粒体对花器官的能量供应,若atp6 基因不能正常转录,将导致使线粒体供能不足,最终导致花粉败育。因此,可以推测不育系atp6 基因发生的特异性RNA 编辑很可能是引起大豆CMS 的原因,但仍需进一步通过将不育系发生RNA 编辑的atp6 基因转入拟南芥和百脉根等模式植物或直接转入大豆,通过在开花期进行花粉育性分析,以证实推测的正确性。
3材料与方法
3.1材料
3.1.1植物材料
供试大豆RN 型细胞质雄性不育系JLCMS9A及其对应保持系JLCMS9B 为本实验室保存。将不育系和保持系种子分别用发芽纸卷好,置于盛有1/3体积水的500 mL 烧杯中,30℃培养箱暗培养7 d后,各取黄化苗10 g,利用差速及密度梯度离心法(费晓艳, 2011)提取大豆线粒体基因组DNA。在吉林省农业科学院范家屯杂交大豆实验基地取开花期的不育系和保持系花苞为材料,利用RNA 提取试剂盒提取总RNA。
3.1.2试剂
Prime ScriptTM Ⅱ cDNA 第一链合成试剂盒和PrimeSTAR HS DNA 酶购自大连宝生物公司;植物总RNA 提取试剂盒购自北京天根生化科技公司;QuickGel Extraction 琼脂糖凝胶回收试剂盒和Pure-Plasmid mini 质粒提取试剂盒购自北京康为世纪生物公司;pEASY-Blunt 亚克隆载体和Trans-T1 感受态细胞购自北京全试金生物公司;其余为国产分析纯试剂。
3.2方法
3.2.1 PCR与RT-PCR
用cDNA 第一链合成试剂盒将不育系和保持系总RNA 合成cDNA。根据Genbank 公布的大豆atp1、atp6、atp9、coxⅡ、coxⅢ和cob 基因ORF 区设计引物,引物序列见表2,均由北京华大基因合成。以此引物对提取的线粒体基因组DNA 和合成的cDNA 分别用PrimeSTAR HS DNA 酶进行PCR 扩增,并重复3 次,分别取每次扩增产物进行下一步操作。
3.2.2扩增片段的回收、克隆和鉴定
扩增得到的各基因PCR 产物经1.2%琼脂糖凝胶电泳检测正确后切胶,用琼脂糖凝胶试剂盒进行胶回收,回收产物与pEASY-Blunt 载体连接,以Trans-T1 大肠杆菌感受态细胞进行转化,筛选阳性克隆,将阳性重组子转入卡那霉素抗性LB 培养基37℃过夜培养,用质粒提取试剂盒提取质粒,用目的基因引物和通用引物M13+/- 进行双重PCR 检测。3.2.3测序与分析
每个基因经3 次不同PCR 扩增并鉴定正确的阳性克隆由天津华大基因测序。测序结果用VectorNTI Suite 8.0 软件进行序列比对和分析;利用RaptorX在线服务器(http://raptorx.uchicago.edu/StructurePrediction/predict/)进行蛋白质结构预测。
作者贡献
刘海军是本研究的实验设计和研究的执行人,并完成论文初稿的写作;赵丽梅和董英山参与实验设计及论文修改;石瑛参与实验设计及数据分析;张春宝是项目的构思者及负责人,参与实验设计、数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学基金(31201224)、吉林省科技发展计划项目(20130206004NY; 201201091)和国家高技术研究发展计划863 计划重点项目(2011AA10A105)共同资助。感谢吉林省农业科学院提供实验材料和仪器设备支持。
Brennicke A., Felder A.M., and Binder S., 1999, RNA editing, FEMS Microbiology Reviews, 23(3): 297-316
Fei X.Y., 2011, Mitochondrial genome sequencing and analysis of cytoplasmic male sterile line and its maintainer line in soybean, Thesis for M.S., Jilin University, Supervisor: Dong Y.S., pp.32-34 (费晓艳, 2011, 大豆细胞质雄性不育系及其配套保持系线粒体基因组的测序与分析, 硕士学位论文, 吉林大学, 导师: 董英山, pp.32-34)
Gray M.W., Hanic-Joyce P.J., and Covello P.S., 1992, Transcription, processing and editing in plant mitochondria, Annu. Rev. Plant Physiol. Plant Mol. Biol., 43(1): 145-175
Han L.T., Yang S.P., Yu D.Y., and Gai J.Y., 2010, Comparative studies of RNA editing of atp6 gene between cytoplasmic male sterile line and its maintainer line in soybean (Glycine max L. Merr.), Dadou Kexue (Soybean Science), 29 (3):361-365 (韩利涛, 杨守萍, 喻德跃, 盖钧镒, 2010, 细胞质雄性不育系与保持系atp6 基因的RNA 编辑比较研究, 大豆科学, 29(3): 361-365)
Howad W., and Kempken F., 1997, Cell type-specific loss of atp6 RNA editing in cytoplasmic male sterile Sorghum bicolor, Proc. Natl. Acad. Sci. USA, 94(20): 11090-11095
Jiang W., Yang S.P., Yu D.Y., and Gai J.Y., 2013, A comparative study of ATPase subunit 9 (Atp9) gene between cytoplasmicmale sterile line and its maintainer line in soybeans, Afr. J. Biotechnol., 10(51): 10387-10392
Kong J., Tan Y.P., Chen Z.Y ., Li S.Q., and Zhu Y.G., 2006, Study on the editing sites in transcripts of functional genesof HL cytoplasmic male sterility rice mitochondria duringmicrogametogenesis, Wuhan Zhiwuxue Yanjiu (Journal ofWuhan Botanical Research), 24(2): 95-99 (孔进, 谭艳平, 陈祖玉, 李绍清, 朱英国, 2006, 水稻红莲型不育系雄性配子发育过程中线粒体功能基因转录本的编辑位点研究, 武汉植物学研究, 24(2): 95-99)
Malek O., Lattig K., Hiesel R., Brennicke A., and Knoop V.,1996, RNA editing in bryophytes and a molecular phylogenyof land plants, EMBO J., 15(6): 1403-1411Rurek M., Szklarczyk M., Adamczyk N., Michalik B., and AugustyniakH., 2001, Differences in editing of mitochondrialnad3 transcripts from CMS and fertile carrots, Acta Biochim.Pol., 48(3): 711-717
Stahl R., Sun S., L'Homme Y., Ketela T., and Brown G.G., 1994, RNA editing of transcripts of a chimeric mitochondrial gene associated with cytoplasmic male-sterility in Brassica, Nucleic Acids Res., 22(11): 2109-2113
Sun H., Zhao L.M., and Huang M., 1994, Studies on cytoplasmic-nuclear male sterile soybean, Chinese Science Bulletin, 39(2): 175-176
Wang J., Cao M.J., Pan G.T., Lu Y.L., and Rong T.Z., 2009, RNA editing of mitochondrial functional genes atp6 and cox2 in maize (Zea mays L.), Mitochondrion, 9(5): 364-369
Wei L., Yan Z.X., and Ding Y., 2008, Mitochondrial RNA editing of F0-ATPase subunit 9 gene (atp9) transcripts of Yunnan purple rice cytoplasmic male sterile line and its maintainer line, Acta Physiologiae Plantarum, 30(5): 657-662
Yang J.H., Zhang M.F., and Yu J.Q., 2007, Alterations of RNA editing for the mitochondrial ATP9 gene in a new orf220-type cytoplasmic male-sterile line of stem mustard (Brassica juncea var. tumida), J. Integr. Plant Biol., 49(5): 672-677
Zhao L.M., Sun H., Wang S.M., Wang Y.Q., Huan M., and Li J.P., 2004, Breeding of hybrid soybean HybSoy1, Zhongguo Youliao Zuowu Xuebao (Chinese Journal of Oil Crop Sciences),26(3): 15-17 (赵丽梅, 孙寰, 王曙明, 王跃强, 黄梅, 李建平,2004, 大豆杂交种杂交豆1 号选育报告, 中国油料作物学报, 26(3): 15-17)