作者
作者  通讯作者
通讯作者
豆科基因组学与遗传学, 2012 年, 第 3 卷, 第 3 篇
收稿日期: 2012年07月11日 接受日期: 2012年07月11日 发表日期: 2012年07月11日
引用格式(中文):
魏海超等, 2012, 大豆中GmRAD与GmDIV基因的in silico鉴定及表达分析, 豆科基因组学与遗传学(online), 3(3):14-22 (doi: 10.5376/lgg.cn.2012.03.0003)
引用格式(英文):
Wei et al., 2012, In silico Identification and Expression Analysis of the GmRAD and GmDIV Genes in Soybean, DoukeJiyinzuxue Yu Yichuanxue (online) (Legume Genomics and Genetics), 3(3): 14-22 (doi: 10.5376/lgg.cn.2012.03.0003)
RAD与DIV属于MYB转录因子家族成员,在金鱼草研究中发现它们对花对称性的形成起着重要作用。本研究利用全基因组信息鉴定了大豆中的GmRAD与GmDIV基因,分析了其蛋白的理化性质、二级结构,基因结构与其在染色体上的分布,并对其系统发生和表达方式进行了研究。研究表明:大豆中存在5个GmRAD基因和5个GmDIV基因,它们分别属于MYB家族的R1和R2R3类,分布在大豆的9条染色体上;进化分析显示GmRAD和GmDIV基因具有不同的进化历程,GmDIV比GmRAD基因更趋于单元发生;表达分析证明,GmDIV和GmRAD成员具有不同的表达模式,其中GmRAD3和GmRAD5以及GmDIV1、GmDIV3、GmDIV4和 GmDIV5在花组织中有较高的表达。
花是被子植物进行有性繁殖的主要器官,千姿百态的花型是植物吸引动物传媒和被特异授粉者识别的重要形态学特征。被子植物始祖的花为辐射对称性花,伴随着特化传粉者的出现才有两侧对称性花的出现。两侧对称花型被认为是由原始辐射对称花型经过多次反复独立进化而来(Endress, 1999; Stebbins, 1974)。研究植物花型发育的分子调控机理,对于认识高等植物发育进化的历程和发展人工改造植物花器 官形状的新方法具有重要的意义。
在过去的十多年中,对模式植物金鱼草的研究发现了由CYCLOIDEA(CYC)、DICHOTOMA(DICH)、RADIALIS(RAD)和DIVARICATA(DIV)共同控制的两侧对称花型发育的分子调控网络。野生型金鱼草的花由2枚背部花瓣,2枚侧部花瓣和1枚腹部花瓣组成;其中背部花瓣和侧部花瓣的形状是不对称的,而腹部花瓣为对映体对称。两个植物特有的TCP家族转录因子CYC和DICH共同控制了金鱼草的背部花瓣属性,当这两个基因发生突变时,所有的花瓣均呈现腹部花瓣的特征,花型由两侧对称变成辐射对称(Luo et al., 1996; 1999)。植物MYB转录因子RAD同样参与了背部花瓣属性的控制,rad突变体与cyc:dich突变体具有类似的表型,在花原基发育的早期,RAD在背部区域表达,但是略晚于CYC,CYC和DICH基因可以激活RAD的表达(Corley et al., 2005)。另一个MYB转录因子DIV蛋白决定了腹部花瓣的特征,DIV基因早期在所有花瓣原基中表达,晚期集中在腹部花瓣原基中表达;在cyc:dich:div三突变体中,所有花瓣均表现为侧部花瓣的特征(Almeida et al., 1997; Corley et al., 2005; Galego and Almeida, 2002)。DIV是一个由307个氨基酸构成的MYB转录因子,含有R2R3两个MYB保守域;RAD是一个只含有一个MYB保守域的93个氨基酸的MYB转录因子。RAD和DIV蛋白具有高度相似的保守结构,可能通过目标序列的竞争结合,或者两者直接结合使DIV所决定的背部属性在背部及侧部受到抑制(Corley et al., 2005)。在野生型金鱼草花发育过程中,两个TCP转录因子CYC和DICH在花原基的背部表达,激活RAD基因的表达;而RAD抑制DIV在背部和侧部花原基中作用,从而使花瓣的背部属性在近轴的背部花瓣和侧部花瓣得到不同程度的体现,形成具有三种不同形态的两侧对称花(Corley et al., 2005; Galego and Almeida, 2002; Cui et al., 2010)。金鱼草中两侧对称花型发育的分子调控网络为认知植物花器官形态发育提供了基础,近年的研究证实在与金鱼草同属唇形目近缘的苦苣苔科和车前科中,由TCP转录因子和MYB转录因子共同调控花型发育的基因调控网络与种类次生辐射对称花的形成相关,并且证实此基因调控网络的蜕变导致虫媒传粉向风媒传粉症候的演化(Zhou et al., 2008; Preston et al., 2011)。
大豆具有典型的两侧对称花型,但是其花瓣的对称性排列方式与金鱼草截然相反。大豆的花瓣是由1枚对称的背部旗瓣、2枚不对称的侧部翼瓣和2枚不对称的腹部龙骨瓣所构成。两侧对称花型在豆科和玄参科中花瓣排列方式也体现了它们独立起源的事实(Donoghue et al., 1998; Stebbins, 1974)。尽管TCP转录因子在控制豆科百脉根两侧对称花的形成过程中起着决定作用(Feng et al., 2006; Wang et al., 2008),但是对于其下游的MYB转录因子是否也被特异的激活导致其两侧对称花的发育仍然不清楚。本研究利用生物信息学的手段鉴定了大豆中RAD与DIV的同源基因,对其蛋白结构、基因结构和理化性质的分析,利用RNA-seq的数据对它们在9个不同时期不同器官的表达情况进行了研究,为进一步研究大豆中GmRAD与GmDIV基因的功能提供了基础。
1结果与分析
1.1大豆 GmRAD与 GmDIV同源基因的鉴定
利用金鱼草中RAD与DIV的氨基酸序列,在大豆蛋白数据库phytozome v7.0中进行Blastp筛选,将所有可能的候选基因构建系统发育树,最终确定了大豆中与金鱼草中RAD同一分支的基因有5个:GmRAD1(Glyma02g18210.1)、GmRAD2(Glyma04g16390.1)、GmRAD3(Glyma03g28050.1)、GmRAD4(Glyma06g46590.1)和GmRAD5(Glyma19g30810.1);大豆中与金鱼草中DIV同一分支的基因有6个:GmDIV1(Glyma02g39000.1)、GmDIV2(Glyma20g16630.1)、GmDIV3(Glyma14g37050.1)、GmDIV4(Glyma11g22960.1)、GmDIV5(Glyma18g07250.1)和Glyma15g36670.1。研究中发现,Glyma15g36670.1与GmDIV2(Glyma20g16630.1)一致区域的相似度为 67%,但是此基因仅含有一个MYB保守区,只有108个氨基酸,推测其可能是GmDIV基因功能演化的一个产物,在下面研究中未被包括。
利用Clustal X(Version 2.1)分别对GmRAD 和 GmDIV蛋白序列进行比较分析结果如图1所示。这些基因所编码的蛋白分别与金鱼草中的RAD、DIV有较高的相似度,与RAD的氨基酸序列相比,GmRAD1的相似度为 66%,GmRAD2为 69%,GmRAD3为72%,GmRAD4为 67%,GmRAD5为58%;与DIV的氨基酸序列相比较,GmDIV1的相似度为59%,GmDIV2为48%,GmDIV3为 60%,GmDIV4为58%,GmDIV5为 60%。GmRAD的氨基酸序列比GmDIV的氨基酸序列分别与RAD和 DIV的氨基酸序列总体上表现出更高的相似性。
|
图1 金鱼草与大豆中RAD 和DIV 氨基酸序列比对 Figure 1 Amino acid alignments of RAD and DIV protein sequences in Antirrhinum majus and Glycine max
|
利用SMART(http://s mart.embl-heidelberg.de/)对这些基因的保守区进行了分析,分析的结果显示在GmRAD中,GmRAD1~GmRAD4均一个具有典型的MYB结构域,位置为:9~59位氨基酸,而GmRAD5在此区间有14个氨基酸的缺失(图1),已经无法检测到典型的MYB结构域,推测此缺失可能导致其功能发生较大的变化。GmDIV1~GmDIV5都具有典型的R2R3MYB保守域的结构,其第一个保守域的位置在29~81位氨基酸之间,第二个保守域的位置在136~188位氨基酸之间,其中GmDIV2两 个保守区之间的距离最大为72个氨基酸。
1.2大豆 GmRAD和 GmDIV的理化性质和二级结构分析
为了进一步确定大豆中GmRAD与GmDIV蛋白质的理化性质,我们分析了它们编码的氨基酸组成及理化性质(表1)。GmRAD1~GmRAD4氨基酸的数目在92~97之间,分子量为10 515.8~10 965.2 Da。
|
表1 大豆与金鱼草中RAD 与DIV 理化分析
Table 1 The biochemical analysis of RAD and DIV in Antirrhinum majus and Glycine max
|
GmRAD5由于中间14个氨基酸缺失,理化性质发生较大差异,其氨基酸数目为82,分子量为9 581 Da。GmDIV1~GmDIV5氨基酸数目在307~327之间,分子量在34 429.1~37 015.9 Da之间。与金鱼草中RAD与DIV相比,GmRAD与 GmDIV蛋白质都在碱性范围内,表明蛋白分子中富含较多的碱性氨基酸,脂肪族氨基酸指数在55~80之间,疏水指数在-1.05~-0.5之间,蛋白的亲水性较高。这些蛋白的不稳定指数在42~85之间,均大于40,为不稳定蛋白。
对GmRAD与GmDIV二级结构的比较发现(表2),大豆中GmRAD与GmDIV蛋白与金鱼草结构高度相似,均由α-螺旋、无规则卷曲和扩展链结构三种形式组成,且构成α-螺旋的氨基酸数目都比较相近(GmRAD, 39~48; GmDIV, 62~89)。金鱼草中RAD含有4个α-螺旋区,大豆的RAD同源蛋白具有相似的结构(GmRAD5序列不完整, 只含有3个α-螺旋);金鱼草DIV含有7个α-螺旋,大豆中的DIV同源基因含有5~7个α-螺旋。在MYB基因家族中,α-螺旋在MYB转录因子发挥识别功能中起着主要作用,因此含有的α-螺旋的不同可能会预示着这些同源基因的功能具有多样性。
|
表2 大豆与金鱼草中RAD 与DIV 二级结构分析
Table 2 The secondary structure analysis of RAD and DIV in Antirrhinum majus and Glycine max
|
1.3大豆 GmRAD和GmDIV的基因结构与染色体上 的分布
利用 GSDS在线工具制作内含子外显子结构图(图 2),与金鱼草中RAD和DIV的基因结构相比,大豆中GmRAD与 GmDIV基因的内含子长度具有较大的差异。在大豆中GmRAD基因的内含子长度明显小于金鱼草中的RAD基因内含子长度(829 bp)。其中GmRAD1~GmRAD4的内含子长度分别为351 bp、515 bp、639 bp和383 bp;GmRAD5含有两个内含子,长度依次是38 bp和94 bp。金鱼草DIV基因的内含子长度为260 bp,与GmDIV的内含子长度也存在显著的差别。其中GmDIV1的内含子长度为336 bp,GmDIV3~GmDIV5的内含子的长度分别为372 bp、441 bp和420 bp;GmDIV2的结构比较特殊,含有4个内含子,长度依次为:140 bp、176 bp、87 bp和320 bp。GmRAD中内含子长度减少和GmDIV基因内含子长度增加的差异提示,两者可能经历了不同的进化选择过程。
|
图2 金鱼草与大豆中RAD 与DIV 基因结构
Figure 2 The gene structures of RAD and DIV in Antirrhinum majus and Glycine max
|
利用大豆基因组数据库中基因的位置信息,制作了大豆中GmRAD与GmDIV在染色体上的分图(图3)。从图3中可以看出,大豆的5个GmRAD基因分别定位于2、3、4、6和19号五个染色体上,GmDIV基因分别定位于2、11、14、18和20号五个染色体上。其中GmRAD1与GmDIV1定位在2号染色体上,其余基因分别分布在不同染色体的不同位置上,提示这些基因可能在大豆多倍化过程中可能经历了不同形式的选择。
|
图3 大豆中GmRAD 与GmDIV 在染色体上分布
Figure 3 The distribution of GmRAD and GmDIV genes in Glycine max chromosomes
|
1.4大豆GmRAD与GmDIV的系统发育分析
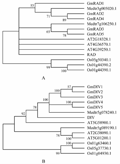
利用金鱼草的RAD与DIV蛋白质序列,在拟南芥,蒺藜苜蓿和水稻中分别找到与金鱼草RAD和DIV同源性较高的基因分别3个、2个和3个。利用这些序列和GmRAD与GmDIV构建系统发育树(图4)。从图4中可以看出,单子叶禾本科植物水稻的RAD和DIV均处于一个独立的进化枝上,说明双子叶中的RAD和DIV基因具有更高的亲缘关系;在双子叶枝中,RAD和DIV的进化方式存在很大的差异,DIV相较与RAD更趋于单元发生。在豆科模式植物蒺藜苜蓿中,RAD和DIV的进化模式呈现出明显的不同:在蒺藜苜蓿中有两个基因与金鱼草的RAD亲缘关系较近,而与DIV亲缘关系较近的只有一个基因。大豆中GmRAD基因同蒺藜苜蓿一样,可以区分为与RAD平行的两个进化枝,其中的GmRAD1、GmRAD2和GmRAD4枝经历了两次分化;它们与GmRAD3、GmRAD5可能来源于不同始祖基因;而大豆中GmDIV与单一的蒺藜苜蓿DIV蛋白聚类在同一进化枝上,并在随后经历了三次分化,说明GmDIV可能来源于相同的始祖基因。系统发育分析比较显示,大豆中GmRAD的进化途径与GmDIV的进化途径明显相同。
|
图4 利用MEGA 5 构建了五个物种的RAD 与DIV 蛋白的NJ 系统发育树
注: 每一分枝显示bootstrap (1 000 次)支持度大于50%的序列; 大豆, GmRAD1~GmRAD5, GmDIV1~GmDIV5; 金鱼草, RAD, DIV; 蒺藜苜蓿, Medtr5g078240.1, Medtr5g089190.1, Medtr3g005020.1, Medtr7g106250.1; 拟南芥, AT4G36570.1, AT4G39250.1, AT2G18328.1, AT5G58900.1, AT5G01200.1, AT2G38090.1; 水稻, Os05g37730.1, Os01g04930.1, Os01g63460.1, Os01g44390.2, Os05g50340.1, Os01g44390.1
Figure 4 Phylogram based on NJ analysis of RAD and DIV proteins from five species using MEGA5
Note: Branches with bootstrap support (1 000 replicates) over 50% are indicated for each branch; The amino acid sequences from the five species are: Glycine max, GmRAD1~GmRAD5, GmDIV1~GmDIV5; Antirrhinum majus, RAD, DIV; Medicago, Medtr5g078240.1, Medtr5g089190.1, Medtr3g005020.1, Medtr7g106250.1; Arabidopsis thaliana, AT4G36570.1, AT4G39250.1, AT2G18328.1, AT5G58900.1, AT5G01200.1, AT2G38090.1; Oryza sativa, Os05g37730.1, Os01g04930.1, Os01g63460.1, Os01g44390.2, Os05g50340.1, Os01g44390.1
|
1.5大豆GmRAD和GmDIV基因的表达分析
利用大豆转录组的数据(Libault et al., 2010),对GmRAD和GmDIV基因在9个不同表达时期及部位(播种84 h后植株的根毛, 播种120 h后植株的根毛, 根瘤, 茎顶端分生组织, 花, 绿色果荚, 叶片, 根和根尖)进行了基因表达分析(图5)。分析结果显示,GmRAD1、GmRAD3~GmRAD5主要在花瓣、叶片和绿色果荚中表达,GmRAD2主要在叶片与根尖中表达;GmDIV1、GmDIV3~GmDIV5主要在绿色果荚、叶片表达,在播种84 h后植株的根毛、根瘤中也有表达,而GmDIV2主要在播种84 h后植株的根毛和茎顶端分生组织中表达。以RPM(Reads Per Million reads)为标准,在花组织中GmRAD1~GmRAD5的表达相对量分别为:0.97、0.00、41.52、0.00和13.52;GmDIV1~GmDIV15的表达相对量分别为:21.25、0.00、64.70、29.9和31.8。在GmRAD基因中,GmRAD3和GmRAD5更有可能参与了花发育的调控,其余基因可能已经被招募到其它器官的发育进程中;在GmDIV基因中,GmDIV1、GmDIV3、GmDIV4和GmDIV5均有可能参与花发育的调控。
|
图5 大豆中GmRAD 与GmDIV 基因表达谱分析
注: 1: 播种84 h 后根毛; 2: 播种120 h 后根毛; 3: 根瘤; 4: 茎顶端分生组织; 5: 花; 6: 绿色果荚; 7: 叶片; 8: 根; 9: 根尖; 方块内颜色显示基因表达水平, 红色最低, 黑色居中, 绿色最高
Figure 5 The expression profiles of GmRAD and GmDIV genes in soybean
Note: 1: Root hair 84HAS; 2: Root hair 120HAS; 3: Nodule; 4: SAM; 5: Flower; 6: Green pod; 7: Leaf; 8: Root; 9: Root tip; Color in square represents the level of gene expression, red is the lowest, black is the middle, green is the highest
|
2讨论
植物中MYB转录因子是植物中最大的一类转录因子家族,广泛参与了植物的发育和代谢调控,所 包含的MYB结构域从一个到四个不等,其结构域数 目和构成与其功能密切相关(Feller et al., 2011)。MYB保守区约含51或52个氨基酸残基,可以形成三段螺旋结构,且在第二段和第三段螺旋之间折转成一定的角度相连接,形成了螺旋-转角-螺旋的结构,构成了一个疏水核心;第一个螺旋目前还没有明确的结构意义,而第二个螺旋同DNA的磷酸骨架相接触,稳固了与DNA的结合,第三个螺旋为识别螺旋,通过氨基酸侧链以及DNA碱基之间的氢键同DNA相识别(Ogata et al., 1996; Dubos et al., 2010)。MYB转录因子基因RAD与DIV作为TCP基因调控的下游基因,它们在控制花对称性的作用机制已经在模式植物金鱼草中得到了阐明(Corley et al., 2005),本研究分析和鉴定了大豆中的GmRAD和GmDIV基因,证实大豆中GmRAD和GmDIV基因至少存在5个拷贝,GmRAD1~GmRAD4基因均具有一个典型的MYB结构域,GmDIV1~GmDIV5均具有典型的R2R3MYB保守域的结构:GmRAD5在MYB保守域存在的14个氨基酸的缺失;同时发现存在着一个与GmDIV2同源性较高但失去一个MYB保守域的Glyma15g36670.1蛋白,大豆中GmRAD和GmDIV基因的多样性为深入研究MYB基因的功能演化提 供了一个很好的体系。
在大豆中GmRAD和GmDIV基因均存在多个拷贝,这与大豆基因组的复制有关,已有的研究结果表明,大豆基因组至少发生了两次复制,引起了整个基因组的高度重复(约75%的基因以多拷贝形式出现) (Schmutz et al., 2010)。对GmRAD系统发育分析比较显示:大豆中GmRAD1、GmRAD2、GmRAD4和GmRAD3、GmRAD5来源于不同的始祖基因,其始祖基因的分化可能早于豆科蝶形花亚科的出现,而大豆GmDIV1~GmDIV5基因则起源于相同的始祖基因,说明这两者之间经历了不同的进化过程。在蝶形花亚科模式植物百脉根的研究中证实,CYC-like基因同样决定和控制着背部花瓣的特征,在独立起源的不同类型两侧对称花中,控制花的两侧对称性的不同机制可能具有共同的分子起源(Feng et al., 2006)。随后在蝶形花亚科的豌豆中(Wang et al., 2008)证明,PsCYC1、PsCYC2和PsCYC3同样参与了豌豆两侧对称花的形成,并与花瓣内部不对称性的形成有关。在豆科中CYC-like基因可能来源与两次基因加倍,第一次基因加倍发生在豆科蝶形花亚科分化之前,导致LEGCYC GROUP Ⅰ和GROUP Ⅱ两类CYC基因的产生;第二次基因加倍发生在在蝶形花亚科分化的早期,LEGCYC GROUP Ⅰ内部又发生基因加倍事件,从而产生了LEGCYC GROUP ⅠA与ⅠB两类基因,因此在豆科中至少存在3个CYC-like基因(Citerne et al., 2003; Fukuda et al., 2003; Ree et al., 2004)。因此,豆科中控制花型TCP基因进化途径与GmRAD和GmDIV基因进化途径不尽一致,模式植物金鱼草中调控两侧对称花发育的分子调控网络的MYB转录因子在大豆中可能发生了功能冗余和亚功能化。大豆中控制两侧花对称性发育基因调控网络的因子在进化中呈现出功能多样性,其花型的调控可能存在更复杂的机制。
3材料与方法
3.1材料
本实验研究所用的金鱼草RAD与DIV蛋白质序列均来自NCBI网站(http://www.ncbi.nlm.nih.gov)其登录号分别为2CJJ_A和AAL78741,大豆、蒺藜苜蓿和水稻全基因组的数据、蛋白数据等均来自于http://www.phytozome.net/soybean网站,拟南芥全基因组的数据来自TAIR网站(http://www.arabidopsis.org/)。
3.2大豆GmRAD与GmDIV基因鉴定及基因结构分析
利用金鱼草中RAD与DIV蛋白序列在大豆基因组数据库(http://www.phytozome.com/),蛋白数据库中进行Blastp查询,为了能得到全部的GmRAD与GmDIV相关基因,E值设为1;利用SMART(http://smart.embl-heidelberg.de/)在线工具对蛋白保守域进行了判定。利用GSDS在线工具(http://gsds.cbi. pku.edu.cn/)制作了GmDIV与GmRAD基因的外显子-内含子结构图。根据phytozome v7.0中标注的基因信息,手工标注大豆GmRAD与GmDIV在染色体中的位置。
3.3大豆GmRAD与GmDIV理化性质分析
利用http://www.expasy.org和http://pbil.ibcp.fr/ htm/index.php网站,对GmRAD与GmDIV同源蛋白的分子量、等电点、亲水性、稳定性和二级结构等方面进行预测,得到GmRAD与GmDIV的基本性质。
3.4多序列比对及系统发育树的构建
将检索到的GmRAD与GmDIV相关序列保存成FASTA格式,利用Clustal X(Version 2.1)对金鱼草、大豆、蒺藜苜蓿、拟南芥、水稻的RAD与DIV相关蛋白进行比对分析,利用MEGA软件(version 5.05) (Tamura et al., 2011)使用Neighbor-Joining的方法对蛋白质序列构建系统发育树,bootstrap 1 000次。
3.5大豆GmRAD与GmDIV基因表达分析
根据已发表的对大豆转录组的测序结果(Libault et al., 2010),在其公布的大豆51 529个基因的表达数据中筛选大豆GmRAD与GmDIV基因的相关数据,检测大豆GmRAD与GmDIV基因在不同组织中的表达情况。将得到的数据使用R软件(Version 2.14.0)环境下的heatmap.2程序进行处理,制作成热图(heatmap)。
作者贡献
冯献忠、杨桂文为本研究的设计者及负责人;魏海超负责样品采集,试验设计与分析,及论文初稿的写作;郝荣华和李方正参与数据分析及论文修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学重大研究计划培育项目(91131008)和山东省杰出青年基金项目(JQ200909)共同资助。
Almeida J., Rocheta M., and Galego L., 1997, Genetic control of flower shape in Antirrhinum majus, Development, 124 (7): 1387-1392
Citerne H.L., Luo D., Pennington R.T., Coen E., and Cronk Q.C., 2003, A phylogenomic investigation of CYCLOIDEA-like TCP genes in the Leguminosae, Plant Physiol., 131(3): 1042-1053
Corley S.B., Carpenter R., Copsey L., and Coen E., 2005, Floral asymmetry involves an interplay between TCP and MYB transcription factors in Antirrhinum, Proc. Natl. Acad. Sci., USA, 102(14): 5068-5073
Cui M.L., Copsey L., Green A.A., Bangham J.A., and Coen E., 2010, Quantitative control of organ shape by combinatorial gene activity, Plos Biol., 8(11): e1000538
Donoghue M.J., Ree R.H., and Baum D.A., 1998, Phylogeny and the evolution of flower symmetry in the Asteridae, Trends in Plant Science, 3(8): 311-317
Dubos C., Stracke R., Grotewold E., Weisshaar B., Martin C., and Lepiniec L., 2010, MYB transcription factors in Arabidopsis, Trends in Plant Science, 15(10): 573-581
Endress P.K., 1999, Symmetry in flowers: diversity and evolution, Int. J. Plant Sci., 160(S6): S3-S23
Feller A., Machemer K., Braun E.L., and Grotewold E., 2011, Evolutionary and comparative analysis of MYB and bHLH plant transcription factors, Plant J., 66(1): 94-116
Feng X., Zhao Z., Tian Z., Xu S., Luo Y., Cai Z., Wang Y., Yang J., Wang Z., Weng L., Chen J., Zheng L., Guo X., Luo J., Sato S., Tabata S., Ma W., Cao X., Hu X., Sun C., and Luo D., 2006, Control of petal shape and floral zygomorphy in Lotus japonicus, Proc. Natl. Acad. Sci., USA, 103 (13): 4970-4975
Fukuda T., Yokoyama J., and Maki M., 2003, Molecular evolution of cycloidea-like genes in Fabaceae, J. Mol. Evol., 57(5): 588-597
Galego L., and Almeida J., 2002, Role of DIVARICATA in the control of dorsoventral asymmetry in Antirrhinum flowers, Genes Dev., 16(7): 880-891
Libault M., Farmer A., Joshi T., Takahashi K., Langley R.J., Franklin L.D., He J., Xu D., May G., and Stacey G., 2010, An integrated transcriptome atlas of the crop model Glycine max, and its use in comparative analyses in plants, Plant J., 63(1): 86-99
Luo D., Carpenter R., Copsey L., Vincent C., Clark J., and Coen E., 1999, Control of organ asymmetry in flowers of Antirrhinum, Cell, 99(4): 367-376
Luo D., Carpenter R., Vincent C., Clark J., and Coen E., 1996, Origin of floral asymmetry in Antirrhinum, Nature, 383 (6603): 794-799
Ogata K., Kanei-Ishii C., Sasaki M., Hatanaka H., Nagadoi A., Enari M., Nakamura H., Nishimura Y., Ishii S., and Sarai A., 1996, The cavity in the hydrophobic core of Myb DNA-binding domain is reserved for DNA recognition and trans-activation, Nat. Struct. Biol., 3(2): 178-187
Preston J.C., Martinez C.C., and Hileman L.C., 2011, Gradual disintegration of the floral symmetry gene network is implicated in the evolution of a wind-pollination syndrome, Proc. Natl. Acad. Sci., USA, 108(6): 2343-2348
Ree R.H., Citerne H.L., Lavin M., and Cronk Q.C., 2004, Hetero- geneous selection on LEGCYC paralogs in relation to flower morphology and the phylogeny of Lupinus (Leguminosae), Mol. Biol. Evol., 21(2): 321-331
Schmutz J., Cannon S.B., Schlueter J., Ma J., Mitros T., Nelson W., Hyten D.L., Song Q., Thelen J.J., Cheng J., Xu D., Hellsten U., May G.D., Yu Y., Sakurai T., Umezawa T., Bhattacharyya M.K., Sandhu D., Valliyodan B., Lindquist E., Peto M., Grant D., Shu S., Goodstein D., Barry K., Futrell-Griggs M., Abernathy B., Du J., Tian Z., Zhu L., Gill N., Joshi T., Libault M., Sethuraman A., Zhang X.C., Shinozaki K., Nguyen H.T., Wing R.A., Cregan P., Specht J., Grimwood J., Rokhsar D., Stacey G., Shoemaker R.C., and Jackson S.A., 2010, Genome sequence of the palaeo- polyploid soybean, Nature, 463(7278): 178-183
Stebbins G.L., 1974, Flowering plants: evolution above the species level, Belknap Press of Harvard University Press, University of Michigan, pp.399
Tamura K., Peterson D., Peterson N., Stecher G., Nei M., and Kumar S., 2011, MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods, Mol. Biol. Evol., 28(10): 2731-2739
Wang Z., Luo Y., Li X., Wang L., Xu S., Yang J., Weng L., Sato S., Tabata S., Ambrose M., Rameau C., Feng X., Hu X., and Luo D., 2008, Genetic control of floral zygomorphy in pea (Pisum sativum L.), Proc. Natl. Acad. Sci., USA, 105 (30): 10414-10419
Zhou X.R., Wang Y.Z., Smith J.F., and Chen R., 2008, Altered expression patterns of TCP and MYB genes relating to the floral developmental transition from initial zygomorphy to actinomorphy in Bournea (Gesneriaceae), New Phytol., 178 (3): 532-543

.png)

.png)